ДНК- и РНК-вакцины: современное состояние, требования к качеству и особенности проведения доклинических исследований | Горяев
1. Tang DC, DeVit M, Johnston SA. Genetic immunization is a simple method for eliciting an immune response. Nature. 1992;356(6365):152–4. https://doi.org/10.1038/356152a0
2. Ulmer JB, Donnelly JJ, Parker SE, Rhodes GH, Felgner PL, Dwarki VJ, et al. Heterologous protection against influenza by injection of DNA encoding a viral protein. Science. 1993;259(5102):1745–9. https://doi.org/10.1126/science.8456302
3. Donnelly JJ, Ulmer JB, Shiver JW, Liu MA. DNA vaccines. Annu Rev Immunol. 1997;15:617–48. https://doi.org/10.1146/annurev.immunol.15.1.617
4. Gurunathan S, Klinman DM, Seder RA. DNA vaccines: immunology, application, and optimization. Annu Rev Immunol. 2000;18:927–74. https://doi.org/10.1146/annurev.immunol.18.1.927
Gurunathan S, Klinman DM, Seder RA. DNA vaccines: immunology, application, and optimization. Annu Rev Immunol. 2000;18:927–74. https://doi.org/10.1146/annurev.immunol.18.1.927
5. Hobernik D, Bros M. DNA vaccines — how far from clinical use? Int J Mol Sci. 2018;19(11):3605. https://doi.org/10.3390/ijms19113605
6. Liu MA, Ulmer JB. Human clinical trials of plasmid DNA vaccines. Adv Genet. 2005;55:25–40. https://doi.org/10.1016/S0065-2660(05)55002-8
7. Weniger BG, Anglin IE, Tong T, Pensiero M, Pullen JK, Nucleic Acid Delivery Devices for HIV Vaccines Workshop Group. Workshop report: nucleic acid delivery devices for HIV vaccines: workshop proceedings, National Institute of Allergy and Infectious Diseases, Bethesda, Maryland, USA, May 21, 2015. Vaccine.
8. Pardi N, Hogan MJ, Porter FW, Weissman D. mRNA vaccines — a new era in vaccinology. Nat Rev Drug Discov. 2018;17(4):261–79. https://doi.org/10.1038/nrd.2017.243
9. Kumaragurubaran K, Kaliaperumal K. DNA vaccine: the miniature miracle. Vet. World. 2013;6(4):228–32. https://doi.org/10.5455/vetworld.2013.228-232
10. Cranenburgh R. Development of the ideal DNA vaccine requires the optimization of delivery strategies and plasmid vectors. BioPharm International. 2011;2011 Suppl.(7). http://www.biopharminternational.com/dna-vaccine-delivery
 Genet Vaccines Ther. 2003;1:2. https://doi.org/10.1186/1479-0556-1-2
Genet Vaccines Ther. 2003;1:2. https://doi.org/10.1186/1479-0556-1-212. Li L, Petrovsky N. Molecular mechanisms for enhanced DNA vaccine immunogenicity. Expert Rev Vaccines. 2016;15(3):313–29. https://doi.org/10.1586/14760584.2016.1124762
13. Liu Z, Chen O, Wall JBJ, Zheng M, Zhou Y, Wang L, et al. Systematic comparison of 2A peptides for cloning multigenes in a polycistronic vector. Sci Rep. 2017;7(1):2193. https://doi.org/10.1038/s41598-017-02460-2
14. Li L, Petrovsky N. Molecular adjuvants for DNA vaccines. Curr Issues Mol Biol. 2017;22:17–40. https://doi.org/10.21775/cimb.022.017
15. Darquet AM, Cameron B, Wils P, Scherman D, Crouzet J. A new DNA vehicle for nonviral gene delivery: supercoiled minicircle. Gene Ther. 1997;4:1341–9. https://doi.org/10.1038/sj.gt.3300540
Gene Ther. 1997;4:1341–9. https://doi.org/10.1038/sj.gt.3300540
16. Hardee CL, Arévalo-Soliz LM, Hornstein BD, Zechiedrich L. Advances in non-viral DNA vectors for gene therapy. Genes (Basel). 2017;8(2):65. https://doi.org/10.3390/genes8020065
17. Stenler S, Blomberg P, Smith CE. Safety and efficacy of DNA vaccines: plasmids vs. minicircles. Hum Vaccin Immunother. 2014;10(5):1306–8. https://doi.org/10.4161/hv.28077
18. Riede O, Seifert K, Oswald D, Endmann A, Hock C, Winkler A, et al. Preclinical safety and tolerability of a repeatedly administered human leishmaniasis DNA vaccine. Gene Therapy. 2015;22(8):628–35. https://doi.org/10.1038/gt.2015.35
19. Pushko P, Ишмухаметов АА, Вredenbeek PP, Lukashevich IS. Экспериментальные живые аттенуированные вакцины против желтой лихорадки на основе инфекционных ДНК. Эпидемиология и вакцинопрофилактика. 2019;18(1):18–25. https://doi.org/10.31631/2073-3046-2019-18-1-18-25
Экспериментальные живые аттенуированные вакцины против желтой лихорадки на основе инфекционных ДНК. Эпидемиология и вакцинопрофилактика. 2019;18(1):18–25. https://doi.org/10.31631/2073-3046-2019-18-1-18-25
20. Pushko P, Lukashevich IS, Weaver SC, Tretyakova I. DNA-launched live-attenuated vaccines for biodefense applications. Expert Rev Vaccines. 2016;15(9):1223–34. https://doi.org/10.1080/14760584.2016.1175943
21. Dallmeier K, Neyts J. Bacterial artificial chromosomes. Patent WIPO N WO2014174078; 2014.22. Ulmer JB, Mason PW, Geall A, Mandl CW. RNA-based vaccines. Vaccine. 2012;30(30):4414–8. https://doi.org/10.1016/j.vaccine.2012.04.060
23. Lundstrom K. RNA-based drugs and vaccines. Expert Rev Vaccines. 2015;14(2):253–63. https://doi.org/10.1586/14760584.2015.959932
Expert Rev Vaccines. 2015;14(2):253–63. https://doi.org/10.1586/14760584.2015.959932
24. Sahin U, Karikó K, Türeci Ö. mRNA-based therapeutics — developing a new class of drugs. Nat Rev Drug Discov. 2014;13(10):759–80. https://doi.org/10.1038/nrd4278
26. Weissman D. mRNA transcript therapy. Expert Rev Vaccines. 2015;14(2):265–81. https://doi.org/10.1586/14760584.2015.973859
27. Youn H, Chung JK. Modified mRNA as an alternative to plasmid DNA (pDNA) for transcript replacement and vaccination therapy. Expert Opin Biol Ther. 2015;15(9):1337–48. https://doi.org/10.1517/14712598.2015.1057563
2015;15(9):1337–48. https://doi.org/10.1517/14712598.2015.1057563
28. Lundstrom K. Latest development on RNA-based drugs and vaccines. Future Sci OA. 2018;4(5):FSO300. https://doi.org/10.4155/fsoa-2017-0151
29. Eberhardt W, Doller A, Akool el-S, Pfeilschifter J. Modulation of mRNA stability as a novel therapeutic approach. Pharmacol Ther. 2007;114(1):56–73. https://doi.org/10.1016/j.pharmthera.2007.01.00230. Atkins GJ, Fleeton MN, Sheahan BJ. Therapeutic and prophylactic applications of alphavirus vectors. Expert Rev Mol Med. 2008;10:e33. https://doi.org/10.1017/S1462399408000859
31. Brito LA, Kommareddy S, Maione D, Uematsu Y, Giovani C, Berlanda Scorza F, et al. Self-amplifying mRNA vaccines. Adv Genet. 2015;89:179–233. https://doi.org/10.1016/bs.adgen.2014.10.005
Adv Genet. 2015;89:179–233. https://doi.org/10.1016/bs.adgen.2014.10.005
32. Klinman DM, Klaschik S, Tross D, Shirota H, Steinhagen F. FDA guidance on prophylactic DNA vaccines: analysis and recommendations. Vaccine. 2010;28(16):2801–5. https://doi.org/10.1016/j.vaccine.2009.11.025
33. Klug B, Reinhardt J, Robertson J. Current status of regulations for DNA vaccines. In: Thalhamer J, Weiss R, Scheiblhofer S, eds. Gene Vaccines. New York: Springer; 2012. P. 285–95. https://doi.org/10.1007/978-3-7091-0439-2_14
34. Bahl K, Senn JJ, Yuzhakov O, Bulychev A, Brito LA, Hassett KJ, et al. Preclinical and clinical demonstration of immunogenicity by mRNA vaccines against h20N8 and H7N9 influenza viruses. Mol Ther. 2017;25(6):1316–27. https://doi.org/10.1016/j.ymthe. 2017.03.035
2017.03.035
МикроРНК человека всесторонне проанализировали
Новости онкологии
31 октября 2019
Матричные РНК передают информацию из генов в белки, в то время как микроРНК играют ключевую роль в регуляции работы генов. Ученые из МФТИ и Медико-генетического научного центра описали сложность взаимодействия микроРНК с матричной и другими РНК человека. Работа опубликована в журнале Frontiers in Genetics.
Иллюстрация. Рибонуклеиновая кислота. Дизайнер: @tsarcyanide, пресс-служба МФТИ
Что такое микроРНК и белки-аргонавты?
Рибонуклеиновая кислота (РНК) – один из основных типов молекул, который реализует генетическую информацию в клетке.
В клетках микроРНК работают в комплексе с белками семейства аргонавтов (AGO). Такой небольшой микроРНК-AGO комплекс соединяется с мРНК в одной из ее частей. С какой мРНК и в какой ее части связаться, определяет микроРНК (для человека их известно ~2,5 тысячи). Белок AGO либо просто блокирует производство белка с мРНК, либо совсем уничтожает мРНК, «разрезая» ее.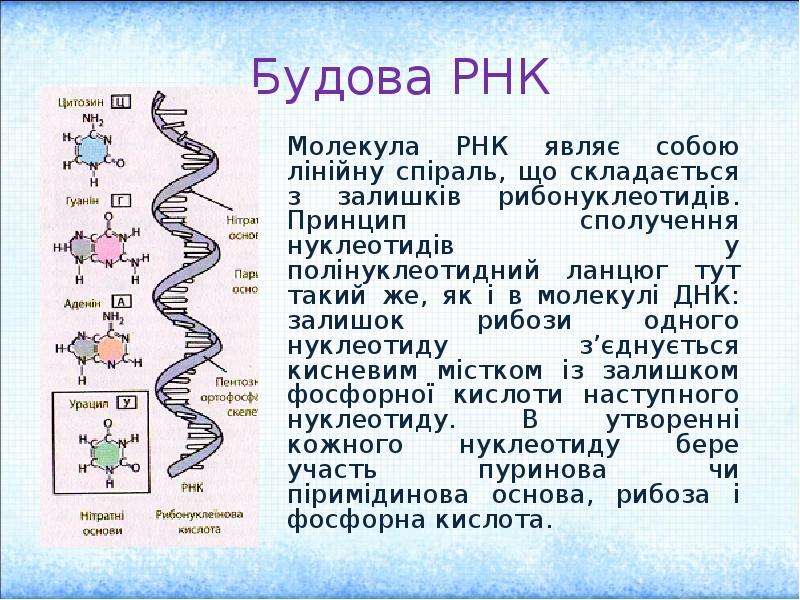 Таким образом, если комплекс микроРНК-AGO вступит во взаимодействие с определенными, парными для него мРНК, то белок с нее синтезироваться больше не сможет. В этом случае получается, что гены, которые кодировали эту мРНК, «замалчиваются», то есть микроРНК, «захватив» мРНК, повлияла на работу генов.
Таким образом, если комплекс микроРНК-AGO вступит во взаимодействие с определенными, парными для него мРНК, то белок с нее синтезироваться больше не сможет. В этом случае получается, что гены, которые кодировали эту мРНК, «замалчиваются», то есть микроРНК, «захватив» мРНК, повлияла на работу генов.
Поэтому, несмотря на то, что микроРНК напрямую взаимодействует с мРНК, под этим взаимодействием также подразумевают взаимодействие между микроРНК и геном, кодирующим эту мРНК. Подобное «замалчивание» – один из многих механизмов регуляции экспрессии генов. Под регуляцией экспрессии генов подразумеваются клеточные механизмы, позволяющие управлять производительностью того или иного гена: полностью или частично выключать или включать его работу. Неправильная регуляция экспрессии генов вследствие «поломки» в функционировании микроРНК может привести к патологиям, в том числе к развитию раковой опухоли.
Представление о взаимодействиях между мРНК и микроРНК в настоящий момент далеко от полного. Для человека сейчас известно ~20 тысяч мРНК и 2,5 тысячи микроРНК. Однако четкого понимания, кто из них с кем соединяется – нет. В своей предыдущей работе исследователи показали, что компьютерные программы, предназначенные для предсказания взаимодействий микроРНК и мРНК, работают не лучшим образом.
Для человека сейчас известно ~20 тысяч мРНК и 2,5 тысячи микроРНК. Однако четкого понимания, кто из них с кем соединяется – нет. В своей предыдущей работе исследователи показали, что компьютерные программы, предназначенные для предсказания взаимодействий микроРНК и мРНК, работают не лучшим образом.
В новой работе ученые решили совместить экспериментальные данные о количестве образующихся в клетке мРНК и микроРНК с данными о взаимодействии между ними для двух типов клеток человека. На примере этих данных они рассмотрели, как связано количество конкретной микроРНК в клетке с тем, как много парных ей мРНК производится в этой же клетке. Можно предположить, что чем больше микроРНК образуется, тем больше соединений она должна образовывать. Оказалось, что это не так. Помимо этого, исследователи разбирались, сколько пар образуется и каким образом, то есть с одинаковыми или разными микроРНК. Говоря по-научному, генетики исследовали, как связаны уровень экспрессии и активность связывания для микроРНК и мРНК. Также они выяснили, как поведение таких пар зависит от типа клеток.
Также они выяснили, как поведение таких пар зависит от типа клеток.
Ольга Плотникова, один из авторов работы, аспирантка МФТИ, рассказывает: «Наше исследование посвящено изучению взаимодействий микроРНК и генов. Известно, что микроРНК – это важные некодирующие малые РНК, которые регулируют экспрессию генов. Ранее мы опубликовали статью, где показали, что программы, которыми пользуются для предсказания взаимодействий микроРНК и генов, работают не очень хорошо. Поэтому нам хотелось получить полную картину взаимодействий микроРНК: кто, с кем и как. Для этого мы проанализировали две единственные на данный момент научные работы с экспериментальными данными по полному интерактому между микроРНК и генами в двух разных клеточных линиях человека. Затем мы соотнесли эти данные с результатами других экспериментальных работ, где определялся уровень экспрессии микроРНК и мРНК в этих же клеточных линиях. Мы показали, что не все гены активно регулируются микроРНК, а потенциал регуляции микроРНК не зависит напрямую от уровня ее экспрессии. Мы также смогли сравнить, как отличаются микроРНК-взаимодействия в двух клеточных линиях».
Мы также смогли сравнить, как отличаются микроРНК-взаимодействия в двух клеточных линиях».
Методы
Основная проблема экспериментального изучения микроРНК взаимодействий – лимитированность методов. Одна группа методов позволяет одним экспериментом проверить одно взаимодействие (так называемые Reporter assay), другая группа – выявить все места связывания с микроРНК, однако она не дает информации о том, какая именно микроРНК связывалась в этом месте (так называемые CLIP-методы). В методе CLIP фиксируется то, что соединилось с белком AGO, и «вытягивается» за этот белок для дальнейшего распознавания провзаимодействовашей мРНК. Таким образом, можно выявить все места связывания микроРНК-мРНК, но при этом не знать, какая из тысяч микроРНК провзаимодействовала.
Недавно были разработаны две близкие методики (методы CLASH и CLEAR-CLIP), которые являются усовершенствованными CLIP-технологиями. Эти методы очень сложны и на данный момент применены только на двух раковых клеточных линиях человека: почек и печени. В данной работе также использовались данные о количестве образующихся мРНК и микроРНК в каждой из упомянутых клеточных линий (данные об экспрессии). Для выявления областей мРНК, где взаимодействие с микроРНК точно происходит, дополнительно ученые использовали экспериментальные данные 79 CLIP-экспериментов, которые не содержат информации о том, какая микроРНК взаимодействует, но позволяют подтвердить, что в данном месте есть взаимодействие с микроРНК.
Эти методы очень сложны и на данный момент применены только на двух раковых клеточных линиях человека: почек и печени. В данной работе также использовались данные о количестве образующихся мРНК и микроРНК в каждой из упомянутых клеточных линий (данные об экспрессии). Для выявления областей мРНК, где взаимодействие с микроРНК точно происходит, дополнительно ученые использовали экспериментальные данные 79 CLIP-экспериментов, которые не содержат информации о том, какая микроРНК взаимодействует, но позволяют подтвердить, что в данном месте есть взаимодействие с микроРНК.
Результаты исследования
In silico ученые доказали, что данные полного взаимодействия микроРНК и генов, полученные усовершенствованными CLIP-методами, в двух разных клеточных линиях человека похожи, и что их можно сравнивать. Было показано, что большая часть комплексов микроРНК – мРНК образуется малым количеством мРНК и микроРНК. Например, только 1–2% кодирующих генов образуют больше десяти различных взаимодействий. Также были выявлены интересные мРНК, которые проявляют «губчатый эффект» – такие мРНК связывались в разных частях мРНК с большим количеством микроРНК (>50). Кроме того, исследователям удалось выявить группу микроРНК, которые, с одной стороны, слабо экспрессируются, а с другой, имеют много взаимодействий. Такой результат не очевиден: кажется, что чем сильнее экспрессируется конкретная микроРНК, тем больше она будет соединяться с различными мРНК.
Также были выявлены интересные мРНК, которые проявляют «губчатый эффект» – такие мРНК связывались в разных частях мРНК с большим количеством микроРНК (>50). Кроме того, исследователям удалось выявить группу микроРНК, которые, с одной стороны, слабо экспрессируются, а с другой, имеют много взаимодействий. Такой результат не очевиден: кажется, что чем сильнее экспрессируется конкретная микроРНК, тем больше она будет соединяться с различными мРНК.
Другая часть научной работы была посвящена созданию коллекции надежных микроРНК-связывающих областей, то есть мест, где мРНК с микроРНК точно провзаимодействуют. Созданная на ее основе онлайн-программа анализирует, находится ли интересующая позиция в геноме определенного человека в месте связывания с микроРНК. Так программа позволяет выявить нарушение связывания с микроРНК и нарушение регуляции генов, а значит, и возможную причину наследственных заболеваний. В перспективе она может быть использована, например, при анализе генома больных.
Картирование полного взаимодействия микроРНК и генов человека может помочь в расшифровке молекулярных основ наследственных и приобретенных заболеваний.
В сентябре этого года МФТИ, в составе консорциум на базе Курчатовского центра, прошел отбор на создание одного из трех Центров геномных исследований мирового уровня, предусмотренных национальным проектом «Наука».
Источник: mipt.ru
О том, как еда изменяет едоков | Научные открытия и технические новинки из Германии | DW
Обмен веществ в живом организме — процесс чрезвычайно сложный, особенно когда речь идет о высокоразвитых организмах. Поэтому столь важную роль играют механизмы, регулирующие этот процесс. Сравнительно недавно ученые обнаружили, что в регуляции обмена веществ участвуют не только ферменты, гормоны, факторы роста и тому подобные соединения, но и так называемые микроРНК. Вообще РНК, то есть рибонуклеиновые кислоты, выполняют в живых клетках множество самых разных функций: так, матричная РНК служит посредником в передаче наследственной информации, закодированной в ДНК, рибосомам, синтезирующим на основе этой информации белки, транспортная РНК доставляет аминокислоты к месту синтеза белков. Есть и другие классы этих так называемых малых некодирующих РНК — они участвуют, например, в регуляции генов или играют роль биологических катализаторов химических реакций в организме.
Есть и другие классы этих так называемых малых некодирующих РНК — они участвуют, например, в регуляции генов или играют роль биологических катализаторов химических реакций в организме.
Седьмой класс питательных веществ
Так вот, о том, что микроРНК, то есть короткие, длиной всего в 2 десятка нуклеотидов, молекулы рибонуклеиновых кислот, способны замедлять или даже блокировать производство того или иного белка, ученые уже знали. Но то, что теперь опубликовала в авторитетном научном журнале Cell Research группа китайских исследователей, вызвало у специалистов немалое изумление. Профессор цитологии Чэнь-Ю Чжан (Chen-Yu Zhang) и его коллеги из Нанкинского университета обследовали 50 добровольцев и обнаружили в их крови и тканях… микроРНК растительного происхождения.
Это и само по себе стало изрядной неожиданностью, поскольку до сих пор считалось, что все растительные ДНК и РНК, попадающие в организм человека с пищей, полностью разлагаются, разрушаются в процессе переваривания. Но еще большее удивление вызвал тот факт, что эти растительные микроРНК участвуют в регуляции метаболизма человека наравне с его собственными микроРНК. По словам руководителя исследования, это открытие заставляет совершенно по-новому взглянуть на роль питания в жизни человека: «Считается, что существует шесть классов питательных веществ — белки, жиры, углеводы, витамины, минеральные вещества и вода. Однако теперь выясняется, что еще и растительные микроРНК, судя по всему, оказывают на активность наших генов, а значит, и на наш обмен веществ, самое непосредственное воздействие. Это дает основание считать их седьмым классом питательных веществ».
Но еще большее удивление вызвал тот факт, что эти растительные микроРНК участвуют в регуляции метаболизма человека наравне с его собственными микроРНК. По словам руководителя исследования, это открытие заставляет совершенно по-новому взглянуть на роль питания в жизни человека: «Считается, что существует шесть классов питательных веществ — белки, жиры, углеводы, витамины, минеральные вещества и вода. Однако теперь выясняется, что еще и растительные микроРНК, судя по всему, оказывают на активность наших генов, а значит, и на наш обмен веществ, самое непосредственное воздействие. Это дает основание считать их седьмым классом питательных веществ».
Рис как фактор, регулирующий расщепление холестерина
В частности, Чэнь-Ю Чжан обнаружил у всех обследованных добровольцев в плазме крови и клетках печени микроРНК типа MIR168a. Весьма обильно эти молекулы присутствуют в рисе. Опыты на трансгенных мышах показали, что в организме человека MIR168a блокирует синтез чрезвычайно важного белка — так называемого клеточного рецептора липопротеинов низкой плотности. Этот белок самым непосредственным образом связан с транспортировкой холестерина и его расщеплением в печени. Таким образом, потребление риса в пищу не только обеспечивает организм человека энергией, но и регулирует активность одного из важных генов, влияя тем самым на обмен веществ и на здоровье человека. Ведь повышенный уровень содержания в крови липопротеинов низкой плотности увеличивает риск атеросклероза.
Этот белок самым непосредственным образом связан с транспортировкой холестерина и его расщеплением в печени. Таким образом, потребление риса в пищу не только обеспечивает организм человека энергией, но и регулирует активность одного из важных генов, влияя тем самым на обмен веществ и на здоровье человека. Ведь повышенный уровень содержания в крови липопротеинов низкой плотности увеличивает риск атеросклероза.
Как растительные микроРНК умудряются уцелеть в пищеварительном тракте человека и проникнуть оттуда в кровь, пока неясно, признает Чэнь-Ю Чжан: «Нам неизвестен этот механизм в деталях. Однако мы полагаем, что эти растительные микроРНК могут захватываться клетками эндотелия сосудов кишечной стенки. При этом мембраны эндотелиальных клеток формируют особые внеклеточные структуры, в которые, как в оболочку, заключаются микроРНК. В таких миниатюрных пузырьках, называемых экзосомами, микроРНК поступают в кровоток».
Новые основы старой китайской медицины
По мнению ученого, его открытие позволяет по-новому объяснить лечебные свойства лекарственных трав, широко применяемых в традиционной китайской медицине. В ходе экспериментов, результаты которых еще только ждут опубликования, Чэнь-Ю Чжан подмешивал экстракт из растения, известный в Китае как эффективное средство против симптомов гриппа, в корм подопытным мышам, которых предварительно инфицировали вирусом инфлюэнцы. Вскоре микроРНК этого растения обнаружились в легочной ткани мышей, где они заблокировали синтез белка, необходимого для размножения вируса, и тем самым предотвратили заболевание. «Это открытие поистине революционно, — не без гордости говорит исследователь. — Возможно, мы начнем вскоре применять различные чужеродные микроРНК — и не только растительные, но и животные, — для лечения болезней. А вводить эти препараты в организм можно будет просто с пищей».
В ходе экспериментов, результаты которых еще только ждут опубликования, Чэнь-Ю Чжан подмешивал экстракт из растения, известный в Китае как эффективное средство против симптомов гриппа, в корм подопытным мышам, которых предварительно инфицировали вирусом инфлюэнцы. Вскоре микроРНК этого растения обнаружились в легочной ткани мышей, где они заблокировали синтез белка, необходимого для размножения вируса, и тем самым предотвратили заболевание. «Это открытие поистине революционно, — не без гордости говорит исследователь. — Возможно, мы начнем вскоре применять различные чужеродные микроРНК — и не только растительные, но и животные, — для лечения болезней. А вводить эти препараты в организм можно будет просто с пищей».
Собственно, идея использовать микроРНК в качестве биологически активного компонента лекарств обсуждается в фармацевтике уже давно. Но до сих пор все эксперименты упирались в одну неразрешимую проблему: как доставить микроРНК точно и целенаправленно в нужное место в организме. Исследования китайских цитологов показали, что природа уже давно предусмотрительно создала такие пути и что функция пищи очевидно не сводится к одному лишь обеспечению организма энергией.
Исследования китайских цитологов показали, что природа уже давно предусмотрительно создала такие пути и что функция пищи очевидно не сводится к одному лишь обеспечению организма энергией.
Автор: Владимир Фрадкин
Редактор: Марина Борисова
Что такое ПЦР-анализ?
Что такое ПЦР-анализ?
ПЦР — это метод лабораторной диагностики, направленный на выявление возбудителей инфекционных заболеваний. Трёхбуквенный вариант — это аббревиатура названия «полимеразная цепная реакция». Собственно, в этом названии и отражается суть метода, но для того, чтобы разобраться, придётся основательно вспомнить школьный курс биологии.
Но сначала – немного истории. Метод ПЦР был разработан в 1983 году Кэри Мюллисом, за что он был удостоен Нобелевской премии. Изначально метод использовался в основном для научных целей, но затем, разглядев его перспективность и эффективность, метод стали продвигать в практическую медицину.
В литературе очень часто встречается такое образное описание ПЦР: это метод, с помощью которого учёные могут находить иглу в стоге сена и затем строить стог из этих игл. В принципе, очень точное описание. Если продолжать сравнение, то игла – это небольшой участок генетического материала микроорганизма, а стог сена – это организм человека, в котором данный микроорганизм поселился.
Что показывает анализ ПЦР
Анализ позволяет обнаружить присутствие генетического материала инфекционных возбудителей. ПЦР в гинекологии и в урологии широко применяется для выявления скрытых и труднодиагностируемых инфекций. В том числе выполняется ПЦР и на ВИЧ.
Принцип работы
За генетическую информацию в живом организме любого размера отвечает ДНК — двухспиральная дезоксирибонуклеиновая кислота, состоящая из последовательности четырех нуклеотидов, которые принято обозначать буквами А (аденин), Г (гуанин), Т (тимидин) и Ц (цитозин). Одно из основных правил генетики – правило комплементарности, то есть нуклеотиды соседних спиралей ДНК соединяются только в определеном порядке: А с Т, Г с Ц, и никак иначе.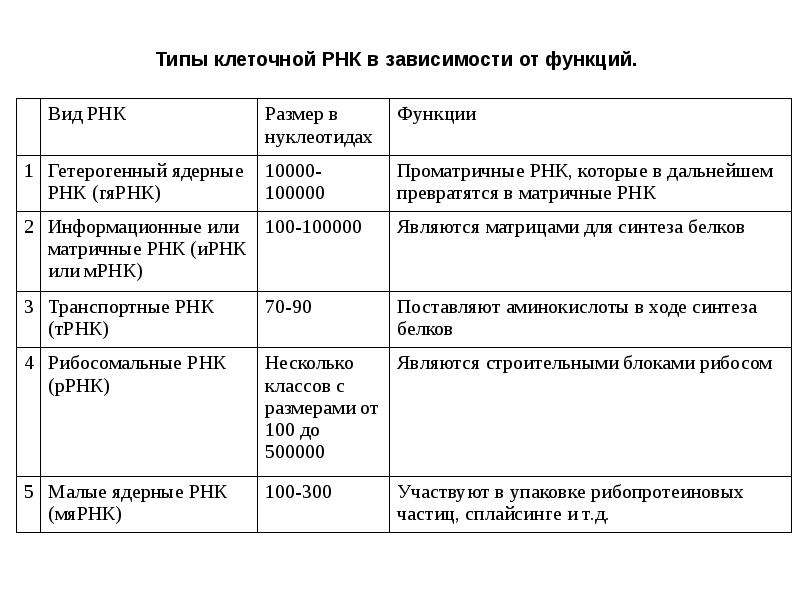
У каждого живого создания (бактерия, вирус, рыба, зверь) – своя ДНК, причём для выявления конкретного организма достаточно иметь лишь небольшой участок этого хранилища генетической информации. Некоторые виды микроорганизмов, например, вирус иммунодефицита человека, хранят генетическую информацию в другой нуклеиновой кислоте – РНК, но и её фрагменты можно находить с помощью ПЦР.
Именно на обнаружении этого небольшого, но уникального для каждого отдельного организма участка и построен принцип ПЦР. Для каждого возбудителя создан свой специфический генетический детектор, эталонный фрагмент ДНК, который по принципу комплементарности точно обнаруживает «свой» фрагмент ДНК и запускает реакцию создания огромного количества его копий.
Один цикл ПЦР длится около трёх минут, количество копий растёт в геометрической прогрессии. Таким образом, за несколько часов количество фрагментов увеличивается в несколько миллиардов раз. Понятно, что теперь определить, какой возбудитель у данной конкретной инфекции, достаточно легко.
Достоинства
Теория – это, безусловно, замечательно, но что мы имеем на выходе? Какую практическую выгоду получает человек, когда отправляется на поиски вредоносных микроорганизмов, вооруженный ПЦР?
- Безусловно, одно из главных достоинств — это универсальностьметода. ПЦР позволяет обнаруживать любые ДНК и РНК, даже когда бессильны другие методы. Оборудование, используемое для ПЦР, стандартно, оно не зависит от того, что именно и где именно мы ищем.
- Следующий плюс — высокая специфичность. В материале, направленном на исследование, определяется уникальная последовательность нуклеотидов, характерная только для конкретного возбудителя. Таким образом, можно говорить, что специфичность метода достигает 100%. Кроме того, это позволяет одновременно, в одном и том же материале, проводить поиск нескольких возбудителей без какого-либо ущерба для качества ответа.
- Метод обладает высочайшей чувствительностью – мы уже говорили о том, что возможно найти всего один фрагмент генетического материала возбудителя.

- Несомненное преимущество метода – оперативность. Постановка реакции занимает несколько часов, таким образом, вся диагностика, от момента сдачи материала на анализ до получения результата, отнимет всего один день.
- При помощи ПЦР определяют возбудителя , а не реакцию на его внедрение со стороны организма. Таким образом, появилась возможность точно диагностировать заболевание еще в инкубационном периоде, когда нет никаких клинических или лабораторных признаков болезни.
расшифровка и применение — АЛТ Украина ЛТД
К нам часто поступает множество вопросов, касающихся такого метода лабораторной диагностики как ПЦР. В своей статье, мы постарались рассмотреть самые актуальные из них.
Надеемся наш материал будет полезным для Вас!
Что такое ПЦР-диагностика?
ПЦР-анализ (полимеразная цепная реакция) — это высокоточный метод диагностики многочисленных инфекций, основанный на анализе генетического материала (ДНК и РНК).
Для диагностики используется слюна, кровь, мокрота, выделения половых органов и прочее биологические материалы, в зависимости от целей исследования.
Какие опасные инфекции можно выявлять в реанимации методом ПЦР?
Методом ПЦР можно выявлять любые опасные инфекции, которые приводят к критическому состоянию пациента. Своевременное выявление инфекционного агента, вызвавшего данное состояние, позволит правильно скорректировать лечение и спасти жизнь.
Подойдёт ли этот метод для скрининга, для подтверждения диагноза?
Скрининговый анализ, в общем понимании, действительно не совсем подходящий метод для неотложной медицины. Скрининг, по большей части, является профилактическим мероприятием и представляет собой массовые тотальные исследования по выявлению скрытых, внешне не проявляющихся или недавно начавшихся патологических процессов у пациентов. Например, ПЦР-диагностика прекрасно используется в гинекологической практике для скрининга вируса папиломы человека (ВПЧ) или болезней, передающихся половым путём (ЗППП).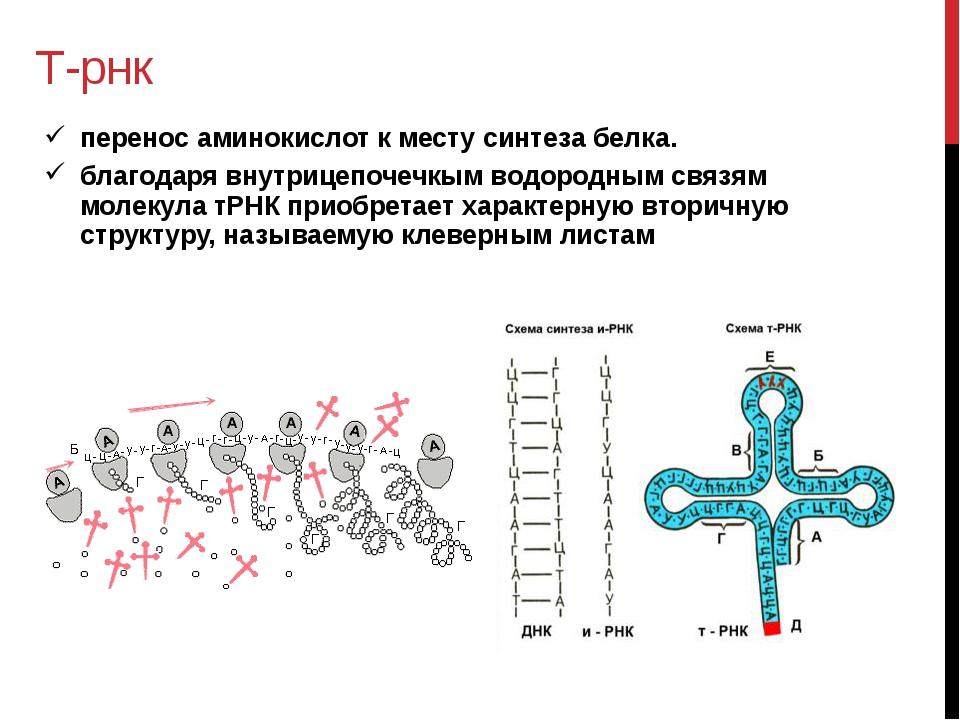 Также ПЦР метод широко используется для скринингового тестирования плазмы крови на предмет выявления вирусов гепатита В, С, D, E. И так далее.
Также ПЦР метод широко используется для скринингового тестирования плазмы крови на предмет выявления вирусов гепатита В, С, D, E. И так далее.
Для скрининга эффективнее использовать ныне устаревающий метод ИФА или современной ИХЛА?
С методами ИФА и ИХЛА не всё так однозначно. Они выявляют косвенные признаки патологических изменений и это является их главным недостатком. Организму на вредное воздействие необходимо вначале сформировать иммунный ответ, наработать антитела, которые в последующем возможно выявить только со временем. Может возникнуть ситуация, когда организм уже заражен какой-либо инфекцией, а антител к ней еще нет или их титр находится ниже порога чувствительности тест-набора. Или наоборот – возбудителя уже нет в организме, а антитела ещё выявляются.
Хронологически метод ПЦР в клинической практике появился позже ИФА и ИХЛА и по факту является более современным. По показателям специфичности и чувствительности ПЦР выше данных иммунологических методов. К тому же ПЦР-анализ выявляет прямую первопричину процессов – их генетическую основу, а не следствие. Для получения нужного результата при постановке ПЦР нет необходимости выявлять весь геном мишени. Задачи по анализу всего генома решаются с помощью других методов (например, таргетный или полногеномный анализ с помощью секвенирования нового поколения – NGS). С помощью ПЦР-теста выявляется высоко консервативный фрагмент ДНК (определённая нуклеотидная последовательность), однозначно характеризующий мишень. А при методе ПЦР в реальном времени можно даже определить изначальное количество мишени в пробе.
Для получения нужного результата при постановке ПЦР нет необходимости выявлять весь геном мишени. Задачи по анализу всего генома решаются с помощью других методов (например, таргетный или полногеномный анализ с помощью секвенирования нового поколения – NGS). С помощью ПЦР-теста выявляется высоко консервативный фрагмент ДНК (определённая нуклеотидная последовательность), однозначно характеризующий мишень. А при методе ПЦР в реальном времени можно даже определить изначальное количество мишени в пробе.
Что этот метод даст для лечения и выявления мультирезистентного штама микобактерий?
Выявление мультирезистентных шаммов позволит изначально не использовать те антибиотики, к которым у возбудителя существует генетически обусловленная стойкость, а использовать другие препараты. Например против штаммов Mycobacterium tuberculosis, устойчивых к Изониазиду и Рифампицину сразу применять фторхинолоны или инъекционные препараты.
Можно ли при ВИЧ вылечить чувствительные штаммы?
Вирус иммунодефицита человека (ВИЧ) вообще не восприимчив ко антибиотикам и противомикробным препаратам, потому что не является бактерией. Против вирусов необходимо применять противовирусные препараты. Трудность лечения вирусных заболеваний вообще и СПИДа в частности обусловлена их биологической особенностью – активная фаза жизнедеятельности вируса проходит только внутри живой клетки. Поэтому вирусные частицы защищены от воздействий внешней среды. Уничтожить вирус можно только с клеткой, в случае с ВИЧ – с клеткой иммунной системы.
Против вирусов необходимо применять противовирусные препараты. Трудность лечения вирусных заболеваний вообще и СПИДа в частности обусловлена их биологической особенностью – активная фаза жизнедеятельности вируса проходит только внутри живой клетки. Поэтому вирусные частицы защищены от воздействий внешней среды. Уничтожить вирус можно только с клеткой, в случае с ВИЧ – с клеткой иммунной системы.
Как исследование соотношения субпопуляций лейкоцитов выявляет онкомаркеры?
Числовое значение соотношения субпопуляций лейкоцитов является индикаторным или маркерным показателем наличия онкологического процесса, т.е. одним из онкомаркеров. В норме и при патологии данное значение отличается. Например, при значениях CD4+/CD8+ в диапазоне от 1,2 до 2,6 может свидетельствовать о возможном наличии в организме множественной миеломы или солидных опухолей. Увеличение значения более до 3 и более может указывать на острый Т-лимфобластный лейкоз или хронический Т-лейкозе. И т.д. Другими словами –в данном случае преобладание Т-хелперов над Т-киллерами специфически информативно.
Но онкомаркеры могут быть и генетическими. На сегодняшний момент обнаружены тысячи мутантных аллелей генов, которые с той или иной вероятностью могут приводить к развитию онкологического процесса. На рынке существует множество тест-систем для выявления онкомаркеров разных производителей.
Рак шейки матки целесообразно предотвращать (или диагностировать на ранней – внутриэпителиальной стадии) до его развития путём разработки и внедрения государственных программ по тотальному скринингу женщин на наличие ВПЧ с регулярными подтверждающими цитологическими исследованиями. Также огромным фактором по своевременному предотвращению данного заболевания будет внедрение вакцинации против ВПЧ всех младенцев при рождении (и мальчиков и девочек).
ПЦР (COVID-19), РНК коронавируса SARS-CoV-2
ПЦР исследование направлено на выявление РНК (генетического материала) возбудителя, вызвавшего пандемию COVID 19 – коронавируса SARS CoV-2.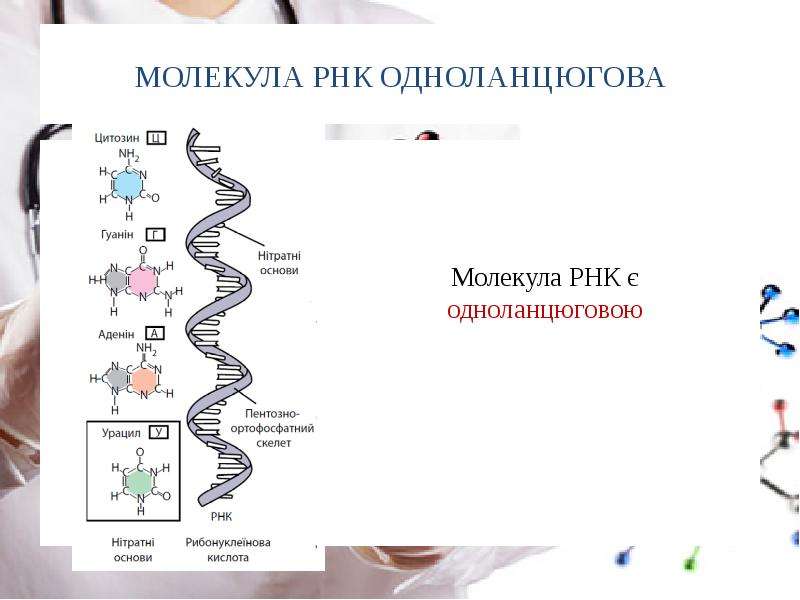 Является специфическим тестом, подтверждающим инфекцию COVID 19.
Является специфическим тестом, подтверждающим инфекцию COVID 19.
SARS-CoV-2 – новый тип коронавируса, высокозаразен. Источником заражения может быть заболевший человек, имеющий клинические признаки и симптомы заболевания; внешне здоровый, находящийся в инкубационном (скрытом) периоде болезни; или внешне здоровый бессимптомный носитель. Распространяется коронавирус воздушно-капельным (при кашле, разговоре), воздушно-пылевым (воздух) и контактным путём (дверные ручки, поручни и т.д). Входные ворота инфекции – слизистая оболочка носоглотки и ротоглотки, по мере прогрессирования инфекции проникает в нижние дыхательные пути и поражает ткань лёгких и мелкие сосуды. К подозрительным признакам относятся сухой кашель без насморка и отделения мокроты, нарушение обоняния и вкуса, мышечные боли, высокая температура, при продолжении инфекции – одышка, чувство нехватки воздуха и сдавления в грудной клетке. Течение заболевания варьирует от бессимптомных форм до тяжелого течения с развитием воспаления легких, дыхательной недостаточности и поражения других органов и систем.
Исследование в медицинских офисах лаборатории проводится только для лиц без признаков ОРВИ, без температуры, не имевших в течение пары недель контактов с больным COVID 19. При себе обязательно иметь паспорт, анонимно исследование не проводится. Сдать мазок можно в часы работы с пробами на ковид в специально выделенных медицинских офисах, либо воспользоваться службой выезда на дом или услугой предоставления набора для самостоятельного взятия биоматериала.
При наличии признаков острого респираторного заболевания или при подозрении на COVID, рекомендуется вызвать участкового терапевта, неотложную или скорую помощь в зависимости от состояния.
В каких случаях назначается исследование?
- При возвращении из поездки (зарубежной или из близлежащих регионов)
- При необходимости предъявить результат теста по месту требования (для перелёта, в санаторно-курортные учреждения, для госпитализации и т.д)
- Перед посещением родственников, находящихся в группе риска по тяжелому течению COVID: возраст старше 65 лет, наличие сахарного диабета, артериальной гипертензии и других отягощающих заболеваний
Что означают результаты теста?
Формат выдачи результата – качественный.
В норме ответ «не обнаружено».
Результат «обнаружено» означает наличие возбудителя (выявление РНК коронавируса) и является подтверждающим исследованием для постановки диагноза COVID. Независимо от того, имеются признаки заболевания или нет, пациент обязан обратиться к врачу и соблюдать карантин 2 недели до получения отрицательного результата следующего ПЦР исследования. Все данные пациентов, имеющих положительные результаты ПЦР- тестов, передаются в органы Роспотребнадзора.
Сроки выполнения теста.
2 дня
Как подготовиться к анализу?
Материал для исследования – мазок из носоглотки и ротоглотки. Материал для исследования можно сдать в течение дня, при этом нужно воздержаться от использования местных препаратов и приёма пищи в течение 2ух часов до исследования. Пить воду можно. Анализ можно сдать только при предоставлении всех паспортных данных, анонимная сдача невозможна.
Бессимптомные пациенты с COVID-19 выделяют столько же вирусной РНК, сколько больные
Исследователи из Южной Кореи опубликовали результаты наблюдений за молодыми людьми, зараженными коронавирусом SARS-CoV-2, часть которых имела симптомы COVID-19, а другие были бессимптомными. Оказалось, что люди, не имеющие симптомов, также могут быть распространителями вируса.
Оказалось, что люди, не имеющие симптомов, также могут быть распространителями вируса.
Участниками исследования стали жители южнокорейского города Тэгу, где в феврале 2020 года была зарегистрирована крупная вспышка коронавирусной инфекции в местном религиозном сообществе. Инфицированных было так много, что представители минздрава Южной Кореи решили изолировать бессимптомных и малосимптомных граждан в общественных медицинских центрах — временных медучреждениях, построенных во время пандемии.
Наблюдения за изолированными гражданами проводились с 6 по 26 марта 2020 года. Всего в исследовании участвовали 303 пациента из Тэгу с лабораторно подтвержденным заражением SARS-CoV-2. Средний возраст испытуемых составлял 25 лет (от 22 до 36), 66,3% были женщинами. Только у 3,9% были хронические заболевания: у десяти человек гипертония, у одного рак и еще у одного — астма. Участники исследования находились в изолированных палатах (92%) или в общих помещениях с отдельными комнатами (8%). Пациентов ежедневно опрашивали по телефону на предмет симптомов и периодически брали у них образцы биоматериала. Температуру измеряли дважды в день.
Пациентов ежедневно опрашивали по телефону на предмет симптомов и периодически брали у них образцы биоматериала. Температуру измеряли дважды в день.
Тест на SARS-CoV-2 методом ОТ-ПЦР РВ проводился с помощью тест-набора Allplex 2020-nCoV (Seegene). Определяли три генетических маркер: гены envelope (env), РНК-зависимой РНК-полимеразы (RdRp) и нуклеокапсидного белка N. Если значение Ct (пороговое число циклов) было меньше 40, результаты теста считались положительными; по Ct также оценивали вирусную нагрузку.
Мазки из носоглотки и ротоглотки, а также образцы мокроты из нижних дыхательных путей. анализировали на 8, 9, 15 и 16-й дни изоляции. Необходимость тестирования на 10, 17, 18 и 19-й дни определял врач. Бессимптомными считались пациенты, у которых не было признаков заражения SARS-CoV-2 на протяжении всего периода наблюдений с момента постановки диагноза. Пресимптомными — пациенты, у которых признаки заражения появились в период наблюдения.
Из 303 человек 110 (36,3%) не имели никаких симптомов на момент изоляции.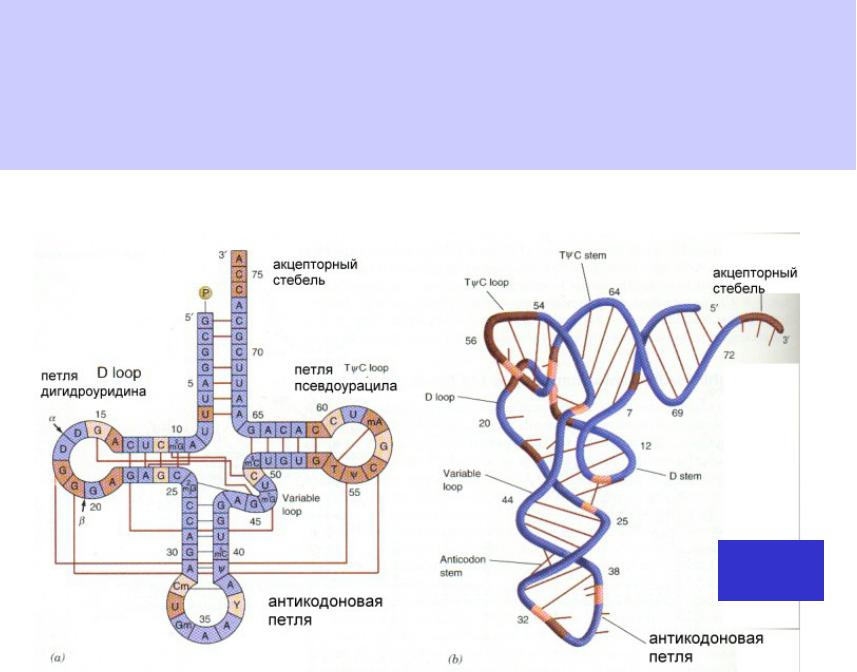 Из них у 21 человека (19,1%) симптомы развились в период наблюдений. В среднем с момента постановки диагноза до появления признаков инфицирования у них проходило 15 дней (от 13 до 20). Остальные 193 человека на момент изоляции имели ряд характерных симптомов COVID-19: кашель, насморк и заложенный нос, выделение мокроты, боли в горле, утрата обоняния; менее чем у 10% отмечались диарея, утрата вкусовых ощущений, боли в животе, рвота.
Из них у 21 человека (19,1%) симптомы развились в период наблюдений. В среднем с момента постановки диагноза до появления признаков инфицирования у них проходило 15 дней (от 13 до 20). Остальные 193 человека на момент изоляции имели ряд характерных симптомов COVID-19: кашель, насморк и заложенный нос, выделение мокроты, боли в горле, утрата обоняния; менее чем у 10% отмечались диарея, утрата вкусовых ощущений, боли в животе, рвота.
К 15-16 марта 2020 изоляция завершилась для 84 человек с двумя последовательными отрицательными результатами тестов: 26 бессимптомных и 58 имевших симптомы.
Доля испытуемых с отрицательным результатом среди бессимптомных пациентов на 14 и 21-й день составляла 33,7% и 75,2%, а для пациентов с симптомами, включая пресимптоматических — 29,6% и 69,9%. В среднем с момента от постановки диагноза до первого отрицательного результата прошло 17 дней у бессимптомных инфицированных людей и 19,5 дней — у пациентов с симптомами.
Пороговое число Ct для гена RdRp из мазков, взятых из верхних дыхательных путей, изменялось медленнее у людей с выраженными симптомами COVID-19, однако Ct для гена env из мокроты, полученной из нижних дыхательных путей, медленнее изменялось у бессимптомных пациентов. Вро всех остальных случаях значимой разницы между показателями Ct симптомных и бессимптомных обнаружено не было.
Вро всех остальных случаях значимой разницы между показателями Ct симптомных и бессимптомных обнаружено не было.
Таким образом, у 80,9% бессимптомных пациентов с положительным результатом теста на РНК SARS-CoV-2 признаки болезни так и не появились на протяжении 24 дней. При этом динамика Ct у них в целом была такой же, как у пациентов с признаками COVID-19. Иначе говоря, вирусная нагрузка в обеих группах изначально была достаточно высокой и снижалась с одинаковой скоростью. Это позволяет предполагать, что риск распространения инфекции пациентами без симптомов COVID-19 довольно велик.
Исследователи напоминают, что обнаружение вирусной РНК не следует приравнивать к присутствию живого вируса, способного заражать других людей. Поэтому для оценки рисков распространения SARS-CoV-2 бессимптомными носителями необходимы дополнительные исследования, например, культивирование вируса из образцов.
Расшифровка COVID-19 | Гарвардская медицинская школа
Эта статья является частью продолжающегося охвата Гарвардской медицинской школы медицины, биомедицинских исследований, медицинского образования и политики, связанной с пандемией SARS-CoV-2 и заболеванием COVID-19.
Почему одни люди заболевают SARS-CoV-2, а другие, инфицированные вирусом, не испытывают никаких симптомов?
Почему у некоторых людей появляются неврологические, сердечно-сосудистые или желудочно-кишечные симптомы, а у других инфекция остается в дыхательной системе?
Поскольку пожилой возраст и сопутствующие заболевания подвергают людей большему риску серьезного заболевания или смерти от COVID-19, почему некоторые молодые и в остальном здоровые люди с трудом переносят болезнь, в то время как некоторые пожилые люди с множественными факторами риска переносят ее лишь с незначительными? симптомы?
Помимо индивидуального поведения, ранее существовавших условий и чистой удачи, ответы могут лежать в генах людей.
Узнайте больше о HMS здесь
«У нас есть возможность увидеть, есть ли варианты генов, которые отличают людей, более восприимчивых к вирусу, от более устойчивых», — сказал Тинг Ву, профессор генетики в Гарвардской медицинской школе.
генетиков, в том числе многие из Института Блаватника в HMS, объединились с учеными из разных дисциплин, чтобы исследовать такие важные вопросы, как эти, когда пандемия COVID-19 охватывает весь земной шар, заражая миллионы и убивая сотни тысяч.
Они работают день и ночь, чтобы понять новый коронавирус в надежде, что их идеи помогут коллегам-исследователям определять или разрабатывать эффективные методы лечения, разрабатывать вакцины, отслеживать инфекции и предотвращать будущие болезни и смерть.
В то время как большинство исследований в кампусе HMS в марте было свернуто для защиты здоровья населения, несколько лабораторий Департамента генетики получили разрешение на продолжение ограниченных личных операций для проектов, которые могут оказать немедленное влияние на пандемию COVID-19.Другие нашли способы внести свой вклад из дома.
«Мы все пытаемся использовать в этом свой опыт», — сказала Дженни Ян, аспирант биологических и биомедицинских наук в HMS, специализирующаяся на генетике и геномике.
В то время как одни генетики HMS тщательно исследуют геном человека, другие анализируют геном вируса, чтобы отследить распространение различных клонов по всему миру и помочь определить, влияют ли какие-либо мутации на активность вируса.
Некоторые из них изучают динамику генов в клеточных культурах и на моделях животных, чтобы раскрыть подсказки о том, как организм реагирует на вирус на молекулярном уровне, будь то открытие доступа SARS-CoV-2 или запуск каскада, который приводит к повреждению тканей и органов или беглое воспаление.
Третьи применяют свои знания о технологиях на основе ДНК и РНК для улучшения существующих тестов и разработки новых, которые могут диагностировать COVID-19 или выявлять доказательства перенесенной инфекции.
Неясно, какой из этих экспериментов даст полезную информацию и насколько быстро. В конце концов, в науке количество неудач превышает успех, а открытие требует времени.
Что поддерживает исследователей, так это возможность делать открытия или разработки инструментов, которые выявляют слабые стороны вируса или сильные стороны человеческого тела, информируют общественную политику, защищают здоровье человека сегодня или помогают предотвратить будущие пандемии.
«Вирус вызвал столько страданий и экономический спад, мы должны стрелять в него всем, что можем», — сказал Харальд Рингбауэр, научный сотрудник HMS в области генетики. «Сейчас важно узнать больше о вирусе и его поведении, и у генетики есть огромный потенциал, чтобы научить нас этим вещам».
Получение полной картины того, какие гены, если таковые имеются, влияют на восприимчивость человека к новому коронавирусу, потребует много времени и анализа геномов многих тысяч людей. Тем временем Ву и его коллеги начинают толчок, используя существующий ресурс, проект Personal Genome Project (PGP), и рассматривая крайние случаи, крайности.
«Мы вылавливаем генетических аномалий — тех, кто находится далеко на концах кривой распространения COVID-19», — сказал Джордж Черч, , профессор генетики Роберта Уинтропа в HMS и один из основателей факультета Wyss.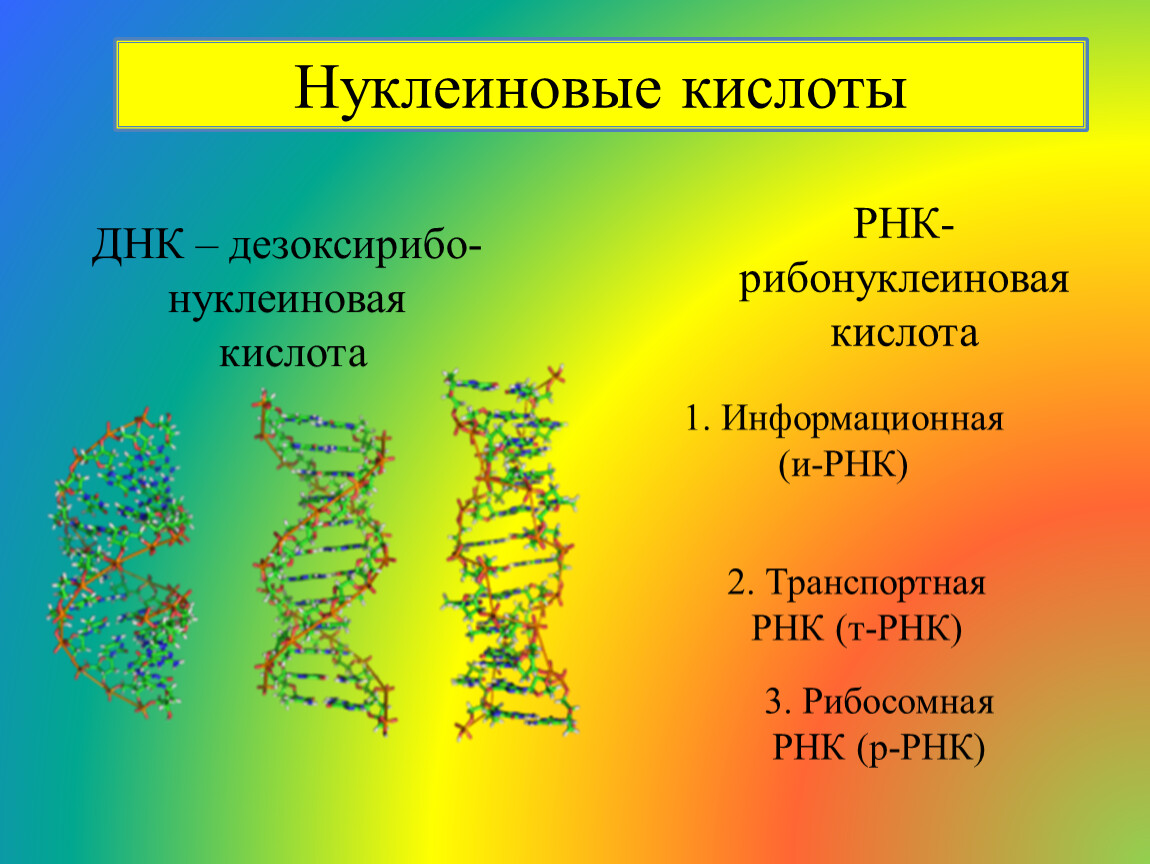 Институт биологической инженерии, лаборатория которого занимается множеством усилий, связанных с пандемией COVID-19.
Институт биологической инженерии, лаборатория которого занимается множеством усилий, связанных с пандемией COVID-19.
«Нам не обязательно проводить глубокое погружение в 7 миллиардов человек, чтобы помочь нам предсказать, кто с большей вероятностью заболеет, а кто протекает бессимптомно, но не следует контактировать с другими людьми, потому что они все еще распространяют вирус», — сказал он.
Запущенная в 2005 году в церковной лаборатории и теперь охватывающая несколько стран, PGP приглашает добровольцев делиться информацией о своем здоровье и генетике для научных исследований. В настоящее время программа включает более 6000 участников, около 500 из которых загрузили свои полные последовательности генома.Церковь — одна из них.
Команда планирует искать подсказки в данных, собранных участниками, таких как почтовый индекс, пол, раса и этническая принадлежность, состояние здоровья, курение и курение, а также подробности о любых симптомах COVID-19, результатах тестов и потенциальных возможностях. подверженности воздействию вируса. Если отдельные участники выделяются, команда может проанализировать любую присланную ими ДНК.
подверженности воздействию вируса. Если отдельные участники выделяются, команда может проанализировать любую присланную ими ДНК.
Ву также предполагает привлечь больше участников в горячие точки COVID-19, чтобы помочь выявить соответствующие варианты генов, используя результаты опроса для определения приоритетности участников для секвенирования генов.
Если команда PGP или другие исследователи действительно найдут варианты, которые повышают или снижают риск серьезного заболевания или смерти от COVID-19, возможно, что люди могут быть проверены на эти варианты. Это, в свою очередь, может упростить определение того, кто может наиболее безопасно работать на передовой в определенных отраслях, а кто должен быть более защищен, сказал Ву, а также кому следует принимать дополнительные меры предосторожности в общинах, например, люди, находящиеся на высоком уровне. риск переноса и распространения вируса без появления самих симптомов.
PGP предлагает только один из многих способов исследовать генетику инфекции SARS-CoV-2. У него есть свои слабые стороны — например, репозиторий меньше, чем у других международных генетических баз данных, — но он уже получил одобрение IRB и может похвастаться активной базой пациентов.
У него есть свои слабые стороны — например, репозиторий меньше, чем у других международных генетических баз данных, — но он уже получил одобрение IRB и может похвастаться активной базой пациентов.
«PGP уникален по своей готовности», — сказал Ву, который также является ассоциированным преподавателем Wyss. «Этот раунд может принести или не принести какие-либо полезные результаты, но он показывает, насколько хорошо существующая программа может быть подготовлена к реагированию на возникающие потребности — и насколько полезным может быть предварительное планирование.«
«При достаточном внимании и финансировании подобные программы могут быть готовы к реагированию на следующую пандемию», — добавила она.
Члены команды проекта — Сара Уэйт Заранек, Александр Уэйт Заранек, Ранджан Ахуджа, Майкл Чоу, Джейсон Бобе, Престон Эстеп и Джантин Луншоф.
Какое отношение имеет миграция групп ранних людей из Африки к распространению SARS-CoV-2 в 2020 году? Сходные методы анализа генома проливают свет на обоих.
По мере того как SARS-CoV-2 заражает людей и размножается, отдельные вирусы развиваются, приобретая генетические мутации. Различные наборы мутаций позволяют различать разные линии вируса, которые могут быть связаны с разными частями мира. Отслеживание этих мутаций позволяет исследователям изучать, как вирус перемещается.
Вирусы, взятые у группы пациентов в Батон-Руж, например, могут иметь уникальный генетический отпечаток, что указывает на локальное распространение SARS-CoV-2, в то время как у пациента в Нью-Йорке может быть почти идентичная версия вируса. одному, циркулирующему в Италии, что свидетельствует о том, что путешествия людей способствовали заражению.
Генетическое секвенирование и вычислительные методы, используемые для отслеживания движения линий SARS-CoV-2 и привязки их к определенным местам, напоминают те, которые использовались для реконструкции того, как группы людей десятки тысяч лет назад были связаны друг с другом и как они перемещались. земной шар основан на ДНК, извлеченной из древних человеческих костей. Это не случайно: многие инструменты, используемые в исследованиях древней ДНК, были впервые разработаны для вирусологии.
земной шар основан на ДНК, извлеченной из древних человеческих костей. Это не случайно: многие инструменты, используемые в исследованиях древней ДНК, были впервые разработаны для вирусологии.
генетиков HMS, привыкших исследовать предысторию, теперь обращаются к COVID-19.
«Мы надеемся повторно использовать в вирусологии опыт, полученный при изучении древней ДНК», — сказал постдок Рингбауэр, работающий в лаборатории специалиста по древней ДНК Дэвида Райха, профессора генетики в HMS. Райх также работает в Гарвардском университете, Институте Броуда Массачусетского технологического института и Гарварда.
«Конечно, над новым коронавирусом уже работают великие ученые, — сказал Рингбауэр. «Мы просто надеемся, что сможем внести небольшой вклад».
Исследователи древней ДНК могут предложить несколько навыков, например, знакомство с анализом больших наборов данных.По словам Рингбауэра, когда ученые изучали штаммы вирусов Зика и Эбола в последнее десятилетие, они работали с несколькими сотнями последовательностей. По состоянию на 10 июня было собрано более 42 000 последовательностей генома SARS-CoV-2.
По состоянию на 10 июня было собрано более 42 000 последовательностей генома SARS-CoV-2.
Вирус реплицируется путем клонирования своей РНК, создавая идентичные копии самого себя (за исключением любых новых мутаций). Большая часть человеческой ДНК, напротив, реплицируется посредством рекомбинации, смешивания генетического материала от двух родителей.
Но исследователи древней ДНК также специализируются на изучении двух исключений из этого правила: Y-хромосомы, которые передаются от мужского пола к мужскому потомству без рекомбинации, и митохондриальная ДНК, которая полностью наследуется от матери.
Отслеживание сходства и различий в Y-хромосоме и митохондриальной ДНК, которые эволюционируют подобно вирусной РНК, позволило сузить сроки миграции из Африки и других миграций и реконструировать материнские и отцовские генеалогические деревья на протяжении тысячелетий, так же как отслеживание вирусная РНК от пациента к пациенту может реконструировать генеалогическое древо SARS-CoV-2 в режиме реального времени.
Когда они закончат сбор и очистку данных, Рингбауэр и Райх проанализируют геномы SARS-CoV-2, чтобы помочь исследователям ответить на многие вопросы о пандемии, в том числе: Насколько подвижен вирус? Сколько пламени раздувается искрами, возникшими на месте, а не где-то далеко? Большинство случаев вызвано несколькими суперраспространителями или большинство людей в среднем мало взаимодействуют с инфицированными?
И изменяют ли какие-либо мутации поведение вируса, делая его более или менее сложным для передачи или более или менее вероятно вызывая болезнь — по сути, создавая новый штамм?
С ответами, которые могут информировать общественную политику, этот проект был одним из 62 предложений, получивших финансирование в первом раунде грантов Массачусетского консорциума по готовности к патогенам (MassCPR).
Вирусы не могут заразить людей, если некоторые из наших клеток не впустят их. Определенные белки на поверхности клеток предоставляют доступ SARS-CoV-2, в то время как другие внутри клетки кооптируются, чтобы помочь вирусу размножаться.
Раскрывая каждый из задействованных белков, ученые открывают новые возможности для поиска лекарств, которые действуют на белки и затрудняют закрепление вируса.
Исследователи определили основных виновников, таких как рецептор ACE2 и молекулярные ножницы, известные как TMPRSS2, которые открывают приветственный коврик для нового коронавируса.Три генетика HMS внесли свой вклад в международное исследование Nature Medicine в апреле, в котором подробно описывалась активность генов, вырабатывающих эти белки, в клетках, выстилающих дыхательные пути, кровеносные сосуды, сердце, роговицу и кишечник.
Участниками были Дебора Хунг, профессор генетики Массачусетской больницы общего профиля; Кристин Зайдман, профессор медицины Томаса В. Смита в HMS и Brigham and Women’s Hospital; и Джонатан Сейдман, Генриетта Б. и Фредерик Х.Bugher Foundation Профессор генетики в HMS.
Но список генов и белков может быть еще не полным. Норберт Перримон, профессор биологии развития Джеймса Стилмана в HMS, надеется добавить к этому результаты с помощью генетического скрининга клеток плодовой мушки.
Почему летают клетки? Во-первых, геномы человека и других млекопитающих сложны; один ген может производить более одного белка, а один белок может быть произведен более чем одним геном. Геномы мух проще, что упрощает выявление истинных участников проникновения вирусов.
«Мы могли бы идентифицировать белки у мух, которые имеют эквиваленты у людей, но это было бы упущено в исследованиях на млекопитающих», — сказал Перримон.
Многие, но не все, основные биологические механизмы, обнаруженные у мух, справедливы и для других организмов, включая людей. Если команда Перримона сможет составить список генов и белков, которые, по-видимому, связаны с проникновением SARS-CoV-2 в клетки мух, исследователи могут сравнить его со списками, полученными в результате исследований на людях и млекопитающих, и использовать совпадения, чтобы сосредоточить внимание на наиболее важных факторах. вероятно, окажется важным.
Однако команде нужно сначала преодолеть несколько препятствий. Например, мухи в дикой природе не заражаются COVID-19, поэтому Перримон и научный сотрудник по генетике Рагхувир (Рам) Вишваната должны выяснить, пропускают ли клетки мухи частицы SARS-CoV-2 естественным путем. В противном случае исследователи сконструируют клетки для экспрессии человеческих ACE2 и TMPRSS2, и если это сработает, они смогут наблюдать, какие дополнительные гены задействуются, когда вирус проникает внутрь.
Например, мухи в дикой природе не заражаются COVID-19, поэтому Перримон и научный сотрудник по генетике Рагхувир (Рам) Вишваната должны выяснить, пропускают ли клетки мухи частицы SARS-CoV-2 естественным путем. В противном случае исследователи сконструируют клетки для экспрессии человеческих ACE2 и TMPRSS2, и если это сработает, они смогут наблюдать, какие дополнительные гены задействуются, когда вирус проникает внутрь.
Идея не так уж надумана.Мухи тоже не болеют гриппом, но тот же метод позволил ученым использовать насекомых, чтобы узнать, как вирус гриппа проникает в клетки.
«Если это сработает, мы должны получить некоторые интересные результаты», — сказал Перримон. «Если нет, что ж, это конец проекта».
Если клетки мух так или иначе предоставляют доступ к SARS-CoV-2, Вишваната будет использовать платформу, которую он разработал на основе инструмента редактирования генов CRISPR, чтобы быстро идентифицировать все гены, которые, по-видимому, вовлечены.
Вишваната использовал платформу в начале этого года для обнаружения белков, которые клетки мух используют для проникновения патогенов насекомых, когда ученые объявили, что некоторые из тех же белков могут быть вовлечены в инфекцию SARS-CoV-2.
Хотя исследователи не уверены, что их эксперимент удастся, у них достаточно уверенности, чтобы поверить, что стоит попробовать.
«Если бы мы смогли найти новые факторы, вовлеченные в проникновение вируса, которые привели бы к полному« списку деталей », это было бы хорошим достижением», — сказал Перримон.«Мы были бы счастливы».
Когда Дженни Ян и ее коллеги начали изучать РНК — молекулы, которые переводят инструкции ДНК в белки — у червей Caenorhabditis elegans , она не ожидала, что выводы команды могут иметь отношение к глобальной пандемии.
«Мы — лаборатория фундаментальных наук», — сказал Ян, научный сотрудник лаборатории Скотта Кеннеди, профессора генетики Филипа и Айи Ледер в HMS.«Нас интересуют такие вещи, как экспрессия генов и биология червей».
Многие РНК заканчиваются цепочкой повторяющихся адениновых оснований: так называемым поли (А) хвостом. В мае в журнале Nature группа Кеннеди сообщила, что они обнаружили ранее неизвестный тип РНК с повторяющимся урацилом и гуаниновым хвостом у C. elegans . Исследователи назвали их поли (UG) или pUG РНК.
elegans . Исследователи назвали их поли (UG) или pUG РНК.
И pUG, и поли (A) хвосты позволяют РНК рекрутировать другие белки, которые выполняют важные функции, такие как РНК-полимераза, которая создает копии молекул РНК.
На той неделе, когда исследователи разместили свою рукопись на сервере препринтов в декабре 2019 года, в Китае было зарегистрировано первое заболевание, которое сейчас известно как COVID-19. Вскоре стало ясно, что SARS-CoV-2 принадлежит к семейству вирусов, геномы которых состоят из РНК, а не ДНК.
Бесчисленные группы бросились применять знания РНК для борьбы с новой угрозой. Кеннеди интересовался, может ли его команда внести свой вклад.
Несколько исследователей в лаборатории сейчас пытаются выяснить: содержит ли коронавирус РНК pUG? Если да, то для чего он их использует? И могут ли они тогда стать мишенью для лечения?
Команда обнаружила несколько многообещающих признаков, но работа только началась, и еще не ясно, реальны ли первоначальные сигналы .
«Достаточно мотивировать, но мы еще не уверены», — сказал Ян. «Это все еще очень предварительная информация. У нас есть некоторые подсказки, которые нам нужно проверить».
Если SARS-CoV-2 действительно использует РНК pUG для рекрутирования РНК-полимеразы, ученые могут попытаться нарушить сигналы, которые прикрепляют хвост, или задействовать фермент, чтобы вирус не мог реплицироваться.
Если вирус содержит РНК pUG, но они не ведут себя так же, как у червей, Кеннеди и его коллеги имеют несколько других предположений относительно того, что могут делать молекулы и как они могут предлагать противовирусные стратегии.
И если исследователи не обнаружат никаких РНК pUG, лаборатория, скорее всего, вернет все свое внимание к работе, которую она проводила перед пандемией — если только не появится новое вдохновение.
«Это прекрасная вещь в исследованиях; вы не знаете, куда вас приведет проект», — сказал Ян.
Тестирование на текущую или прошлую инфекцию SARS-CoV-2 является краеугольным камнем понимания и контроля распространения COVID-19, но возможности и надежность тестирования в США. С. еще не удовлетворили потребности нации. Исследователи пытаются это изменить.
С. еще не удовлетворили потребности нации. Исследователи пытаются это изменить.
Лаборатория Стивена Элледжа, профессора генетики и медицины Грегора Менделя в HMS и Brigham and Women’s, обновила свой комплексный инструмент обнаружения антител VirScan, чтобы выявить прошлое воздействие SARS-CoV-2 в образцах крови и помочь иммунологам. а эпидемиологи лучше оценивают летальность вируса, уровни воздействия на население, а также краткосрочное и долгосрочное воздействие на иммунную систему. За эти усилия Элледж получила финансирование MassCPR.
Церковная лаборатория объединила общенациональные усилия по созданию более быстрых, дешевых и доступных тестов для диагностики COVID-19. То же самое и с лабораториями Конни Чепко, профессора генетики и нейробиологии Bullard в HMS, и Стивена МакКэрролла, профессора биомедицинских наук и генетики HMS Дороти и Милтона Флиер.
Брайан Рабе, кандидат биологических и биомедицинских наук в лаборатории Cepko, адаптирует существующий тест для обнаружения SARS-CoV-2 в образцах пациентов за полчаса и без необходимости в специализированном оборудовании или обучении.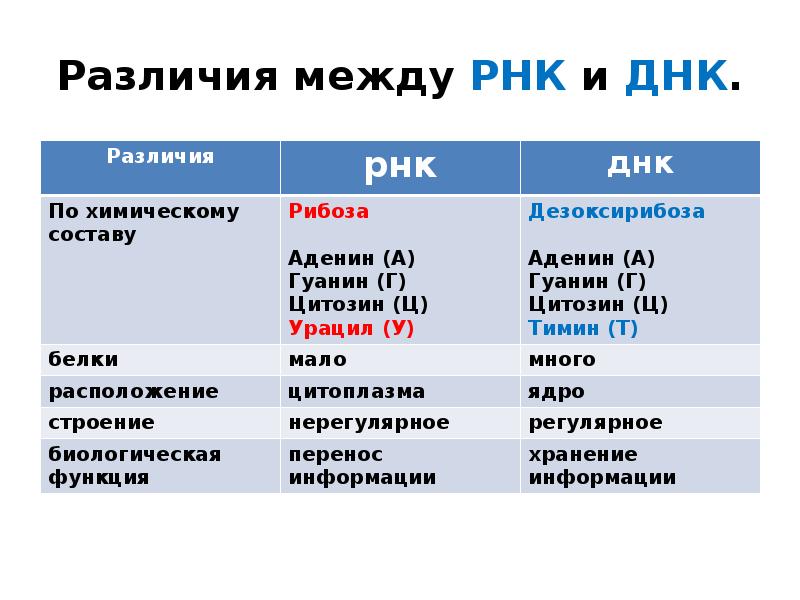 Работа основана на опыте Cepko в использовании вирусов в качестве инструментов исследования.
Работа основана на опыте Cepko в использовании вирусов в качестве инструментов исследования.
Rabe и его коллеги работают над такими улучшениями, как стабилизация и деактивация вирусных частиц в образцах пациентов. Исследователи надеются, что если они смогут внести необходимые изменения, тест в конечном итоге можно будет развернуть в кабинетах врачей и других медицинских учреждениях.
Между тем, научный сотрудник по генетике Кертис Мелло и другие члены команды МакКэрролла сосредоточились на другой технологии — нанолитровых каплях в сочетании с цифровой ПЦР — чтобы более точно измерить и охарактеризовать вирусную РНК в образцах пациентов и, таким образом, обеспечить более надежную диагностику COVID-19.
Этот метод разработан для отличия больших молекул вирусной РНК, которые продуцируются живыми реплицирующимися вирусами, от фрагментированной вирусной РНК, которая может выделяться спустя долгое время после активной инфекции.
Текущие тесты, напротив, обычно дают положительный результат, если обнаруживают любую вирусную РНК, целую или сломанную. По словам исследователей, это может объяснить по крайней мере некоторые из случаев, когда у людей положительный результат теста на COVID-19, даже если их симптомы исчезли.
По словам исследователей, это может объяснить по крайней мере некоторые из случаев, когда у людей положительный результат теста на COVID-19, даже если их симптомы исчезли.
Согласно методике МакКэрролла, отдельные образцы пациентов заключаются в микроскопические капли вместе с химическими веществами, необходимыми для усиления и обнаружения генетического материала коронавируса.Одновременно можно анализировать десятки тысяч этих капель.
Капельки спроектированы так, чтобы светиться, если они содержат РНК SARS-CoV-2. Те, которые содержат длинные неповрежденные фрагменты РНК, светятся разными цветами. Светящиеся капли подсчитываются в цифровом виде, когда они проходят через микрожидкостное устройство. По словам исследователей, цифровой подсчет должен обеспечивать более точные результаты, чем традиционная ПЦР (полимеразная цепная реакция), стандартный метод амплификации генетического материала.
Цели — помочь эпидемиологам, помочь измерить инфекционность на разных этапах болезни и быстро обнаружить активные вирусные частицы на поверхностях, чтобы люди могли сохранить пространство, безопасное для жизни и работы.
Дополнительными членами лаборатории, работающими над проектом, являются Нолан Камитаки, научный сотрудник по генетике, и Хизер де Ривера, исследователь в области естественных наук. Исследование поддержано отделом генетики HMS.
Изображения, сверху: iStock, N / A, © Martin Frouz and Jiří Svoboda, Perrimon lab, Kennedy lab, Pond5
Расшифровка структур РНК
Curr Opin Struct Biol. Авторская рукопись; доступно в PMC 2017 26 февраля.
Опубликован в окончательной отредактированной форме как:
PMCID: PMC4785074
NIHMSID: NIHMS764989
Центр личных динамических регуляторов, Медицинская школа Стэнфордского университета, Стэнфорд, Калифорния 94305, США
См. Другие статьи в PMC, в которых цитируется опубликованная статья.Abstract
Структуры молекул РНК важны для их архитектурных, регуляторных и каталитических функций. Недавние достижения в области высокопроизводительного секвенирования позволили разработать методы исследования структур РНК в масштабе всего транскриптома — так называемых структур РНК. Здесь мы рассматриваем современные технологии исследования структур РНК и выделяем выводы, полученные в результате этих исследований. Мы также указываем на пределы существующих методов и обсуждаем возможные направления для будущих улучшений.
Здесь мы рассматриваем современные технологии исследования структур РНК и выделяем выводы, полученные в результате этих исследований. Мы также указываем на пределы существующих методов и обсуждаем возможные направления для будущих улучшений.
Введение
Помимо передачи генетической информации, такой как ДНК, молекулы РНК сложены в изысканные структуры. Гибкие и динамические структуры молекул РНК лежат в основе их разнообразных функций. Подобно изучению белков, структурный анализ молекул РНК позволил разобраться в механизмах регуляции генов на самых разных уровнях. Структурные элементы в мРНК контролируют их субклеточную локализацию, а внутренний сайт входа в рибосомы (IRES) формирует трехмерные структуры, чтобы управлять кэп-независимой трансляцией (обзор [1]).Доступность мотивов в мРНК модулирует взаимодействие с РНК-связывающими белками (RBP) и микроРНК. Структура РНК также важна для некодирующих РНК. Структурные мотивы в длинных некодирующих РНК обеспечивают их ядерную локализацию, стабильность и взаимодействие с аппаратом модификации хроматина (rev. [2,3]). Уникальные структуры рибопереключателей позволяют им связывать определенные метаболиты и, в свою очередь, преобразовывать связывание лиганда в регуляторную активность генов. Третичные структуры, построенные на основных структурных элементах, необходимы рибозимам для катализа широкого спектра биохимических реакций, включая сплайсинг, лигирование и трансляцию.
[2,3]). Уникальные структуры рибопереключателей позволяют им связывать определенные метаболиты и, в свою очередь, преобразовывать связывание лиганда в регуляторную активность генов. Третичные структуры, построенные на основных структурных элементах, необходимы рибозимам для катализа широкого спектра биохимических реакций, включая сплайсинг, лигирование и трансляцию.
Традиционные методы определения структуры, такие как рентгеновская кристаллография, ядерный магнитный резонанс (ЯМР) и криоэлектронная микроскопия (крио-ЭМ), создают трехмерные структуры с высоким разрешением, но для них требуются очищенные, структурно стабильные и почти статичные молекулы РНК. Поэтому их нелегко применить для изучения подавляющего большинства РНК, которые часто находятся в нескольких конформациях, динамических, сложных и постоянно регулируемых клеточной средой [4,5].
Другой класс методов, химическое и ферментативное зондирование, решает проблему структуры РНК под другим углом.Нуклеотиды в структурах РНК имеют различную доступность в зависимости от их спаривания оснований и других взаимодействий. В химическом зондировании используются различные соединения, которые реагируют с нуклеотидами в соответствии с их окружением и генерируют объемные аддукты или точки разрыва, которые можно измерить с помощью секвенирования по Сэнгеру или высокопроизводительного секвенирования как остановки или окончания обратной транскрипции [6]. Обычно используемые химические вещества включают селективное 2′-гидроксилацилирование, анализируемое химическими веществами для удлинения праймера (SHAPE), такими как 1-метил-7-нитроизатовый ангидрид (1M7), N -метилизатовый ангидрид (NMIA), который ацилирует 2′-OH в одноцепочечные (ss) области () [7], гидроксильные радикалы, которые разрушают незащищенные фосфатные скелеты, диметилсульфат (DMS), который метилирует ss-аденозины и цитозины, 1-циклогексил- (2-морфолиноэтил) карбодиимид мет-п-толуолсульфонат ( CMCT), который модифицирует ss-уридины и гуанины, кетоксал, который модифицирует ss-гуанины, и поточное зондирование, которое использует преимущества спонтанной деградации в ss-областях.
В химическом зондировании используются различные соединения, которые реагируют с нуклеотидами в соответствии с их окружением и генерируют объемные аддукты или точки разрыва, которые можно измерить с помощью секвенирования по Сэнгеру или высокопроизводительного секвенирования как остановки или окончания обратной транскрипции [6]. Обычно используемые химические вещества включают селективное 2′-гидроксилацилирование, анализируемое химическими веществами для удлинения праймера (SHAPE), такими как 1-метил-7-нитроизатовый ангидрид (1M7), N -метилизатовый ангидрид (NMIA), который ацилирует 2′-OH в одноцепочечные (ss) области () [7], гидроксильные радикалы, которые разрушают незащищенные фосфатные скелеты, диметилсульфат (DMS), который метилирует ss-аденозины и цитозины, 1-циклогексил- (2-морфолиноэтил) карбодиимид мет-п-толуолсульфонат ( CMCT), который модифицирует ss-уридины и гуанины, кетоксал, который модифицирует ss-гуанины, и поточное зондирование, которое использует преимущества спонтанной деградации в ss-областях. Сходным образом, ферментативное зондирование использует двухцепочечные (ds) специфические РНКазы для разрезания РНК, оставляя ds / ss-специфические точки разрыва, которые указывают на статус спаривания оснований (8-11). Затем данные химического и ферментативного зондирования включаются в качестве ограничений в различные алгоритмы прогнозирования структуры РНК для повышения точности прогноза [12]. Такие экспериментальные данные существенно дополняют структурные предсказания, основанные на минимизации свободной энергии или эволюционной ко-вариации.
Сходным образом, ферментативное зондирование использует двухцепочечные (ds) специфические РНКазы для разрезания РНК, оставляя ds / ss-специфические точки разрыва, которые указывают на статус спаривания оснований (8-11). Затем данные химического и ферментативного зондирования включаются в качестве ограничений в различные алгоритмы прогнозирования структуры РНК для повышения точности прогноза [12]. Такие экспериментальные данные существенно дополняют структурные предсказания, основанные на минимизации свободной энергии или эволюционной ко-вариации.
Таблица 1
Методы глобального определения структур РНК.Методы перечислены не по лабораториям, которые их разработали, а по стратегии и используемым химическим веществам. Обычная ФОРМА очень неэффективна для анализа in vivo , но является одним из самых ранних методов исследования структуры. Ферментативное зондирование не является полезным для анализа in vivo , но обеспечивает первый глобальный анализ структурома РНК. RING-MaP и SHAPE-MaP были разработаны для анализа одиночной РНК, но эта стратегия полезна для анализа структур in vivo . HiCLIP и RPL еще не достигли глобального анализа структуром РНК, но представляют интересные направления будущего
RING-MaP и SHAPE-MaP были разработаны для анализа одиночной РНК, но эта стратегия полезна для анализа структур in vivo . HiCLIP и RPL еще не достигли глобального анализа структуром РНК, но представляют интересные направления будущего
| Методы | Зонд | Данные | Предполагаемый | Характеристики и ограничения | Ссылки |
|---|---|---|---|---|---|
| Химическое зондирование | 1M7, DMS, CMCT, гидроксил, и т. д. | 1D | 2D | Все нуклеотиды, в основном in vitro , нет информации о прямом спаривании оснований | [6,7] |
| Ферментативная | РНКаза I, V1, P1, S1 | 1D | 2D | Все нуклеотиды, только in vitro , информация о прямом спаривании оснований отсутствует | [8–11] |
| SHAPE-MaP, RING-MaP | 1M7 и DMS | 1D | 2D / 3D | Дополнительная корреляция для баз, но только на короткие дистанции.Адаптируется к in vivo и глобально | [26 •, 27 •] |
| Вторичная структура in vivo | |||||
| DMS-seq | DMS | 1D | 2D | In vivo опрашивает только A / C, нет информации о прямом сопряжении оснований | [16 ••, 17 ••, 20 ••] |
| icSHAPE | NAI, NAI-N 3 | 1D | 2D | In vivo , все нуклеотиды, минимальный фон, нет информации о прямом спаривании оснований | [18,19 ••] |
| Структура вторичного и более высокого порядка in vivo | |||||
| CLASH, hiCLIP | УФ-сшивание | 2D | In vivo , прямое дуплексное определение, только dsRBP-связанные структуры | [28,29,30 ••] | |
| RPL | Без сшивки | 2D | Lysed клетки, прямое дуплексное определение, низкая эффективность и точность | [31] | |
Несколько отличных обзоров были посвящены химии и компьютерному анализу экспериментов по химическому и ферментативному зондированию [12–15].Здесь мы сосредоточимся на применении химического зондирования in vivo в масштабе всего транскриптома . Эти методы предоставили важную информацию о физиологическом состоянии молекул РНК. Тем не менее, эти методы генерируют только одномерную усредненную структурную информацию, что далеко не достаточно для полного описания структуромы РНК. Кроме того, мы также обсуждаем недавние усилия по разработке методов, которые исследуют дальнодействующие, альтернативные и сложные структуры РНК.
Методы
in vivo РНК-структуромного зондированияНедавние достижения в области высокопроизводительного секвенирования вывели эксперименты по химическому и ферментативному зондированию в глобальный масштаб.Хотя ферментные и большинство химических зондов можно применять только к образцам РНК in vitro, некоторые из реагентов, такие как DMS и недавно разработанный NAI (имидазолид 2-метилникотиновой кислоты) и NAI – N 3 , имеют соответствующие периоды полураспада ( ∼34 мин для NAI и NAI – N 3 ) и легко проникают в клетку, таким образом обеспечивая in vivo зондирования структур РНК () [16 ••, 17 ••, 18,19 ••]. Вместе технологии высокопроизводительного секвенирования и in vivo зондирующих химикатов позволили определить структуру РНК in vivo.
Первые сообщения о in vivo структурном зондировании использовали DMS, реагент алкилирования, для модификации аденинов и цитозинов, которые обнаруживаются как остановки обратной транскрипции () [16 ••, 17 ••, 20 ••]. Хотя эти исследования отличаются некоторыми экспериментальными деталями, они схожи по концепции и основным этапам. Алкилирование ss-оснований блокирует обратную транскрипцию и, таким образом, сообщает о статусе спаривания оснований в последовательности РНК. Одним из недостатков является то, что методы на основе DMS опрашивают только две из четырех баз.Поскольку модификация оснований влияет на их способность к спариванию оснований, модификации молекул РНК титровали для достижения кинетики «однократного попадания», при которой изменяется примерно один из 200 нуклеотидов. Кинетика однократного попадания приводит к другому недостатку: большинство фрагментов РНК немодифицированы, и, таким образом, секвенирование этих РНК дает высокий фон.
Обычные реагенты SHAPE изменяют все четыре нуклеотида единообразно, но применимы только к образцам in vitro . Чтобы преодолеть это ограничение, Спитале и его коллеги синтезировали новые типы реагентов SHAPE, которые легко проникают в клетку и эффективно реагируют с одноцепочечными нуклеотидами [18].Было показано, что эти химические вещества действуют в клетках бактерий, дрожжей, мух, мышей и человека. Чтобы еще больше снизить высокий фон из-за условий модификации с однократным попаданием, Спитале и его коллеги представили активную азидную метку для ацилирующего агента NAI, которая позволяет легко очищать модифицированные фрагменты РНК с помощью системы биотин-стептавидин (icSHAPE: in vivo ). щелкните ФОРМА) [19 ••]. Этот дополнительный этап отбора значительно снижает фон и обеспечивает более чувствительный и специфический анализ модифицированных фрагментов РНК.
Поскольку каждая молекула РНК и конформация модифицируются с очень низкой частотой в экспериментах по химическому зондированию, для создания профиля структуры требуется большое количество считываний секвенирования, даже при выборе биотина. Таким образом, полученная структурная информация представляет собой среднее значение всех конформаций. Несмотря на свои ограничения, DMS и NAI – N 3 на сегодняшний день являются лучшими реагентами для химического исследования структуры РНК in vivo .
Новое понимание структуры РНК
Данные химического зондирования структур РНК обычно анализируются и используются двумя способами: зондирование и прогнозирование (PnP) и зонд и среднее значение (PnA).Для подхода PnP профили реактивности включаются в качестве ограничений в методы прогнозирования вторичной структуры для создания структурных моделей, которые наилучшим образом объясняют профиль реактивности (см. Обзор [12]). Хотя этот подход увеличивает точность предсказания структуры, надежные модели могут быть получены только для очень коротких молекул РНК или областей со стабильными единственными структурными конформациями [21]. Следовательно, применение этого подхода ко всей структуроме РНК нецелесообразно. С другой стороны, подход PnA обычно выравнивает большое количество заранее определенных мотивов последовательности РНК для получения профиля реактивности метагена, который является средним значением для каждой уже усредненной структуры РНК.Используя подход PnA, недавние исследования выявили интересные особенности РНК-структурома in vivo и предоставили много нового понимания регуляторных функций РНК-структур (). Несколько исследований также сравнили структур in vivo с структур in vitro и обнаружили особенности, которые программируются только последовательностью, и те, на которые влияет клеточная среда.
Структром РНК: новые идеи, но неполная картина. (a – i) Новые открытия о структуроме РНК из недавних исследований глобального структурного зондирования.Большинство этих структурных особенностей очевидны на уровне метагена, но не на уровне отдельного гена. (a) Пример набора треков реактивности химического зонда для типичного гена мРНК, включая зондирование in vivo, и in vitro, и разницу между ними (трек VTD). В нескольких исследованиях наблюдали меньшую структуру in vivo , чем in vitro и активное разворачивание структур in vivo [17 ••, 19 ••]. (b) Структуры в CDS, но не UTRs, демонстрируют очевидную 3-ную периодичность как in vitro , так и in vivo [8,17 ••, 19 ••, 22].Область непосредственно перед стартовым кодоном и рядом со стоп-кодоном менее структурирована, чем в среднем [16 ••, 17 ••, 19 ••]. Однако относительное содержание структуры между UTR и CDS различается у разных организмов, больше в CDS для дрожжей и растений, но больше в UTR для животных [8]. (c) Сайты паузы рибосомы, а не отрицательные контроли, демонстрируют очевидную 3-нуклеотидную периодичность перед положением, в котором расположена рибосома (обозначено E (выход), P (пептидил-тРНК) и A (аминоацил-тРНК)) [19 ••]. (d) m 6 Модификация делает окружающую последовательность менее структурированной [19 ••], чем отрицательные контроли ( Mettl3 — / — теряет больше всего m 6 A). (e) Связывание RBP изменяет химическое окружение целевой РНК, таким образом оставляя след [19]. (f) Анализ онтологии генов A. thaliana показывает, что PPV (положительная прогностическая ценность), мера того, насколько близка предсказанная структура in silico к химически исследованной структуре, является высокой для РНК, кодирующих домашние белки, но низкий для тех, кто кодирует белки реакции на стресс / стимул [16]. (g) Метагенный анализ альтернативного сплайсинга показывает более высокую структуру (меньшую реактивность) перед 5′-сайтом сплайсинга (5’SS) перед пропущенным экзоном [16]. (h) Анализ hiCLIP STAU1 в клетках человека обнаруживает большое количество вторичных структур, некоторые из которых охватывают большие расстояния [30]. (i) Метагенный анализ альтернативных сайтов полиаденилирования A. thaliana выявил картину низкой реакционной способности, за которой следует высокая реактивность [16 ••]. (j – l) Важные аспекты структуромы, которые все еще плохо изучены.Диаграммы построены в формате линейной структуры. (j) Одна и та же последовательность может принимать различные структурные состояния in vivo , которые конкурируют друг с другом или с RBP и miRNA. (k) Сложные структуры и структуры высокого порядка, т. Е. Взаимосвязанный псевдоузел. (l) Взаимодействия при спаривании оснований с большим радиусом действия, которые могут охватывать множество тысяч оснований.
Задолго до того, как стал возможен анализ структур РНК, Шабалина et al. выполнил in silico фолдинг мРНК человека и мыши и обнаружил удивительную тринуклеотидную периодичность вероятности спаривания оснований в кодирующих областях, которая отсутствует в UTRs [22].Это предсказание было сначала подтверждено in vitro, для мРНК дрожжей [8], а позже было показано, что оно является повсеместным явлением in vivo у нескольких организмов () [16 ••, 17 ••, 19 ••]. Эти исследования также сообщили о тенденции области стартового кодона, канонической или неканонической, иметь меньшую структуру, свойство, которое, вероятно, облегчает инициацию трансляции.
Применяя подход PnA к проросткам A. thaliana , Динг и его коллеги обнаружили структурные особенности мРНК вокруг альтернативных сайтов сплайсинга и полиаденилирования (16).Данные предполагают, что, помимо мотивов последовательности, структуры вокруг сайтов процессинга мРНК играют важную роль в управлении процессингом. Анализ онтологии генов показал, что мРНК A. thaliana , которые кодируют белки реакции на стресс / стимул, как правило, менее структурированы in vivo , чем те, которые кодируют домашние белки. Однако эти данные следует интерпретировать с осторожностью, поскольку известно, что содержание GC и многие другие факторы могут влиять на относительное структурное содержание молекул РНК.
Основываясь на идее, что клеточные компоненты влияют на структуры РНК и могут приводить к локальным структурным перестройкам, Spitale et al. проанализировал разницу между in vitro и in vivo данными icSHAPE и наблюдал специфические VTD ( in vivo in vitro различие) сигнатуры для нескольких мотивов последовательности [19]. Примечательно, что сигнал VTD позволил детектировать фокальные перестройки структур, индуцированные связыванием RBP, так что присутствие или отсутствие VTD в каждом случае мотива связывания RBP можно использовать для прогнозирования связывания RBP in vivo ().Эта концепция структурного отпечатка РНК аналогична «отпечатку» фактора транскрипции на ДНК. Подобно подходу VTD, Smola et al. использовал 1M7 для обнаружения сайтов связывания с белками на нескольких многочисленных РНК, сравнивая статус модификации между в целлюлозе и ex vivo в условиях [23]. Вместе эти исследования подчеркивают мощь методов, основанных на SHAPE, во взаимодействиях РНК-белок. Spitale et al. далее обнаружил, что модификация РНК N 6 -метиладенозин (m 6 A) является структурным переключателем in vivo , который приводит к распариванию дуплексов РНК () [19 ••]. in vitro анализов и подробная биофизическая характеристика [24,25].Удивительно, хотя in silico и in vitro исследований структуры РНК выявили более значительную структуру вокруг сайтов паузы рибосом [8], этот паттерн не был обнаружен в исследованиях in vivo [17 ••, 19 ••]. Это могло отражать влияние клеточной среды на структуру РНК.
Последние достижения in vitro Методы картирования структуры РНК могут дополнять данные in vivo о структуре РНК. Систематическая «мутация-и-карта» может идентифицировать специфические взаимодействия спаривания оснований и может быть объединена с анализом мультиплексированного гидроксильного радикального расщепления третичной близости для моделирования трехмерных структур РНК с разрешением, близким к атомному [32].Мощная стратегия в будущем может состоять в том, чтобы номинировать структурные мотивы РНК по in vivo, структурным данным, с последующим детальным рассечением in vitro, и 3D-моделированием лучших кандидатов.
Прямое определение спиралей и сложных и динамических структур
Одномерные данные ферментативного и химического исследования структур РНК предоставили интересную информацию о коллективных особенностях структур РНК, но их прямое использование в моделировании структуры было довольно трудным.Во-первых, взаимодействия пар оснований, предсказанные с помощью данных о реактивности, далеки от точности, особенно для дальнодействующих взаимодействий и сложных псевдоузлов. Во-вторых, структуры большинства молекул РНК в живых клетках неоднородны, гибки и динамичны, и поэтому эти структуры лучше описывать как ансамбль. Однако современные методы химического зондирования in vivo структуры РНК позволяют определять среднюю структуру в динамической клеточной среде. Разложение ансамбля структур на их индивидуальные состояния из одномерных данных нецелесообразно.В нескольких недавних исследованиях эти проблемы начали решаться с использованием новых вычислительных и экспериментальных подходов.
Нуклеотиды, которые взаимодействуют друг с другом, демонстрируют коррелированную химическую реактивность, и эту корреляцию можно использовать для обнаружения взаимодействий и классификации различных структурных состояний. Уикс и его коллеги недавно реализовали этот принцип в двух методах: SHAPE-Mutational Profiling (SHAPE-MaP) и RNA INtecting Group-MaP (RING-MaP) [26 •, 27 •]. В обоих методах используются особые условия для облегчения считывания обратной транскрипции на модифицированных нуклеотидах для введения мутаций, обычно более одной для каждой матрицы РНК.Корреляционный и кластерный анализ мутаций в секвенированных считываниях создает ограничения для построения 2D и 3D моделей исследуемой РНК. Эти методы были применены к нескольким свернутым РНК in vitro и дали улучшенные структурные модели. Хотя эта стратегия была протестирована только на нескольких молекулах, они потенциально могут быть адаптированы ко всему транскриптому. Однако использование этих методов для прямой деконволюции больших и сложных РНК может быть проблематичным.
Прямая информация о спаривании оснований также может быть получена путем измерения физической близости между нуклеотидами.Толлервей и его коллеги обнаружили лигированные фрагменты РНК при перекрестном связывании и секвенировании дцРНК, которые связываются белками. Основываясь на этом открытии, они разработали метод под названием Crosslinking, Ligation and Sequencing Hybrids (CLASH) для анализа структур РНК и взаимодействий РНК-РНК [28,29]. Sugimoto и его коллеги применили этот принцип к iCLIP, разработав гибридный iCLIP (hiCLIP) метод, чтобы идентифицировать структуры, связанные с dsRBP STAU1 [30]. В этом подходе фрагменты дцРНК, связанные с STAU1, захватываются с помощью лигирования близости, и каждое секвенированное считывание представляет собой измерение структуры одной молекулы.Метод hiCLIP выявил множество связанных с STAU1 дальнодействующих взаимодействий, разделенных тысячами или более оснований на линейном транскрипте, которые особенно трудно обнаружить с помощью обычных методов. Поскольку каждое считывание секвенирования происходит из одной структурной конформации, лигирование близости также позволяет обнаруживать индивидуальные структурные состояния в небольшом подмножестве структуромы, а не в среднем по структурному ансамблю [30]. Одним из основных ограничений методов CLASH / hiCLIP является низкий процент чтений, которые предоставляют прямую контактную информацию (менее 2%).Поскольку большинство эукариот кодируют большое количество RBP (на самом деле большее количество, чем ДНК-связывающих факторов транскрипции), применение hiCLIP к ним один за другим является трудоемким. Более того, многие структуры РНК вообще не связаны с RBP и поэтому их невозможно идентифицировать с помощью hiCLIP.
Подобно использованию лигирования близости для захвата структур РНК, Ramani и Shendure сообщили о более общем методе, RNA Proximity Ligation (RPL) [31]. В этом подходе использовалось расщепление РНК in situ РНКазой и лигирование по близости для соединения дуплексов в физической близости.Результаты, подтверждающие концепцию, были представлены для нескольких очень распространенных некодирующих РНК, таких как рибосомные РНК и мяРНК. Однако, поскольку сшивание не используется и клетки либо перевариваются эндогенными нуклеазами, либо лизируются и расщепляются экзогенными нуклеазами, случайные лигирования приводят к низкой точности и высокому шуму. Поскольку отбор не производится, процент химерных фрагментов очень низок (менее 0,5%), что затрудняет масштабирование метода для применения в масштабе всего транскриптома.Хотя методы на основе корреляции и близости-лигирования, описанные здесь, еще не достигли анализа структур целых РНК, они представляют собой интересные попытки с разных точек зрения, кроме методов химического зондирования.
Резюме и перспективы на будущее
Полное понимание структуры РНК требует полного описания структур и их динамики. Недавние разработки методов химического зондирования на основе секвенирования позволили получить глобальный обзор профилей локальной структуры РНК.Ожидается, что дальнейшее развитие методов обнаружения пар оснований предоставит столь необходимую информацию о более сложных и динамичных архитектурах РНК. Как специфические нуклеиновые кислоты, так и сшивающие белок-РНК могут быть дополнительно исследованы для нацеливания на различные виды вторичных или третичных структур. Комбинация этих двух различных классов экспериментальных методов необходима для получения полной картины структуры РНК, от нуклеотидов до высокоуровневых архитектур. Хотя 3D-моделирование структур РНК в настоящее время ограничено короткими РНК [32], интеграция методов химического зондирования и перекрестного связывания должна помочь деконволюции структурных ансамблей больших РНК и облегчить изоляцию структурных доменов, чтобы к ним можно было применить 3D-моделирование.Филогенетический анализ структур РНК часто подтверждает их функциональную важность, но страдает от редкости ковариации в большинстве РНК. Интеграция филогенетического анализа с экспериментальными методами объединит сильные стороны обоих подходов и поможет понять функции вновь идентифицированных структур РНК.
Долгосрочная цель изучения структур РНК состоит в том, чтобы не только создать каталог структур, но и сделать вывод об их функциях на основе классификации и корреляции с известными мотивами последовательностей и связыванием белков, онтологией генов, а затем определить их приоритетность для функциональные и механистические исследования.Во многих биологических процессах структуры РНК можно рассматривать как центр, который объединяет входные данные от клеточных компонентов и внеклеточной среды. Нарушения базовой последовательности или этих входных источников регулируют важные функции, основанные на РНК, или приводят к заболеваниям. Например, стрессовые условия способствуют избирательной трансляции мРНК, процессу, который зависит от структур мРНК [33]. Более того, мутации во многих РНК-геликазах приводят к раку [34]. Применение методов определения структуры РНК к этим разнообразным проблемам прольет свет как на основные механизмы экспрессии генов, так и на потенциальные терапевтические возможности для лечения заболеваний.
Благодарности
Мы благодарим сотрудников лаборатории Чанг за обсуждение и приносим свои извинения коллегам, чьи работы не обсуждаются из-за нехватки места. Мы благодарим NIH (R01-HG004361 и P50-HG007735) и Калифорнийский институт регенеративной медицины (HYC) за поддержку. З.Л. является стипендиатом семьи Лейтон в рамках стипендии Фонда Дэймона Руньона-Сона по педиатрическому онкологическому лечению (DRSG-14-15).
Сноски
Заявление о конфликте интересов : HYC является изобретателем по патенту Стэнфордского университета на технологию in vivo SHAPE.
Ссылки и рекомендуемая литература
Документы, представляющие особый интерес, опубликованные в течение периода обзора, были выделены следующим образом:
• особый интерес
•• представляющий особый интерес
1. Mortimer SA, Kidwell MA, Doudna JA. Понимание структуры и функции РНК из полногеномных исследований. Nat Rev Genet. 2014; 15: 469–479. [PubMed] [Google Scholar] 3. Ван И, Кертес М., Спитале Р.С., Сигал Э., Чанг Х.Й. Понимание транскриптома через структуру РНК.Nat Rev Genet. 2011; 12: 641–655. [Бесплатная статья PMC] [PubMed] [Google Scholar] 4. Wiebe NJ, Meyer IM. TRANSAT — метод обнаружения консервативных спиралей функциональных структур РНК, включая временные, псевдузловые и альтернативные структуры. PLoS Comput Biol. 2010; 6: e1000823. [Бесплатная статья PMC] [PubMed] [Google Scholar] 5. Jaeger JA, SantaLucia J, Jr, Tinoco I., Jr Определение структуры РНК и термодинамики. Анну Рев Биохим. 1993. 62: 255–287. [PubMed] [Google Scholar] 6. Ehresmann C, Baudin F, Mougel M, Romby P, Ebel JP, Ehresmann B.Исследование структуры РНК в растворе. Nucleic Acids Res. 1987; 15: 9109–9128. [Бесплатная статья PMC] [PubMed] [Google Scholar] 7. Меринос Э.Дж., Уилкинсон К.А., Кофлан Д.Л., Недели КМ. Анализ структуры РНК при разрешении одного нуклеотида путем селективного 2′-гидроксилацилирования и удлинения праймера (SHAPE) J Am Chem Soc. 2005; 127: 4223–4231. [PubMed] [Google Scholar] 8. Kertesz M, Wan Y, Mazor E, Rinn JL, Nutter RC, Chang HY, Segal E. Измерение вторичной структуры РНК в дрожжах по всему геному. Природа. 2010; 467: 103–107.[Бесплатная статья PMC] [PubMed] [Google Scholar] 9. Ван И, Цюй К., Чжан К.К., Флинн Р.А., Поместье О, Оуян З., Чжан Дж., Спитале Р.С., Снайдер М.П., Сигал Э. и др. Пейзаж и вариации вторичной структуры РНК в транскриптоме человека. Природа. 2014; 505: 706–709. [Бесплатная статья PMC] [PubMed] [Google Scholar] 10. Андервуд Дж. Г., Узилов А. В., Кацман С., Онодера С. С., Майнцер Дж. Э., Мэтьюз Д. Х., Лоу TM, Салама С. Р., Хаусслер Д. FragSeq: исследование структуры РНК по всему транскриптому с использованием высокопроизводительного секвенирования. Нат методы.2010; 7: 995–1001. [Бесплатная статья PMC] [PubMed] [Google Scholar] 11. Чжэн К., Рывкин П., Ли Ф, Драгомир И., Валладарес О, Ян Дж., Цао К., Ван Л.С., Грегори Б.Д. Полногеномное секвенирование двухцепочечной РНК показывает функциональное значение РНК с парными основаниями у Arabidopsis. PLoS Genet. 2010; 6: e1001141. [Бесплатная статья PMC] [PubMed] [Google Scholar] 12. Ситин MG, Мэтьюз DH. Прогнозирование структуры РНК: обзор методов. Методы Мол биол. 2012; 905: 99–122. [PubMed] [Google Scholar] 13. Kwok CK, Tang Y, Assmann SM, Bevilacqua PC.Структурома РНК: исследование структуры транскриптома с секвенированием следующего поколения. Trends Biochem Sci. 2015; 40: 221–232. [PubMed] [Google Scholar] 14. Spitale RC, Флинн Р.А., Торре Е.А., Kool ET, Chang HY. Структурный анализ РНК путем развития химии SHAPE. Wiley Interdiscip Rev RNA. 2014; 5: 867–881. [Бесплатная статья PMC] [PubMed] [Google Scholar] 16 ••. Дин И, Тан И, Квок С.К., Чжан И, Бевилаква П.К., Ассманн С.М. Полногеномное профилирование вторичной структуры РНК in vivo выявляет новые регуляторные особенности.Природа. 2014; 505: 696–700. Эта статья вместе с работами [17 ••, 20 ••] объединили модификацию РНК с помощью DMS и высокопроизводительное секвенирование, чтобы впервые проанализировать структуру РНК in vivo . Эти анализы выявили много интересных структурных особенностей, связанных с отдельными функциональными мотивами и принципами, которые управляют формированием структуры в клетках. [PubMed] [Google Scholar] 17 ••. Рускин С, Зубрадт М, Вашитль С, Келлис М, Вайсман Дж. Полногеномное зондирование структуры РНК выявляет активное развертывание структур мРНК in vivo.Природа. 2014; 505: 701–705. См. Аннотацию к исх. [16 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 19 ••. Spitale RC, Flynn RA, Zhang QC, Crisalli P, Lee B, Jung JW, Kuchelmeister HY, Batista PJ, Torre EA, Kool ET, et al. Структурные импринты in vivo расшифровывают механизмы регуляции РНК. Природа. 2015; 519: 486–490. В этой статье сообщается о недавно разработанном реагенте SHAPE, который обеспечивает очистку и селективное секвенирование модифицированных фрагментов РНК, повышая специфичность и чувствительность in vivo SHAPE.Резкое повышение точности зондирования позволило изучить генетически запрограммированные структурные особенности РНК и структурные отпечатки связывания RBP и модификаций РНК. [Бесплатная статья PMC] [PubMed] [Google Scholar] 20 ••. Talkish J, May G, Lin Y, Woolford JL, Jr, McManus CJ. Mod-seq: высокопроизводительное секвенирование для химического исследования структуры РНК. РНК. 2014; 20: 713–720. См. Аннотацию к исх. [16 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 21. Кладванг В., ВанЛанг СС, Кордеро П., Дас Р.Понимание ошибок моделирования структуры РНК с помощью SHAPE. Биохимия. 2011; 50: 8049–8056. [Бесплатная статья PMC] [PubMed] [Google Scholar] 22. Шабалина С.А., Огурцов А.Ю., Спиридонов Н.А. Периодический паттерн вторичной структуры мРНК, созданный генетическим кодом. Nucleic Acids Res. 2006; 34: 2428–2437. [Бесплатная статья PMC] [PubMed] [Google Scholar] 23. Смола MJ, Calabrese JM, Weeks KM. Обнаружение РНК-белковых взаимодействий в живых клетках с помощью SHAPE. Биохимия. 2015; 54: 6867–6875. [Бесплатная статья PMC] [PubMed] [Google Scholar] 24.Liu N, Dai Q, Zheng G, He C, Parisien M, Pan T. N (6) -метиладенозин-зависимые структурные переключатели РНК регулируют РНК-белковые взаимодействия. Природа. 2015; 518: 560–564. [Бесплатная статья PMC] [PubMed] [Google Scholar] 25. Руст К., Линч С.Р., Батиста П.Дж., Ку К., Чанг Х.Й., Кул Э. Структура и термодинамика N6-метиладенозина в РНК: подпружиненная модификация основания. J Am Chem Soc. 2015; 137: 2107–2115. [Бесплатная статья PMC] [PubMed] [Google Scholar] 26 •. Хоман П.Дж., Фаворов О.В., Лаванда К.А., Курсун О, Ге X, Пусан С, Дохолян Н.В., Недели КМ.Одномолекулярное коррелированное химическое зондирование РНК. Proc Natl Acad Sci U S. A. 2014; 111: 13858–13863. Эта статья вместе с Ref. [27 •] сообщает о новых условиях химического зондирования, которые вводят мутации в сайты модификаций. Кроме того, они используют корреляционный анализ для выявления контактов в пространстве и кластерный анализ для выявления различных состояний в структурном ансамбле. [Бесплатная статья PMC] [PubMed] [Google Scholar] 27 •. Зигфрид Н.А., Пусан С., Райс ГМ, Нельсон Дж. А., Недели КМ. Обнаружение мотивов РНК с помощью методов SHAPE и мутационного профилирования (SHAPE-MaP) Nat.2014; 11: 959–965. См. Аннотацию к исх. [26 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 28. Helwak A, Kudla G, Dudnakova T, Tollervey D. Картирование взаимодействия miRNA человека с помощью CLASH обнаруживает частое неканоническое связывание. Клетка. 2013; 153: 654–665. [Бесплатная статья PMC] [PubMed] [Google Scholar] 29. Kudla G, Granneman S, Hahn D, Beggs JD, Tollervey D. Перекрестное связывание, лигирование и секвенирование гибридов выявляют взаимодействия РНК-РНК в дрожжах. Proc Natl Acad Sci U S. A. 2011; 108: 10010–10015. [Бесплатная статья PMC] [PubMed] [Google Scholar] 30 ••.Sugimoto Y, Vigilante A, Darbo E, Zirra A, Militti C, D’Ambrogio A, Luscombe NM, Ule J. hiCLIP раскрывает in vivo атлас вторичных структур мРНК, распознаваемых Staufen 1. Nature. 2015; 519: 491–494. В этой статье используется перекрестное связывание и лигирование по близости для определения структур РНК, связанных с dsRBP STAU1, и выявлены дальнодействующие и альтернативные структуры РНК, которые невозможно найти с помощью методов химического зондирования. [Бесплатная статья PMC] [PubMed] [Google Scholar] 31. Рамани В., Цю Р., Шендурэ Дж.Высокопроизводительное определение структуры РНК путем лигирования близости. Nat Biotechnol. 2015; 33: 980–984. [Бесплатная статья PMC] [PubMed] [Google Scholar] 32. Cheng CY, Chou FC, Kladwang W, Tian S, Cordero P, Das R. Согласованные глобальные структуры сложных состояний РНК посредством многомерного химического картирования. Элиф. 2015; 4: e07600. [Бесплатная статья PMC] [PubMed] [Google Scholar] 33. Thakor N, Holcik M. IRES-опосредованная трансляция клеточной информационной РНК работает независимо от eIF2alpha во время стресса. Nucleic Acids Res.2012; 40: 541–552. [Бесплатная статья PMC] [PubMed] [Google Scholar]Расшифровка структур РНК
Curr Opin Struct Biol. Авторская рукопись; доступно в PMC 2017 26 февраля.
Опубликован в окончательной отредактированной форме как:
PMCID: PMC4785074
NIHMSID: NIHMS764989
Центр личных динамических регуляторов, Медицинская школа Стэнфордского университета, Стэнфорд, Калифорния 94305, США
См. Другие статьи в PMC, в которых цитируется опубликованная статья.Abstract
Структуры молекул РНК важны для их архитектурных, регуляторных и каталитических функций.Недавние достижения в области высокопроизводительного секвенирования позволили разработать методы исследования структур РНК в масштабе всего транскриптома — так называемых структур РНК. Здесь мы рассматриваем современные технологии исследования структур РНК и выделяем выводы, полученные в результате этих исследований. Мы также указываем на пределы существующих методов и обсуждаем возможные направления для будущих улучшений.
Введение
Помимо передачи генетической информации, такой как ДНК, молекулы РНК сложены в изысканные структуры.Гибкие и динамические структуры молекул РНК лежат в основе их разнообразных функций. Подобно изучению белков, структурный анализ молекул РНК позволил разобраться в механизмах регуляции генов на самых разных уровнях. Структурные элементы в мРНК контролируют их субклеточную локализацию, а внутренний сайт входа в рибосомы (IRES) формирует трехмерные структуры, чтобы управлять кэп-независимой трансляцией (обзор [1]). Доступность мотивов в мРНК модулирует взаимодействие с РНК-связывающими белками (RBP) и микроРНК.Структура РНК также важна для некодирующих РНК. Структурные мотивы в длинных некодирующих РНК обеспечивают их ядерную локализацию, стабильность и взаимодействие с аппаратом модификации хроматина (rev. [2,3]). Уникальные структуры рибопереключателей позволяют им связывать определенные метаболиты и, в свою очередь, преобразовывать связывание лиганда в регуляторную активность генов. Третичные структуры, построенные на основных структурных элементах, необходимы рибозимам для катализа широкого спектра биохимических реакций, включая сплайсинг, лигирование и трансляцию.
Традиционные методы определения структуры, такие как рентгеновская кристаллография, ядерный магнитный резонанс (ЯМР) и криоэлектронная микроскопия (крио-ЭМ), создают трехмерные структуры с высоким разрешением, но для них требуются очищенные, структурно стабильные и почти статичные молекулы РНК. Поэтому их нелегко применить для изучения подавляющего большинства РНК, которые часто находятся в нескольких конформациях, динамических, сложных и постоянно регулируемых клеточной средой [4,5].
Другой класс методов, химическое и ферментативное зондирование, решает проблему структуры РНК под другим углом.Нуклеотиды в структурах РНК имеют различную доступность в зависимости от их спаривания оснований и других взаимодействий. В химическом зондировании используются различные соединения, которые реагируют с нуклеотидами в соответствии с их окружением и генерируют объемные аддукты или точки разрыва, которые можно измерить с помощью секвенирования по Сэнгеру или высокопроизводительного секвенирования как остановки или окончания обратной транскрипции [6]. Обычно используемые химические вещества включают селективное 2′-гидроксилацилирование, анализируемое химическими веществами для удлинения праймера (SHAPE), такими как 1-метил-7-нитроизатовый ангидрид (1M7), N -метилизатовый ангидрид (NMIA), который ацилирует 2′-OH в одноцепочечные (ss) области () [7], гидроксильные радикалы, которые разрушают незащищенные фосфатные скелеты, диметилсульфат (DMS), который метилирует ss-аденозины и цитозины, 1-циклогексил- (2-морфолиноэтил) карбодиимид мет-п-толуолсульфонат ( CMCT), который модифицирует ss-уридины и гуанины, кетоксал, который модифицирует ss-гуанины, и поточное зондирование, которое использует преимущества спонтанной деградации в ss-областях.Сходным образом, ферментативное зондирование использует двухцепочечные (ds) специфические РНКазы для разрезания РНК, оставляя ds / ss-специфические точки разрыва, которые указывают на статус спаривания оснований (8-11). Затем данные химического и ферментативного зондирования включаются в качестве ограничений в различные алгоритмы прогнозирования структуры РНК для повышения точности прогноза [12]. Такие экспериментальные данные существенно дополняют структурные предсказания, основанные на минимизации свободной энергии или эволюционной ко-вариации.
Таблица 1
Методы глобального определения структур РНК.Методы перечислены не по лабораториям, которые их разработали, а по стратегии и используемым химическим веществам. Обычная ФОРМА очень неэффективна для анализа in vivo , но является одним из самых ранних методов исследования структуры. Ферментативное зондирование не является полезным для анализа in vivo , но обеспечивает первый глобальный анализ структурома РНК. RING-MaP и SHAPE-MaP были разработаны для анализа одиночной РНК, но эта стратегия полезна для анализа структур in vivo . HiCLIP и RPL еще не достигли глобального анализа структуром РНК, но представляют интересные направления будущего
| Методы | Зонд | Данные | Предполагаемый | Характеристики и ограничения | Ссылки |
|---|---|---|---|---|---|
| Химическое зондирование | 1M7, DMS, CMCT, гидроксил, и т. д. | 1D | 2D | Все нуклеотиды, в основном in vitro , нет информации о прямом спаривании оснований | [6,7] |
| Ферментативная | РНКаза I, V1, P1, S1 | 1D | 2D | Все нуклеотиды, только in vitro , информация о прямом спаривании оснований отсутствует | [8–11] |
| SHAPE-MaP, RING-MaP | 1M7 и DMS | 1D | 2D / 3D | Дополнительная корреляция для баз, но только на короткие дистанции.Адаптируется к in vivo и глобально | [26 •, 27 •] |
| Вторичная структура in vivo | |||||
| DMS-seq | DMS | 1D | 2D | In vivo опрашивает только A / C, нет информации о прямом сопряжении оснований | [16 ••, 17 ••, 20 ••] |
| icSHAPE | NAI, NAI-N 3 | 1D | 2D | In vivo , все нуклеотиды, минимальный фон, нет информации о прямом спаривании оснований | [18,19 ••] |
| Структура вторичного и более высокого порядка in vivo | |||||
| CLASH, hiCLIP | УФ-сшивание | 2D | In vivo , прямое дуплексное определение, только dsRBP-связанные структуры | [28,29,30 ••] | |
| RPL | Без сшивки | 2D | Lysed клетки, прямое дуплексное определение, низкая эффективность и точность | [31] | |
Несколько отличных обзоров были посвящены химии и компьютерному анализу экспериментов по химическому и ферментативному зондированию [12–15].Здесь мы сосредоточимся на применении химического зондирования in vivo в масштабе всего транскриптома . Эти методы предоставили важную информацию о физиологическом состоянии молекул РНК. Тем не менее, эти методы генерируют только одномерную усредненную структурную информацию, что далеко не достаточно для полного описания структуромы РНК. Кроме того, мы также обсуждаем недавние усилия по разработке методов, которые исследуют дальнодействующие, альтернативные и сложные структуры РНК.
Методы
in vivo РНК-структуромного зондированияНедавние достижения в области высокопроизводительного секвенирования вывели эксперименты по химическому и ферментативному зондированию в глобальный масштаб.Хотя ферментные и большинство химических зондов можно применять только к образцам РНК in vitro, некоторые из реагентов, такие как DMS и недавно разработанный NAI (имидазолид 2-метилникотиновой кислоты) и NAI – N 3 , имеют соответствующие периоды полураспада ( ∼34 мин для NAI и NAI – N 3 ) и легко проникают в клетку, таким образом обеспечивая in vivo зондирования структур РНК () [16 ••, 17 ••, 18,19 ••]. Вместе технологии высокопроизводительного секвенирования и in vivo зондирующих химикатов позволили определить структуру РНК in vivo.
Первые сообщения о in vivo структурном зондировании использовали DMS, реагент алкилирования, для модификации аденинов и цитозинов, которые обнаруживаются как остановки обратной транскрипции () [16 ••, 17 ••, 20 ••]. Хотя эти исследования отличаются некоторыми экспериментальными деталями, они схожи по концепции и основным этапам. Алкилирование ss-оснований блокирует обратную транскрипцию и, таким образом, сообщает о статусе спаривания оснований в последовательности РНК. Одним из недостатков является то, что методы на основе DMS опрашивают только две из четырех баз.Поскольку модификация оснований влияет на их способность к спариванию оснований, модификации молекул РНК титровали для достижения кинетики «однократного попадания», при которой изменяется примерно один из 200 нуклеотидов. Кинетика однократного попадания приводит к другому недостатку: большинство фрагментов РНК немодифицированы, и, таким образом, секвенирование этих РНК дает высокий фон.
Обычные реагенты SHAPE изменяют все четыре нуклеотида единообразно, но применимы только к образцам in vitro . Чтобы преодолеть это ограничение, Спитале и его коллеги синтезировали новые типы реагентов SHAPE, которые легко проникают в клетку и эффективно реагируют с одноцепочечными нуклеотидами [18].Было показано, что эти химические вещества действуют в клетках бактерий, дрожжей, мух, мышей и человека. Чтобы еще больше снизить высокий фон из-за условий модификации с однократным попаданием, Спитале и его коллеги представили активную азидную метку для ацилирующего агента NAI, которая позволяет легко очищать модифицированные фрагменты РНК с помощью системы биотин-стептавидин (icSHAPE: in vivo ). щелкните ФОРМА) [19 ••]. Этот дополнительный этап отбора значительно снижает фон и обеспечивает более чувствительный и специфический анализ модифицированных фрагментов РНК.
Поскольку каждая молекула РНК и конформация модифицируются с очень низкой частотой в экспериментах по химическому зондированию, для создания профиля структуры требуется большое количество считываний секвенирования, даже при выборе биотина. Таким образом, полученная структурная информация представляет собой среднее значение всех конформаций. Несмотря на свои ограничения, DMS и NAI – N 3 на сегодняшний день являются лучшими реагентами для химического исследования структуры РНК in vivo .
Новое понимание структуры РНК
Данные химического зондирования структур РНК обычно анализируются и используются двумя способами: зондирование и прогнозирование (PnP) и зонд и среднее значение (PnA).Для подхода PnP профили реактивности включаются в качестве ограничений в методы прогнозирования вторичной структуры для создания структурных моделей, которые наилучшим образом объясняют профиль реактивности (см. Обзор [12]). Хотя этот подход увеличивает точность предсказания структуры, надежные модели могут быть получены только для очень коротких молекул РНК или областей со стабильными единственными структурными конформациями [21]. Следовательно, применение этого подхода ко всей структуроме РНК нецелесообразно. С другой стороны, подход PnA обычно выравнивает большое количество заранее определенных мотивов последовательности РНК для получения профиля реактивности метагена, который является средним значением для каждой уже усредненной структуры РНК.Используя подход PnA, недавние исследования выявили интересные особенности РНК-структурома in vivo и предоставили много нового понимания регуляторных функций РНК-структур (). Несколько исследований также сравнили структур in vivo с структур in vitro и обнаружили особенности, которые программируются только последовательностью, и те, на которые влияет клеточная среда.
Структром РНК: новые идеи, но неполная картина. (a – i) Новые открытия о структуроме РНК из недавних исследований глобального структурного зондирования.Большинство этих структурных особенностей очевидны на уровне метагена, но не на уровне отдельного гена. (a) Пример набора треков реактивности химического зонда для типичного гена мРНК, включая зондирование in vivo, и in vitro, и разницу между ними (трек VTD). В нескольких исследованиях наблюдали меньшую структуру in vivo , чем in vitro и активное разворачивание структур in vivo [17 ••, 19 ••]. (b) Структуры в CDS, но не UTRs, демонстрируют очевидную 3-ную периодичность как in vitro , так и in vivo [8,17 ••, 19 ••, 22].Область непосредственно перед стартовым кодоном и рядом со стоп-кодоном менее структурирована, чем в среднем [16 ••, 17 ••, 19 ••]. Однако относительное содержание структуры между UTR и CDS различается у разных организмов, больше в CDS для дрожжей и растений, но больше в UTR для животных [8]. (c) Сайты паузы рибосомы, а не отрицательные контроли, демонстрируют очевидную 3-нуклеотидную периодичность перед положением, в котором расположена рибосома (обозначено E (выход), P (пептидил-тРНК) и A (аминоацил-тРНК)) [19 ••]. (d) m 6 Модификация делает окружающую последовательность менее структурированной [19 ••], чем отрицательные контроли ( Mettl3 — / — теряет больше всего m 6 A). (e) Связывание RBP изменяет химическое окружение целевой РНК, таким образом оставляя след [19]. (f) Анализ онтологии генов A. thaliana показывает, что PPV (положительная прогностическая ценность), мера того, насколько близка предсказанная структура in silico к химически исследованной структуре, является высокой для РНК, кодирующих домашние белки, но низкий для тех, кто кодирует белки реакции на стресс / стимул [16]. (g) Метагенный анализ альтернативного сплайсинга показывает более высокую структуру (меньшую реактивность) перед 5′-сайтом сплайсинга (5’SS) перед пропущенным экзоном [16]. (h) Анализ hiCLIP STAU1 в клетках человека обнаруживает большое количество вторичных структур, некоторые из которых охватывают большие расстояния [30]. (i) Метагенный анализ альтернативных сайтов полиаденилирования A. thaliana выявил картину низкой реакционной способности, за которой следует высокая реактивность [16 ••]. (j – l) Важные аспекты структуромы, которые все еще плохо изучены.Диаграммы построены в формате линейной структуры. (j) Одна и та же последовательность может принимать различные структурные состояния in vivo , которые конкурируют друг с другом или с RBP и miRNA. (k) Сложные структуры и структуры высокого порядка, т. Е. Взаимосвязанный псевдоузел. (l) Взаимодействия при спаривании оснований с большим радиусом действия, которые могут охватывать множество тысяч оснований.
Задолго до того, как стал возможен анализ структур РНК, Шабалина et al. выполнил in silico фолдинг мРНК человека и мыши и обнаружил удивительную тринуклеотидную периодичность вероятности спаривания оснований в кодирующих областях, которая отсутствует в UTRs [22].Это предсказание было сначала подтверждено in vitro, для мРНК дрожжей [8], а позже было показано, что оно является повсеместным явлением in vivo у нескольких организмов () [16 ••, 17 ••, 19 ••]. Эти исследования также сообщили о тенденции области стартового кодона, канонической или неканонической, иметь меньшую структуру, свойство, которое, вероятно, облегчает инициацию трансляции.
Применяя подход PnA к проросткам A. thaliana , Динг и его коллеги обнаружили структурные особенности мРНК вокруг альтернативных сайтов сплайсинга и полиаденилирования (16).Данные предполагают, что, помимо мотивов последовательности, структуры вокруг сайтов процессинга мРНК играют важную роль в управлении процессингом. Анализ онтологии генов показал, что мРНК A. thaliana , которые кодируют белки реакции на стресс / стимул, как правило, менее структурированы in vivo , чем те, которые кодируют домашние белки. Однако эти данные следует интерпретировать с осторожностью, поскольку известно, что содержание GC и многие другие факторы могут влиять на относительное структурное содержание молекул РНК.
Основываясь на идее, что клеточные компоненты влияют на структуры РНК и могут приводить к локальным структурным перестройкам, Spitale et al. проанализировал разницу между in vitro и in vivo данными icSHAPE и наблюдал специфические VTD ( in vivo in vitro различие) сигнатуры для нескольких мотивов последовательности [19]. Примечательно, что сигнал VTD позволил детектировать фокальные перестройки структур, индуцированные связыванием RBP, так что присутствие или отсутствие VTD в каждом случае мотива связывания RBP можно использовать для прогнозирования связывания RBP in vivo ().Эта концепция структурного отпечатка РНК аналогична «отпечатку» фактора транскрипции на ДНК. Подобно подходу VTD, Smola et al. использовал 1M7 для обнаружения сайтов связывания с белками на нескольких многочисленных РНК, сравнивая статус модификации между в целлюлозе и ex vivo в условиях [23]. Вместе эти исследования подчеркивают мощь методов, основанных на SHAPE, во взаимодействиях РНК-белок. Spitale et al. далее обнаружил, что модификация РНК N 6 -метиладенозин (m 6 A) является структурным переключателем in vivo , который приводит к распариванию дуплексов РНК () [19 ••]. in vitro анализов и подробная биофизическая характеристика [24,25].Удивительно, хотя in silico и in vitro исследований структуры РНК выявили более значительную структуру вокруг сайтов паузы рибосом [8], этот паттерн не был обнаружен в исследованиях in vivo [17 ••, 19 ••]. Это могло отражать влияние клеточной среды на структуру РНК.
Последние достижения in vitro Методы картирования структуры РНК могут дополнять данные in vivo о структуре РНК. Систематическая «мутация-и-карта» может идентифицировать специфические взаимодействия спаривания оснований и может быть объединена с анализом мультиплексированного гидроксильного радикального расщепления третичной близости для моделирования трехмерных структур РНК с разрешением, близким к атомному [32].Мощная стратегия в будущем может состоять в том, чтобы номинировать структурные мотивы РНК по in vivo, структурным данным, с последующим детальным рассечением in vitro, и 3D-моделированием лучших кандидатов.
Прямое определение спиралей и сложных и динамических структур
Одномерные данные ферментативного и химического исследования структур РНК предоставили интересную информацию о коллективных особенностях структур РНК, но их прямое использование в моделировании структуры было довольно трудным.Во-первых, взаимодействия пар оснований, предсказанные с помощью данных о реактивности, далеки от точности, особенно для дальнодействующих взаимодействий и сложных псевдоузлов. Во-вторых, структуры большинства молекул РНК в живых клетках неоднородны, гибки и динамичны, и поэтому эти структуры лучше описывать как ансамбль. Однако современные методы химического зондирования in vivo структуры РНК позволяют определять среднюю структуру в динамической клеточной среде. Разложение ансамбля структур на их индивидуальные состояния из одномерных данных нецелесообразно.В нескольких недавних исследованиях эти проблемы начали решаться с использованием новых вычислительных и экспериментальных подходов.
Нуклеотиды, которые взаимодействуют друг с другом, демонстрируют коррелированную химическую реактивность, и эту корреляцию можно использовать для обнаружения взаимодействий и классификации различных структурных состояний. Уикс и его коллеги недавно реализовали этот принцип в двух методах: SHAPE-Mutational Profiling (SHAPE-MaP) и RNA INtecting Group-MaP (RING-MaP) [26 •, 27 •]. В обоих методах используются особые условия для облегчения считывания обратной транскрипции на модифицированных нуклеотидах для введения мутаций, обычно более одной для каждой матрицы РНК.Корреляционный и кластерный анализ мутаций в секвенированных считываниях создает ограничения для построения 2D и 3D моделей исследуемой РНК. Эти методы были применены к нескольким свернутым РНК in vitro и дали улучшенные структурные модели. Хотя эта стратегия была протестирована только на нескольких молекулах, они потенциально могут быть адаптированы ко всему транскриптому. Однако использование этих методов для прямой деконволюции больших и сложных РНК может быть проблематичным.
Прямая информация о спаривании оснований также может быть получена путем измерения физической близости между нуклеотидами.Толлервей и его коллеги обнаружили лигированные фрагменты РНК при перекрестном связывании и секвенировании дцРНК, которые связываются белками. Основываясь на этом открытии, они разработали метод под названием Crosslinking, Ligation and Sequencing Hybrids (CLASH) для анализа структур РНК и взаимодействий РНК-РНК [28,29]. Sugimoto и его коллеги применили этот принцип к iCLIP, разработав гибридный iCLIP (hiCLIP) метод, чтобы идентифицировать структуры, связанные с dsRBP STAU1 [30]. В этом подходе фрагменты дцРНК, связанные с STAU1, захватываются с помощью лигирования близости, и каждое секвенированное считывание представляет собой измерение структуры одной молекулы.Метод hiCLIP выявил множество связанных с STAU1 дальнодействующих взаимодействий, разделенных тысячами или более оснований на линейном транскрипте, которые особенно трудно обнаружить с помощью обычных методов. Поскольку каждое считывание секвенирования происходит из одной структурной конформации, лигирование близости также позволяет обнаруживать индивидуальные структурные состояния в небольшом подмножестве структуромы, а не в среднем по структурному ансамблю [30]. Одним из основных ограничений методов CLASH / hiCLIP является низкий процент чтений, которые предоставляют прямую контактную информацию (менее 2%).Поскольку большинство эукариот кодируют большое количество RBP (на самом деле большее количество, чем ДНК-связывающих факторов транскрипции), применение hiCLIP к ним один за другим является трудоемким. Более того, многие структуры РНК вообще не связаны с RBP и поэтому их невозможно идентифицировать с помощью hiCLIP.
Подобно использованию лигирования близости для захвата структур РНК, Ramani и Shendure сообщили о более общем методе, RNA Proximity Ligation (RPL) [31]. В этом подходе использовалось расщепление РНК in situ РНКазой и лигирование по близости для соединения дуплексов в физической близости.Результаты, подтверждающие концепцию, были представлены для нескольких очень распространенных некодирующих РНК, таких как рибосомные РНК и мяРНК. Однако, поскольку сшивание не используется и клетки либо перевариваются эндогенными нуклеазами, либо лизируются и расщепляются экзогенными нуклеазами, случайные лигирования приводят к низкой точности и высокому шуму. Поскольку отбор не производится, процент химерных фрагментов очень низок (менее 0,5%), что затрудняет масштабирование метода для применения в масштабе всего транскриптома.Хотя методы на основе корреляции и близости-лигирования, описанные здесь, еще не достигли анализа структур целых РНК, они представляют собой интересные попытки с разных точек зрения, кроме методов химического зондирования.
Резюме и перспективы на будущее
Полное понимание структуры РНК требует полного описания структур и их динамики. Недавние разработки методов химического зондирования на основе секвенирования позволили получить глобальный обзор профилей локальной структуры РНК.Ожидается, что дальнейшее развитие методов обнаружения пар оснований предоставит столь необходимую информацию о более сложных и динамичных архитектурах РНК. Как специфические нуклеиновые кислоты, так и сшивающие белок-РНК могут быть дополнительно исследованы для нацеливания на различные виды вторичных или третичных структур. Комбинация этих двух различных классов экспериментальных методов необходима для получения полной картины структуры РНК, от нуклеотидов до высокоуровневых архитектур. Хотя 3D-моделирование структур РНК в настоящее время ограничено короткими РНК [32], интеграция методов химического зондирования и перекрестного связывания должна помочь деконволюции структурных ансамблей больших РНК и облегчить изоляцию структурных доменов, чтобы к ним можно было применить 3D-моделирование.Филогенетический анализ структур РНК часто подтверждает их функциональную важность, но страдает от редкости ковариации в большинстве РНК. Интеграция филогенетического анализа с экспериментальными методами объединит сильные стороны обоих подходов и поможет понять функции вновь идентифицированных структур РНК.
Долгосрочная цель изучения структур РНК состоит в том, чтобы не только создать каталог структур, но и сделать вывод об их функциях на основе классификации и корреляции с известными мотивами последовательностей и связыванием белков, онтологией генов, а затем определить их приоритетность для функциональные и механистические исследования.Во многих биологических процессах структуры РНК можно рассматривать как центр, который объединяет входные данные от клеточных компонентов и внеклеточной среды. Нарушения базовой последовательности или этих входных источников регулируют важные функции, основанные на РНК, или приводят к заболеваниям. Например, стрессовые условия способствуют избирательной трансляции мРНК, процессу, который зависит от структур мРНК [33]. Более того, мутации во многих РНК-геликазах приводят к раку [34]. Применение методов определения структуры РНК к этим разнообразным проблемам прольет свет как на основные механизмы экспрессии генов, так и на потенциальные терапевтические возможности для лечения заболеваний.
Благодарности
Мы благодарим сотрудников лаборатории Чанг за обсуждение и приносим свои извинения коллегам, чьи работы не обсуждаются из-за нехватки места. Мы благодарим NIH (R01-HG004361 и P50-HG007735) и Калифорнийский институт регенеративной медицины (HYC) за поддержку. З.Л. является стипендиатом семьи Лейтон в рамках стипендии Фонда Дэймона Руньона-Сона по педиатрическому онкологическому лечению (DRSG-14-15).
Сноски
Заявление о конфликте интересов : HYC является изобретателем по патенту Стэнфордского университета на технологию in vivo SHAPE.
Ссылки и рекомендуемая литература
Документы, представляющие особый интерес, опубликованные в течение периода обзора, были выделены следующим образом:
• особый интерес
•• представляющий особый интерес
1. Mortimer SA, Kidwell MA, Doudna JA. Понимание структуры и функции РНК из полногеномных исследований. Nat Rev Genet. 2014; 15: 469–479. [PubMed] [Google Scholar] 3. Ван И, Кертес М., Спитале Р.С., Сигал Э., Чанг Х.Й. Понимание транскриптома через структуру РНК.Nat Rev Genet. 2011; 12: 641–655. [Бесплатная статья PMC] [PubMed] [Google Scholar] 4. Wiebe NJ, Meyer IM. TRANSAT — метод обнаружения консервативных спиралей функциональных структур РНК, включая временные, псевдузловые и альтернативные структуры. PLoS Comput Biol. 2010; 6: e1000823. [Бесплатная статья PMC] [PubMed] [Google Scholar] 5. Jaeger JA, SantaLucia J, Jr, Tinoco I., Jr Определение структуры РНК и термодинамики. Анну Рев Биохим. 1993. 62: 255–287. [PubMed] [Google Scholar] 6. Ehresmann C, Baudin F, Mougel M, Romby P, Ebel JP, Ehresmann B.Исследование структуры РНК в растворе. Nucleic Acids Res. 1987; 15: 9109–9128. [Бесплатная статья PMC] [PubMed] [Google Scholar] 7. Меринос Э.Дж., Уилкинсон К.А., Кофлан Д.Л., Недели КМ. Анализ структуры РНК при разрешении одного нуклеотида путем селективного 2′-гидроксилацилирования и удлинения праймера (SHAPE) J Am Chem Soc. 2005; 127: 4223–4231. [PubMed] [Google Scholar] 8. Kertesz M, Wan Y, Mazor E, Rinn JL, Nutter RC, Chang HY, Segal E. Измерение вторичной структуры РНК в дрожжах по всему геному. Природа. 2010; 467: 103–107.[Бесплатная статья PMC] [PubMed] [Google Scholar] 9. Ван И, Цюй К., Чжан К.К., Флинн Р.А., Поместье О, Оуян З., Чжан Дж., Спитале Р.С., Снайдер М.П., Сигал Э. и др. Пейзаж и вариации вторичной структуры РНК в транскриптоме человека. Природа. 2014; 505: 706–709. [Бесплатная статья PMC] [PubMed] [Google Scholar] 10. Андервуд Дж. Г., Узилов А. В., Кацман С., Онодера С. С., Майнцер Дж. Э., Мэтьюз Д. Х., Лоу TM, Салама С. Р., Хаусслер Д. FragSeq: исследование структуры РНК по всему транскриптому с использованием высокопроизводительного секвенирования. Нат методы.2010; 7: 995–1001. [Бесплатная статья PMC] [PubMed] [Google Scholar] 11. Чжэн К., Рывкин П., Ли Ф, Драгомир И., Валладарес О, Ян Дж., Цао К., Ван Л.С., Грегори Б.Д. Полногеномное секвенирование двухцепочечной РНК показывает функциональное значение РНК с парными основаниями у Arabidopsis. PLoS Genet. 2010; 6: e1001141. [Бесплатная статья PMC] [PubMed] [Google Scholar] 12. Ситин MG, Мэтьюз DH. Прогнозирование структуры РНК: обзор методов. Методы Мол биол. 2012; 905: 99–122. [PubMed] [Google Scholar] 13. Kwok CK, Tang Y, Assmann SM, Bevilacqua PC.Структурома РНК: исследование структуры транскриптома с секвенированием следующего поколения. Trends Biochem Sci. 2015; 40: 221–232. [PubMed] [Google Scholar] 14. Spitale RC, Флинн Р.А., Торре Е.А., Kool ET, Chang HY. Структурный анализ РНК путем развития химии SHAPE. Wiley Interdiscip Rev RNA. 2014; 5: 867–881. [Бесплатная статья PMC] [PubMed] [Google Scholar] 16 ••. Дин И, Тан И, Квок С.К., Чжан И, Бевилаква П.К., Ассманн С.М. Полногеномное профилирование вторичной структуры РНК in vivo выявляет новые регуляторные особенности.Природа. 2014; 505: 696–700. Эта статья вместе с работами [17 ••, 20 ••] объединили модификацию РНК с помощью DMS и высокопроизводительное секвенирование, чтобы впервые проанализировать структуру РНК in vivo . Эти анализы выявили много интересных структурных особенностей, связанных с отдельными функциональными мотивами и принципами, которые управляют формированием структуры в клетках. [PubMed] [Google Scholar] 17 ••. Рускин С, Зубрадт М, Вашитль С, Келлис М, Вайсман Дж. Полногеномное зондирование структуры РНК выявляет активное развертывание структур мРНК in vivo.Природа. 2014; 505: 701–705. См. Аннотацию к исх. [16 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 19 ••. Spitale RC, Flynn RA, Zhang QC, Crisalli P, Lee B, Jung JW, Kuchelmeister HY, Batista PJ, Torre EA, Kool ET, et al. Структурные импринты in vivo расшифровывают механизмы регуляции РНК. Природа. 2015; 519: 486–490. В этой статье сообщается о недавно разработанном реагенте SHAPE, который обеспечивает очистку и селективное секвенирование модифицированных фрагментов РНК, повышая специфичность и чувствительность in vivo SHAPE.Резкое повышение точности зондирования позволило изучить генетически запрограммированные структурные особенности РНК и структурные отпечатки связывания RBP и модификаций РНК. [Бесплатная статья PMC] [PubMed] [Google Scholar] 20 ••. Talkish J, May G, Lin Y, Woolford JL, Jr, McManus CJ. Mod-seq: высокопроизводительное секвенирование для химического исследования структуры РНК. РНК. 2014; 20: 713–720. См. Аннотацию к исх. [16 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 21. Кладванг В., ВанЛанг СС, Кордеро П., Дас Р.Понимание ошибок моделирования структуры РНК с помощью SHAPE. Биохимия. 2011; 50: 8049–8056. [Бесплатная статья PMC] [PubMed] [Google Scholar] 22. Шабалина С.А., Огурцов А.Ю., Спиридонов Н.А. Периодический паттерн вторичной структуры мРНК, созданный генетическим кодом. Nucleic Acids Res. 2006; 34: 2428–2437. [Бесплатная статья PMC] [PubMed] [Google Scholar] 23. Смола MJ, Calabrese JM, Weeks KM. Обнаружение РНК-белковых взаимодействий в живых клетках с помощью SHAPE. Биохимия. 2015; 54: 6867–6875. [Бесплатная статья PMC] [PubMed] [Google Scholar] 24.Liu N, Dai Q, Zheng G, He C, Parisien M, Pan T. N (6) -метиладенозин-зависимые структурные переключатели РНК регулируют РНК-белковые взаимодействия. Природа. 2015; 518: 560–564. [Бесплатная статья PMC] [PubMed] [Google Scholar] 25. Руст К., Линч С.Р., Батиста П.Дж., Ку К., Чанг Х.Й., Кул Э. Структура и термодинамика N6-метиладенозина в РНК: подпружиненная модификация основания. J Am Chem Soc. 2015; 137: 2107–2115. [Бесплатная статья PMC] [PubMed] [Google Scholar] 26 •. Хоман П.Дж., Фаворов О.В., Лаванда К.А., Курсун О, Ге X, Пусан С, Дохолян Н.В., Недели КМ.Одномолекулярное коррелированное химическое зондирование РНК. Proc Natl Acad Sci U S. A. 2014; 111: 13858–13863. Эта статья вместе с Ref. [27 •] сообщает о новых условиях химического зондирования, которые вводят мутации в сайты модификаций. Кроме того, они используют корреляционный анализ для выявления контактов в пространстве и кластерный анализ для выявления различных состояний в структурном ансамбле. [Бесплатная статья PMC] [PubMed] [Google Scholar] 27 •. Зигфрид Н.А., Пусан С., Райс ГМ, Нельсон Дж. А., Недели КМ. Обнаружение мотивов РНК с помощью методов SHAPE и мутационного профилирования (SHAPE-MaP) Nat.2014; 11: 959–965. См. Аннотацию к исх. [26 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 28. Helwak A, Kudla G, Dudnakova T, Tollervey D. Картирование взаимодействия miRNA человека с помощью CLASH обнаруживает частое неканоническое связывание. Клетка. 2013; 153: 654–665. [Бесплатная статья PMC] [PubMed] [Google Scholar] 29. Kudla G, Granneman S, Hahn D, Beggs JD, Tollervey D. Перекрестное связывание, лигирование и секвенирование гибридов выявляют взаимодействия РНК-РНК в дрожжах. Proc Natl Acad Sci U S. A. 2011; 108: 10010–10015. [Бесплатная статья PMC] [PubMed] [Google Scholar] 30 ••.Sugimoto Y, Vigilante A, Darbo E, Zirra A, Militti C, D’Ambrogio A, Luscombe NM, Ule J. hiCLIP раскрывает in vivo атлас вторичных структур мРНК, распознаваемых Staufen 1. Nature. 2015; 519: 491–494. В этой статье используется перекрестное связывание и лигирование по близости для определения структур РНК, связанных с dsRBP STAU1, и выявлены дальнодействующие и альтернативные структуры РНК, которые невозможно найти с помощью методов химического зондирования. [Бесплатная статья PMC] [PubMed] [Google Scholar] 31. Рамани В., Цю Р., Шендурэ Дж.Высокопроизводительное определение структуры РНК путем лигирования близости. Nat Biotechnol. 2015; 33: 980–984. [Бесплатная статья PMC] [PubMed] [Google Scholar] 32. Cheng CY, Chou FC, Kladwang W, Tian S, Cordero P, Das R. Согласованные глобальные структуры сложных состояний РНК посредством многомерного химического картирования. Элиф. 2015; 4: e07600. [Бесплатная статья PMC] [PubMed] [Google Scholar] 33. Thakor N, Holcik M. IRES-опосредованная трансляция клеточной информационной РНК работает независимо от eIF2alpha во время стресса. Nucleic Acids Res.2012; 40: 541–552. [Бесплатная статья PMC] [PubMed] [Google Scholar]Расшифровка структур РНК
Curr Opin Struct Biol. Авторская рукопись; доступно в PMC 2017 26 февраля.
Опубликован в окончательной отредактированной форме как:
PMCID: PMC4785074
NIHMSID: NIHMS764989
Центр личных динамических регуляторов, Медицинская школа Стэнфордского университета, Стэнфорд, Калифорния 94305, США
См. Другие статьи в PMC, в которых цитируется опубликованная статья.Abstract
Структуры молекул РНК важны для их архитектурных, регуляторных и каталитических функций.Недавние достижения в области высокопроизводительного секвенирования позволили разработать методы исследования структур РНК в масштабе всего транскриптома — так называемых структур РНК. Здесь мы рассматриваем современные технологии исследования структур РНК и выделяем выводы, полученные в результате этих исследований. Мы также указываем на пределы существующих методов и обсуждаем возможные направления для будущих улучшений.
Введение
Помимо передачи генетической информации, такой как ДНК, молекулы РНК сложены в изысканные структуры.Гибкие и динамические структуры молекул РНК лежат в основе их разнообразных функций. Подобно изучению белков, структурный анализ молекул РНК позволил разобраться в механизмах регуляции генов на самых разных уровнях. Структурные элементы в мРНК контролируют их субклеточную локализацию, а внутренний сайт входа в рибосомы (IRES) формирует трехмерные структуры, чтобы управлять кэп-независимой трансляцией (обзор [1]). Доступность мотивов в мРНК модулирует взаимодействие с РНК-связывающими белками (RBP) и микроРНК.Структура РНК также важна для некодирующих РНК. Структурные мотивы в длинных некодирующих РНК обеспечивают их ядерную локализацию, стабильность и взаимодействие с аппаратом модификации хроматина (rev. [2,3]). Уникальные структуры рибопереключателей позволяют им связывать определенные метаболиты и, в свою очередь, преобразовывать связывание лиганда в регуляторную активность генов. Третичные структуры, построенные на основных структурных элементах, необходимы рибозимам для катализа широкого спектра биохимических реакций, включая сплайсинг, лигирование и трансляцию.
Традиционные методы определения структуры, такие как рентгеновская кристаллография, ядерный магнитный резонанс (ЯМР) и криоэлектронная микроскопия (крио-ЭМ), создают трехмерные структуры с высоким разрешением, но для них требуются очищенные, структурно стабильные и почти статичные молекулы РНК. Поэтому их нелегко применить для изучения подавляющего большинства РНК, которые часто находятся в нескольких конформациях, динамических, сложных и постоянно регулируемых клеточной средой [4,5].
Другой класс методов, химическое и ферментативное зондирование, решает проблему структуры РНК под другим углом.Нуклеотиды в структурах РНК имеют различную доступность в зависимости от их спаривания оснований и других взаимодействий. В химическом зондировании используются различные соединения, которые реагируют с нуклеотидами в соответствии с их окружением и генерируют объемные аддукты или точки разрыва, которые можно измерить с помощью секвенирования по Сэнгеру или высокопроизводительного секвенирования как остановки или окончания обратной транскрипции [6]. Обычно используемые химические вещества включают селективное 2′-гидроксилацилирование, анализируемое химическими веществами для удлинения праймера (SHAPE), такими как 1-метил-7-нитроизатовый ангидрид (1M7), N -метилизатовый ангидрид (NMIA), который ацилирует 2′-OH в одноцепочечные (ss) области () [7], гидроксильные радикалы, которые разрушают незащищенные фосфатные скелеты, диметилсульфат (DMS), который метилирует ss-аденозины и цитозины, 1-циклогексил- (2-морфолиноэтил) карбодиимид мет-п-толуолсульфонат ( CMCT), который модифицирует ss-уридины и гуанины, кетоксал, который модифицирует ss-гуанины, и поточное зондирование, которое использует преимущества спонтанной деградации в ss-областях.Сходным образом, ферментативное зондирование использует двухцепочечные (ds) специфические РНКазы для разрезания РНК, оставляя ds / ss-специфические точки разрыва, которые указывают на статус спаривания оснований (8-11). Затем данные химического и ферментативного зондирования включаются в качестве ограничений в различные алгоритмы прогнозирования структуры РНК для повышения точности прогноза [12]. Такие экспериментальные данные существенно дополняют структурные предсказания, основанные на минимизации свободной энергии или эволюционной ко-вариации.
Таблица 1
Методы глобального определения структур РНК.Методы перечислены не по лабораториям, которые их разработали, а по стратегии и используемым химическим веществам. Обычная ФОРМА очень неэффективна для анализа in vivo , но является одним из самых ранних методов исследования структуры. Ферментативное зондирование не является полезным для анализа in vivo , но обеспечивает первый глобальный анализ структурома РНК. RING-MaP и SHAPE-MaP были разработаны для анализа одиночной РНК, но эта стратегия полезна для анализа структур in vivo . HiCLIP и RPL еще не достигли глобального анализа структуром РНК, но представляют интересные направления будущего
| Методы | Зонд | Данные | Предполагаемый | Характеристики и ограничения | Ссылки |
|---|---|---|---|---|---|
| Химическое зондирование | 1M7, DMS, CMCT, гидроксил, и т. д. | 1D | 2D | Все нуклеотиды, в основном in vitro , нет информации о прямом спаривании оснований | [6,7] |
| Ферментативная | РНКаза I, V1, P1, S1 | 1D | 2D | Все нуклеотиды, только in vitro , информация о прямом спаривании оснований отсутствует | [8–11] |
| SHAPE-MaP, RING-MaP | 1M7 и DMS | 1D | 2D / 3D | Дополнительная корреляция для баз, но только на короткие дистанции.Адаптируется к in vivo и глобально | [26 •, 27 •] |
| Вторичная структура in vivo | |||||
| DMS-seq | DMS | 1D | 2D | In vivo опрашивает только A / C, нет информации о прямом сопряжении оснований | [16 ••, 17 ••, 20 ••] |
| icSHAPE | NAI, NAI-N 3 | 1D | 2D | In vivo , все нуклеотиды, минимальный фон, нет информации о прямом спаривании оснований | [18,19 ••] |
| Структура вторичного и более высокого порядка in vivo | |||||
| CLASH, hiCLIP | УФ-сшивание | 2D | In vivo , прямое дуплексное определение, только dsRBP-связанные структуры | [28,29,30 ••] | |
| RPL | Без сшивки | 2D | Lysed клетки, прямое дуплексное определение, низкая эффективность и точность | [31] | |
Несколько отличных обзоров были посвящены химии и компьютерному анализу экспериментов по химическому и ферментативному зондированию [12–15].Здесь мы сосредоточимся на применении химического зондирования in vivo в масштабе всего транскриптома . Эти методы предоставили важную информацию о физиологическом состоянии молекул РНК. Тем не менее, эти методы генерируют только одномерную усредненную структурную информацию, что далеко не достаточно для полного описания структуромы РНК. Кроме того, мы также обсуждаем недавние усилия по разработке методов, которые исследуют дальнодействующие, альтернативные и сложные структуры РНК.
Методы
in vivo РНК-структуромного зондированияНедавние достижения в области высокопроизводительного секвенирования вывели эксперименты по химическому и ферментативному зондированию в глобальный масштаб.Хотя ферментные и большинство химических зондов можно применять только к образцам РНК in vitro, некоторые из реагентов, такие как DMS и недавно разработанный NAI (имидазолид 2-метилникотиновой кислоты) и NAI – N 3 , имеют соответствующие периоды полураспада ( ∼34 мин для NAI и NAI – N 3 ) и легко проникают в клетку, таким образом обеспечивая in vivo зондирования структур РНК () [16 ••, 17 ••, 18,19 ••]. Вместе технологии высокопроизводительного секвенирования и in vivo зондирующих химикатов позволили определить структуру РНК in vivo.
Первые сообщения о in vivo структурном зондировании использовали DMS, реагент алкилирования, для модификации аденинов и цитозинов, которые обнаруживаются как остановки обратной транскрипции () [16 ••, 17 ••, 20 ••]. Хотя эти исследования отличаются некоторыми экспериментальными деталями, они схожи по концепции и основным этапам. Алкилирование ss-оснований блокирует обратную транскрипцию и, таким образом, сообщает о статусе спаривания оснований в последовательности РНК. Одним из недостатков является то, что методы на основе DMS опрашивают только две из четырех баз.Поскольку модификация оснований влияет на их способность к спариванию оснований, модификации молекул РНК титровали для достижения кинетики «однократного попадания», при которой изменяется примерно один из 200 нуклеотидов. Кинетика однократного попадания приводит к другому недостатку: большинство фрагментов РНК немодифицированы, и, таким образом, секвенирование этих РНК дает высокий фон.
Обычные реагенты SHAPE изменяют все четыре нуклеотида единообразно, но применимы только к образцам in vitro . Чтобы преодолеть это ограничение, Спитале и его коллеги синтезировали новые типы реагентов SHAPE, которые легко проникают в клетку и эффективно реагируют с одноцепочечными нуклеотидами [18].Было показано, что эти химические вещества действуют в клетках бактерий, дрожжей, мух, мышей и человека. Чтобы еще больше снизить высокий фон из-за условий модификации с однократным попаданием, Спитале и его коллеги представили активную азидную метку для ацилирующего агента NAI, которая позволяет легко очищать модифицированные фрагменты РНК с помощью системы биотин-стептавидин (icSHAPE: in vivo ). щелкните ФОРМА) [19 ••]. Этот дополнительный этап отбора значительно снижает фон и обеспечивает более чувствительный и специфический анализ модифицированных фрагментов РНК.
Поскольку каждая молекула РНК и конформация модифицируются с очень низкой частотой в экспериментах по химическому зондированию, для создания профиля структуры требуется большое количество считываний секвенирования, даже при выборе биотина. Таким образом, полученная структурная информация представляет собой среднее значение всех конформаций. Несмотря на свои ограничения, DMS и NAI – N 3 на сегодняшний день являются лучшими реагентами для химического исследования структуры РНК in vivo .
Новое понимание структуры РНК
Данные химического зондирования структур РНК обычно анализируются и используются двумя способами: зондирование и прогнозирование (PnP) и зонд и среднее значение (PnA).Для подхода PnP профили реактивности включаются в качестве ограничений в методы прогнозирования вторичной структуры для создания структурных моделей, которые наилучшим образом объясняют профиль реактивности (см. Обзор [12]). Хотя этот подход увеличивает точность предсказания структуры, надежные модели могут быть получены только для очень коротких молекул РНК или областей со стабильными единственными структурными конформациями [21]. Следовательно, применение этого подхода ко всей структуроме РНК нецелесообразно. С другой стороны, подход PnA обычно выравнивает большое количество заранее определенных мотивов последовательности РНК для получения профиля реактивности метагена, который является средним значением для каждой уже усредненной структуры РНК.Используя подход PnA, недавние исследования выявили интересные особенности РНК-структурома in vivo и предоставили много нового понимания регуляторных функций РНК-структур (). Несколько исследований также сравнили структур in vivo с структур in vitro и обнаружили особенности, которые программируются только последовательностью, и те, на которые влияет клеточная среда.
Структром РНК: новые идеи, но неполная картина. (a – i) Новые открытия о структуроме РНК из недавних исследований глобального структурного зондирования.Большинство этих структурных особенностей очевидны на уровне метагена, но не на уровне отдельного гена. (a) Пример набора треков реактивности химического зонда для типичного гена мРНК, включая зондирование in vivo, и in vitro, и разницу между ними (трек VTD). В нескольких исследованиях наблюдали меньшую структуру in vivo , чем in vitro и активное разворачивание структур in vivo [17 ••, 19 ••]. (b) Структуры в CDS, но не UTRs, демонстрируют очевидную 3-ную периодичность как in vitro , так и in vivo [8,17 ••, 19 ••, 22].Область непосредственно перед стартовым кодоном и рядом со стоп-кодоном менее структурирована, чем в среднем [16 ••, 17 ••, 19 ••]. Однако относительное содержание структуры между UTR и CDS различается у разных организмов, больше в CDS для дрожжей и растений, но больше в UTR для животных [8]. (c) Сайты паузы рибосомы, а не отрицательные контроли, демонстрируют очевидную 3-нуклеотидную периодичность перед положением, в котором расположена рибосома (обозначено E (выход), P (пептидил-тРНК) и A (аминоацил-тРНК)) [19 ••]. (d) m 6 Модификация делает окружающую последовательность менее структурированной [19 ••], чем отрицательные контроли ( Mettl3 — / — теряет больше всего m 6 A). (e) Связывание RBP изменяет химическое окружение целевой РНК, таким образом оставляя след [19]. (f) Анализ онтологии генов A. thaliana показывает, что PPV (положительная прогностическая ценность), мера того, насколько близка предсказанная структура in silico к химически исследованной структуре, является высокой для РНК, кодирующих домашние белки, но низкий для тех, кто кодирует белки реакции на стресс / стимул [16]. (g) Метагенный анализ альтернативного сплайсинга показывает более высокую структуру (меньшую реактивность) перед 5′-сайтом сплайсинга (5’SS) перед пропущенным экзоном [16]. (h) Анализ hiCLIP STAU1 в клетках человека обнаруживает большое количество вторичных структур, некоторые из которых охватывают большие расстояния [30]. (i) Метагенный анализ альтернативных сайтов полиаденилирования A. thaliana выявил картину низкой реакционной способности, за которой следует высокая реактивность [16 ••]. (j – l) Важные аспекты структуромы, которые все еще плохо изучены.Диаграммы построены в формате линейной структуры. (j) Одна и та же последовательность может принимать различные структурные состояния in vivo , которые конкурируют друг с другом или с RBP и miRNA. (k) Сложные структуры и структуры высокого порядка, т. Е. Взаимосвязанный псевдоузел. (l) Взаимодействия при спаривании оснований с большим радиусом действия, которые могут охватывать множество тысяч оснований.
Задолго до того, как стал возможен анализ структур РНК, Шабалина et al. выполнил in silico фолдинг мРНК человека и мыши и обнаружил удивительную тринуклеотидную периодичность вероятности спаривания оснований в кодирующих областях, которая отсутствует в UTRs [22].Это предсказание было сначала подтверждено in vitro, для мРНК дрожжей [8], а позже было показано, что оно является повсеместным явлением in vivo у нескольких организмов () [16 ••, 17 ••, 19 ••]. Эти исследования также сообщили о тенденции области стартового кодона, канонической или неканонической, иметь меньшую структуру, свойство, которое, вероятно, облегчает инициацию трансляции.
Применяя подход PnA к проросткам A. thaliana , Динг и его коллеги обнаружили структурные особенности мРНК вокруг альтернативных сайтов сплайсинга и полиаденилирования (16).Данные предполагают, что, помимо мотивов последовательности, структуры вокруг сайтов процессинга мРНК играют важную роль в управлении процессингом. Анализ онтологии генов показал, что мРНК A. thaliana , которые кодируют белки реакции на стресс / стимул, как правило, менее структурированы in vivo , чем те, которые кодируют домашние белки. Однако эти данные следует интерпретировать с осторожностью, поскольку известно, что содержание GC и многие другие факторы могут влиять на относительное структурное содержание молекул РНК.
Основываясь на идее, что клеточные компоненты влияют на структуры РНК и могут приводить к локальным структурным перестройкам, Spitale et al. проанализировал разницу между in vitro и in vivo данными icSHAPE и наблюдал специфические VTD ( in vivo in vitro различие) сигнатуры для нескольких мотивов последовательности [19]. Примечательно, что сигнал VTD позволил детектировать фокальные перестройки структур, индуцированные связыванием RBP, так что присутствие или отсутствие VTD в каждом случае мотива связывания RBP можно использовать для прогнозирования связывания RBP in vivo ().Эта концепция структурного отпечатка РНК аналогична «отпечатку» фактора транскрипции на ДНК. Подобно подходу VTD, Smola et al. использовал 1M7 для обнаружения сайтов связывания с белками на нескольких многочисленных РНК, сравнивая статус модификации между в целлюлозе и ex vivo в условиях [23]. Вместе эти исследования подчеркивают мощь методов, основанных на SHAPE, во взаимодействиях РНК-белок. Spitale et al. далее обнаружил, что модификация РНК N 6 -метиладенозин (m 6 A) является структурным переключателем in vivo , который приводит к распариванию дуплексов РНК () [19 ••]. in vitro анализов и подробная биофизическая характеристика [24,25].Удивительно, хотя in silico и in vitro исследований структуры РНК выявили более значительную структуру вокруг сайтов паузы рибосом [8], этот паттерн не был обнаружен в исследованиях in vivo [17 ••, 19 ••]. Это могло отражать влияние клеточной среды на структуру РНК.
Последние достижения in vitro Методы картирования структуры РНК могут дополнять данные in vivo о структуре РНК. Систематическая «мутация-и-карта» может идентифицировать специфические взаимодействия спаривания оснований и может быть объединена с анализом мультиплексированного гидроксильного радикального расщепления третичной близости для моделирования трехмерных структур РНК с разрешением, близким к атомному [32].Мощная стратегия в будущем может состоять в том, чтобы номинировать структурные мотивы РНК по in vivo, структурным данным, с последующим детальным рассечением in vitro, и 3D-моделированием лучших кандидатов.
Прямое определение спиралей и сложных и динамических структур
Одномерные данные ферментативного и химического исследования структур РНК предоставили интересную информацию о коллективных особенностях структур РНК, но их прямое использование в моделировании структуры было довольно трудным.Во-первых, взаимодействия пар оснований, предсказанные с помощью данных о реактивности, далеки от точности, особенно для дальнодействующих взаимодействий и сложных псевдоузлов. Во-вторых, структуры большинства молекул РНК в живых клетках неоднородны, гибки и динамичны, и поэтому эти структуры лучше описывать как ансамбль. Однако современные методы химического зондирования in vivo структуры РНК позволяют определять среднюю структуру в динамической клеточной среде. Разложение ансамбля структур на их индивидуальные состояния из одномерных данных нецелесообразно.В нескольких недавних исследованиях эти проблемы начали решаться с использованием новых вычислительных и экспериментальных подходов.
Нуклеотиды, которые взаимодействуют друг с другом, демонстрируют коррелированную химическую реактивность, и эту корреляцию можно использовать для обнаружения взаимодействий и классификации различных структурных состояний. Уикс и его коллеги недавно реализовали этот принцип в двух методах: SHAPE-Mutational Profiling (SHAPE-MaP) и RNA INtecting Group-MaP (RING-MaP) [26 •, 27 •]. В обоих методах используются особые условия для облегчения считывания обратной транскрипции на модифицированных нуклеотидах для введения мутаций, обычно более одной для каждой матрицы РНК.Корреляционный и кластерный анализ мутаций в секвенированных считываниях создает ограничения для построения 2D и 3D моделей исследуемой РНК. Эти методы были применены к нескольким свернутым РНК in vitro и дали улучшенные структурные модели. Хотя эта стратегия была протестирована только на нескольких молекулах, они потенциально могут быть адаптированы ко всему транскриптому. Однако использование этих методов для прямой деконволюции больших и сложных РНК может быть проблематичным.
Прямая информация о спаривании оснований также может быть получена путем измерения физической близости между нуклеотидами.Толлервей и его коллеги обнаружили лигированные фрагменты РНК при перекрестном связывании и секвенировании дцРНК, которые связываются белками. Основываясь на этом открытии, они разработали метод под названием Crosslinking, Ligation and Sequencing Hybrids (CLASH) для анализа структур РНК и взаимодействий РНК-РНК [28,29]. Sugimoto и его коллеги применили этот принцип к iCLIP, разработав гибридный iCLIP (hiCLIP) метод, чтобы идентифицировать структуры, связанные с dsRBP STAU1 [30]. В этом подходе фрагменты дцРНК, связанные с STAU1, захватываются с помощью лигирования близости, и каждое секвенированное считывание представляет собой измерение структуры одной молекулы.Метод hiCLIP выявил множество связанных с STAU1 дальнодействующих взаимодействий, разделенных тысячами или более оснований на линейном транскрипте, которые особенно трудно обнаружить с помощью обычных методов. Поскольку каждое считывание секвенирования происходит из одной структурной конформации, лигирование близости также позволяет обнаруживать индивидуальные структурные состояния в небольшом подмножестве структуромы, а не в среднем по структурному ансамблю [30]. Одним из основных ограничений методов CLASH / hiCLIP является низкий процент чтений, которые предоставляют прямую контактную информацию (менее 2%).Поскольку большинство эукариот кодируют большое количество RBP (на самом деле большее количество, чем ДНК-связывающих факторов транскрипции), применение hiCLIP к ним один за другим является трудоемким. Более того, многие структуры РНК вообще не связаны с RBP и поэтому их невозможно идентифицировать с помощью hiCLIP.
Подобно использованию лигирования близости для захвата структур РНК, Ramani и Shendure сообщили о более общем методе, RNA Proximity Ligation (RPL) [31]. В этом подходе использовалось расщепление РНК in situ РНКазой и лигирование по близости для соединения дуплексов в физической близости.Результаты, подтверждающие концепцию, были представлены для нескольких очень распространенных некодирующих РНК, таких как рибосомные РНК и мяРНК. Однако, поскольку сшивание не используется и клетки либо перевариваются эндогенными нуклеазами, либо лизируются и расщепляются экзогенными нуклеазами, случайные лигирования приводят к низкой точности и высокому шуму. Поскольку отбор не производится, процент химерных фрагментов очень низок (менее 0,5%), что затрудняет масштабирование метода для применения в масштабе всего транскриптома.Хотя методы на основе корреляции и близости-лигирования, описанные здесь, еще не достигли анализа структур целых РНК, они представляют собой интересные попытки с разных точек зрения, кроме методов химического зондирования.
Резюме и перспективы на будущее
Полное понимание структуры РНК требует полного описания структур и их динамики. Недавние разработки методов химического зондирования на основе секвенирования позволили получить глобальный обзор профилей локальной структуры РНК.Ожидается, что дальнейшее развитие методов обнаружения пар оснований предоставит столь необходимую информацию о более сложных и динамичных архитектурах РНК. Как специфические нуклеиновые кислоты, так и сшивающие белок-РНК могут быть дополнительно исследованы для нацеливания на различные виды вторичных или третичных структур. Комбинация этих двух различных классов экспериментальных методов необходима для получения полной картины структуры РНК, от нуклеотидов до высокоуровневых архитектур. Хотя 3D-моделирование структур РНК в настоящее время ограничено короткими РНК [32], интеграция методов химического зондирования и перекрестного связывания должна помочь деконволюции структурных ансамблей больших РНК и облегчить изоляцию структурных доменов, чтобы к ним можно было применить 3D-моделирование.Филогенетический анализ структур РНК часто подтверждает их функциональную важность, но страдает от редкости ковариации в большинстве РНК. Интеграция филогенетического анализа с экспериментальными методами объединит сильные стороны обоих подходов и поможет понять функции вновь идентифицированных структур РНК.
Долгосрочная цель изучения структур РНК состоит в том, чтобы не только создать каталог структур, но и сделать вывод об их функциях на основе классификации и корреляции с известными мотивами последовательностей и связыванием белков, онтологией генов, а затем определить их приоритетность для функциональные и механистические исследования.Во многих биологических процессах структуры РНК можно рассматривать как центр, который объединяет входные данные от клеточных компонентов и внеклеточной среды. Нарушения базовой последовательности или этих входных источников регулируют важные функции, основанные на РНК, или приводят к заболеваниям. Например, стрессовые условия способствуют избирательной трансляции мРНК, процессу, который зависит от структур мРНК [33]. Более того, мутации во многих РНК-геликазах приводят к раку [34]. Применение методов определения структуры РНК к этим разнообразным проблемам прольет свет как на основные механизмы экспрессии генов, так и на потенциальные терапевтические возможности для лечения заболеваний.
Благодарности
Мы благодарим сотрудников лаборатории Чанг за обсуждение и приносим свои извинения коллегам, чьи работы не обсуждаются из-за нехватки места. Мы благодарим NIH (R01-HG004361 и P50-HG007735) и Калифорнийский институт регенеративной медицины (HYC) за поддержку. З.Л. является стипендиатом семьи Лейтон в рамках стипендии Фонда Дэймона Руньона-Сона по педиатрическому онкологическому лечению (DRSG-14-15).
Сноски
Заявление о конфликте интересов : HYC является изобретателем по патенту Стэнфордского университета на технологию in vivo SHAPE.
Ссылки и рекомендуемая литература
Документы, представляющие особый интерес, опубликованные в течение периода обзора, были выделены следующим образом:
• особый интерес
•• представляющий особый интерес
1. Mortimer SA, Kidwell MA, Doudna JA. Понимание структуры и функции РНК из полногеномных исследований. Nat Rev Genet. 2014; 15: 469–479. [PubMed] [Google Scholar] 3. Ван И, Кертес М., Спитале Р.С., Сигал Э., Чанг Х.Й. Понимание транскриптома через структуру РНК.Nat Rev Genet. 2011; 12: 641–655. [Бесплатная статья PMC] [PubMed] [Google Scholar] 4. Wiebe NJ, Meyer IM. TRANSAT — метод обнаружения консервативных спиралей функциональных структур РНК, включая временные, псевдузловые и альтернативные структуры. PLoS Comput Biol. 2010; 6: e1000823. [Бесплатная статья PMC] [PubMed] [Google Scholar] 5. Jaeger JA, SantaLucia J, Jr, Tinoco I., Jr Определение структуры РНК и термодинамики. Анну Рев Биохим. 1993. 62: 255–287. [PubMed] [Google Scholar] 6. Ehresmann C, Baudin F, Mougel M, Romby P, Ebel JP, Ehresmann B.Исследование структуры РНК в растворе. Nucleic Acids Res. 1987; 15: 9109–9128. [Бесплатная статья PMC] [PubMed] [Google Scholar] 7. Меринос Э.Дж., Уилкинсон К.А., Кофлан Д.Л., Недели КМ. Анализ структуры РНК при разрешении одного нуклеотида путем селективного 2′-гидроксилацилирования и удлинения праймера (SHAPE) J Am Chem Soc. 2005; 127: 4223–4231. [PubMed] [Google Scholar] 8. Kertesz M, Wan Y, Mazor E, Rinn JL, Nutter RC, Chang HY, Segal E. Измерение вторичной структуры РНК в дрожжах по всему геному. Природа. 2010; 467: 103–107.[Бесплатная статья PMC] [PubMed] [Google Scholar] 9. Ван И, Цюй К., Чжан К.К., Флинн Р.А., Поместье О, Оуян З., Чжан Дж., Спитале Р.С., Снайдер М.П., Сигал Э. и др. Пейзаж и вариации вторичной структуры РНК в транскриптоме человека. Природа. 2014; 505: 706–709. [Бесплатная статья PMC] [PubMed] [Google Scholar] 10. Андервуд Дж. Г., Узилов А. В., Кацман С., Онодера С. С., Майнцер Дж. Э., Мэтьюз Д. Х., Лоу TM, Салама С. Р., Хаусслер Д. FragSeq: исследование структуры РНК по всему транскриптому с использованием высокопроизводительного секвенирования. Нат методы.2010; 7: 995–1001. [Бесплатная статья PMC] [PubMed] [Google Scholar] 11. Чжэн К., Рывкин П., Ли Ф, Драгомир И., Валладарес О, Ян Дж., Цао К., Ван Л.С., Грегори Б.Д. Полногеномное секвенирование двухцепочечной РНК показывает функциональное значение РНК с парными основаниями у Arabidopsis. PLoS Genet. 2010; 6: e1001141. [Бесплатная статья PMC] [PubMed] [Google Scholar] 12. Ситин MG, Мэтьюз DH. Прогнозирование структуры РНК: обзор методов. Методы Мол биол. 2012; 905: 99–122. [PubMed] [Google Scholar] 13. Kwok CK, Tang Y, Assmann SM, Bevilacqua PC.Структурома РНК: исследование структуры транскриптома с секвенированием следующего поколения. Trends Biochem Sci. 2015; 40: 221–232. [PubMed] [Google Scholar] 14. Spitale RC, Флинн Р.А., Торре Е.А., Kool ET, Chang HY. Структурный анализ РНК путем развития химии SHAPE. Wiley Interdiscip Rev RNA. 2014; 5: 867–881. [Бесплатная статья PMC] [PubMed] [Google Scholar] 16 ••. Дин И, Тан И, Квок С.К., Чжан И, Бевилаква П.К., Ассманн С.М. Полногеномное профилирование вторичной структуры РНК in vivo выявляет новые регуляторные особенности.Природа. 2014; 505: 696–700. Эта статья вместе с работами [17 ••, 20 ••] объединили модификацию РНК с помощью DMS и высокопроизводительное секвенирование, чтобы впервые проанализировать структуру РНК in vivo . Эти анализы выявили много интересных структурных особенностей, связанных с отдельными функциональными мотивами и принципами, которые управляют формированием структуры в клетках. [PubMed] [Google Scholar] 17 ••. Рускин С, Зубрадт М, Вашитль С, Келлис М, Вайсман Дж. Полногеномное зондирование структуры РНК выявляет активное развертывание структур мРНК in vivo.Природа. 2014; 505: 701–705. См. Аннотацию к исх. [16 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 19 ••. Spitale RC, Flynn RA, Zhang QC, Crisalli P, Lee B, Jung JW, Kuchelmeister HY, Batista PJ, Torre EA, Kool ET, et al. Структурные импринты in vivo расшифровывают механизмы регуляции РНК. Природа. 2015; 519: 486–490. В этой статье сообщается о недавно разработанном реагенте SHAPE, который обеспечивает очистку и селективное секвенирование модифицированных фрагментов РНК, повышая специфичность и чувствительность in vivo SHAPE.Резкое повышение точности зондирования позволило изучить генетически запрограммированные структурные особенности РНК и структурные отпечатки связывания RBP и модификаций РНК. [Бесплатная статья PMC] [PubMed] [Google Scholar] 20 ••. Talkish J, May G, Lin Y, Woolford JL, Jr, McManus CJ. Mod-seq: высокопроизводительное секвенирование для химического исследования структуры РНК. РНК. 2014; 20: 713–720. См. Аннотацию к исх. [16 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 21. Кладванг В., ВанЛанг СС, Кордеро П., Дас Р.Понимание ошибок моделирования структуры РНК с помощью SHAPE. Биохимия. 2011; 50: 8049–8056. [Бесплатная статья PMC] [PubMed] [Google Scholar] 22. Шабалина С.А., Огурцов А.Ю., Спиридонов Н.А. Периодический паттерн вторичной структуры мРНК, созданный генетическим кодом. Nucleic Acids Res. 2006; 34: 2428–2437. [Бесплатная статья PMC] [PubMed] [Google Scholar] 23. Смола MJ, Calabrese JM, Weeks KM. Обнаружение РНК-белковых взаимодействий в живых клетках с помощью SHAPE. Биохимия. 2015; 54: 6867–6875. [Бесплатная статья PMC] [PubMed] [Google Scholar] 24.Liu N, Dai Q, Zheng G, He C, Parisien M, Pan T. N (6) -метиладенозин-зависимые структурные переключатели РНК регулируют РНК-белковые взаимодействия. Природа. 2015; 518: 560–564. [Бесплатная статья PMC] [PubMed] [Google Scholar] 25. Руст К., Линч С.Р., Батиста П.Дж., Ку К., Чанг Х.Й., Кул Э. Структура и термодинамика N6-метиладенозина в РНК: подпружиненная модификация основания. J Am Chem Soc. 2015; 137: 2107–2115. [Бесплатная статья PMC] [PubMed] [Google Scholar] 26 •. Хоман П.Дж., Фаворов О.В., Лаванда К.А., Курсун О, Ге X, Пусан С, Дохолян Н.В., Недели КМ.Одномолекулярное коррелированное химическое зондирование РНК. Proc Natl Acad Sci U S. A. 2014; 111: 13858–13863. Эта статья вместе с Ref. [27 •] сообщает о новых условиях химического зондирования, которые вводят мутации в сайты модификаций. Кроме того, они используют корреляционный анализ для выявления контактов в пространстве и кластерный анализ для выявления различных состояний в структурном ансамбле. [Бесплатная статья PMC] [PubMed] [Google Scholar] 27 •. Зигфрид Н.А., Пусан С., Райс ГМ, Нельсон Дж. А., Недели КМ. Обнаружение мотивов РНК с помощью методов SHAPE и мутационного профилирования (SHAPE-MaP) Nat.2014; 11: 959–965. См. Аннотацию к исх. [26 ••] [Бесплатная статья PMC] [PubMed] [Google Scholar] 28. Helwak A, Kudla G, Dudnakova T, Tollervey D. Картирование взаимодействия miRNA человека с помощью CLASH обнаруживает частое неканоническое связывание. Клетка. 2013; 153: 654–665. [Бесплатная статья PMC] [PubMed] [Google Scholar] 29. Kudla G, Granneman S, Hahn D, Beggs JD, Tollervey D. Перекрестное связывание, лигирование и секвенирование гибридов выявляют взаимодействия РНК-РНК в дрожжах. Proc Natl Acad Sci U S. A. 2011; 108: 10010–10015. [Бесплатная статья PMC] [PubMed] [Google Scholar] 30 ••.Sugimoto Y, Vigilante A, Darbo E, Zirra A, Militti C, D’Ambrogio A, Luscombe NM, Ule J. hiCLIP раскрывает in vivo атлас вторичных структур мРНК, распознаваемых Staufen 1. Nature. 2015; 519: 491–494. В этой статье используется перекрестное связывание и лигирование по близости для определения структур РНК, связанных с dsRBP STAU1, и выявлены дальнодействующие и альтернативные структуры РНК, которые невозможно найти с помощью методов химического зондирования. [Бесплатная статья PMC] [PubMed] [Google Scholar] 31. Рамани В., Цю Р., Шендурэ Дж.Высокопроизводительное определение структуры РНК путем лигирования близости. Nat Biotechnol. 2015; 33: 980–984. [Бесплатная статья PMC] [PubMed] [Google Scholar] 32. Cheng CY, Chou FC, Kladwang W, Tian S, Cordero P, Das R. Согласованные глобальные структуры сложных состояний РНК посредством многомерного химического картирования. Элиф. 2015; 4: e07600. [Бесплатная статья PMC] [PubMed] [Google Scholar] 33. Thakor N, Holcik M. IRES-опосредованная трансляция клеточной информационной РНК работает независимо от eIF2alpha во время стресса. Nucleic Acids Res.2012; 40: 541–552. [Бесплатная статья PMC] [PubMed] [Google Scholar]Роль РНК в биологии
РНКв той или иной форме затрагивает почти все в клетке. РНК выполняет широкий спектр функций, от трансляции генетической информации в молекулярные машины и структуры клетки до регулирования активности генов во время развития, клеточной дифференциации и изменения окружающей среды.
РНК — уникальный полимер. Как и ДНК, он может связываться с большой специфичностью как с ДНК, так и с другой РНК посредством комплементарного спаривания оснований.Он также может связывать определенные белки или небольшие молекулы, и, что примечательно, РНК может катализировать химические реакции, включая соединение аминокислот с образованием белков.
Все РНК в клетках сами по себе являются копиями последовательностей ДНК, содержащихся в генах хромосом клетки. Гены, которые копируются — «транскрибируются» — в инструкции по созданию индивидуальных белков, часто называют «кодирующими генами». Гены, которые продуцируют РНК, используемые для других целей, поэтому называются генами «некодирующей РНК».
Молекулы РНК собирают белки и модифицируют другие РНК
Несколько ключевых классов молекул РНК помогают преобразовывать информацию, содержащуюся в ДНК клетки, в функциональные генные продукты, такие как белки. Информационные РНК (мРНК) представляют собой копии отдельных генов, кодирующих белок, и служат в качестве амплифицированного считывания последовательности нуклеиновой кислоты каждого гена. Две ключевые некодирующие РНК участвуют в сборке белков, определяемых мРНК. Рибосомная РНК (рРНК) составляет основной структурный и ферментативный каркас рибосомы, машины, которая синтезирует белки в соответствии с инструкциями, содержащимися в последовательности мРНК.Трансферные РНК (тРНК) используют комплементарные пары оснований для декодирования трехбуквенных «слов» в мРНК, каждое из которых соответствует аминокислоте, которая должна быть последовательно включена в растущую белковую цепь.
Большинство молекул РНК, когда-то транскрибируемых из хромосомной ДНК, требуют структурных или химических модификаций, прежде чем они смогут функционировать. В эукариотических клетках мРНК собираются из более длинных транскриптов РНК сплайсосомой, которая состоит из сплайсосомных РНК и белков-партнеров. Сплайсосомные РНК помогают отбрасывать промежуточные последовательности (интроны) из пре-мРНК-транскриптов и сплавлять вместе сегменты (экзоны) мРНК для создания того, что может быть сложным набором различных мРНК, кодирующих белок, из одного гена.Многие некодирующие РНК также требуют посттранскрипционных модификаций. Например, рибосомные РНК подвергаются многочисленным химическим модификациям, которые необходимы для правильной сборки и функционирования рибосом. Эти модификации вводятся белковыми ферментами в сочетании со специализированными некодирующими РНК (называемыми мяРНК), которые соединяются в пары с рРНК и направляют модифицирующие ферменты в точные места на рРНК.
Некоторые РНК обладают внутренней ферментативной активностью и могут непосредственно катализировать реакции модификации РНК.Эти каталитические РНК включают определенные самосплайсинговые РНК-транскрипты, рибозимы и РНКазу P, фермент РНК, который обрезает концы предшественников тРНК практически во всех клетках.
Молекулы РНК регулируют экспрессию генов
Регулирование производства белков кодирующими генами является основой большей части клеточной и организменной структуры, дифференциации и физиологии. Различные классы некодирующих РНК участвуют в регуляции генов на многих уровнях, влияя на продукцию, стабильность или трансляцию конкретных продуктов гена мРНК.
У прокариот (например, бактерий) малые антисмысловые РНК проявляют различные регуляторные активности генов за счет спаривания оснований, специфичных для их мРНК-мишеней. Также у прокариот распространены рибопереключатели, некодирующие последовательности РНК, которые обычно функционируют как регуляторные домены, содержащиеся в более длинных мРНК. Рибопереключатели регулируют активность мРНК своего хозяина, связываясь с небольшими молекулами, такими как нуклеотиды или аминокислоты, ощущая изобилие этих малых молекул и регулируя гены, которые производят или используют их соответственно.
Эукариотические клетки содержат тысячи малых РНК, связанных с различными путями РНК-интерференции (РНКи). Например, микроРНК (миРНК) представляют собой регуляторные РНК длиной примерно 22 нт, которые образуются из более длинных транскриптов, содержащих определенный вид двухцепочечной «шпильочной» структуры. miRNA связываются с белком класса Argonaute, а пары оснований специфически связаны с мРНК, чтобы ингибировать их стабильность или трансляцию. У растений и животных есть сотни генов miRNA, и каждая miRNA может регулировать активность сотен генов, кодирующих белок.Следовательно, miRNA по отдельности и все вместе оказывают глубокое влияние на развитие и физиологию многоклеточных эукариот.
Малые интерферирующие РНК (миРНК) сходны по длине с микроРНК и также связаны с белками Argonaute. В отличие от миРНК, которые продуцируются из определенных генетических локусов, которые эволюционировали для регулирования мРНК, миРНК могут происходить практически из любой транскрибируемой области генома. siRNA обычно действуют непосредственно на локус, из которого они производятся.Итак, миРНК встречаются в клетках, гены которых находятся в постоянной саморегуляции с помощью РНКи.
Основная роль определенных классов малых некодирующих РНК — защита клетки от вирусов, транспозонов и других последовательностей нуклеиновых кислот, которые представляют потенциальную угрозу клеточному гомеостазу или стабильности генома. Ответ некоторых клеток на вирусную инфекцию включает продукцию миРНК, комплементарных вирусу. Многие эндогенные миРНК в эукариотических клетках определяют подавление транспозонов и повторяющихся последовательностей, которые уже находятся в геноме.Точно так же у животных Piwi-ассоциированные РНК (piRNA) способствуют целостности генома, подавляя транспозоны и повторяющиеся последовательности.
Другой класс регуляторных РНК состоит из различных видов более длинных некодирующих транскриптов, которые обычно функционируют, регулируя экспрессию отдаленных генетических локусов, часто подавляя или стимулируя их транскрипцию. Например, rox-РНК плодовой мушки, по-видимому, облегчают ремоделирование структуры хромосом, позволяя транскрибировать мужскую X-хромосому в два раза быстрее, чем одиночная X-хромосома у женщин, у которых есть две X-хромосомы.Точно так же РНК Xist у млекопитающих помогает инактивировать одну из двух Х-хромосом у женщин, позволяя мужчинам и женщинам иметь эквивалентные уровни экспрессии генов из Х-хромосомы. Xist является одним из примеров более широкого класса очень универсальных регуляторных РНК, известных как длинные межгенные некодирующие РНК (lincRNA). lincRNA могут действовать как каркасы для сборки комплексов транскрипционных регуляторных белков и могут способствовать привлечению определенных комбинаций белковых регуляторов к конкретным генам.
Произошла ошибка при настройке пользовательского файла cookie
Этот сайт использует файлы cookie для повышения производительности. Если ваш браузер не принимает файлы cookie, вы не можете просматривать этот сайт.
Настройка вашего браузера для приема файлов cookie
Существует множество причин, по которым cookie не может быть установлен правильно. Ниже приведены наиболее частые причины:
- В вашем браузере отключены файлы cookie. Вам необходимо сбросить настройки своего браузера, чтобы он принимал файлы cookie, или чтобы спросить, хотите ли вы принимать файлы cookie.
- Ваш браузер спрашивает вас, хотите ли вы принимать файлы cookie, и вы отказались. Чтобы принять файлы cookie с этого сайта, нажмите кнопку «Назад» и примите файлы cookie.
- Ваш браузер не поддерживает файлы cookie. Если вы подозреваете это, попробуйте другой браузер.
- Дата на вашем компьютере в прошлом. Если часы вашего компьютера показывают дату до 1 января 1970 г., браузер автоматически забудет файл cookie. Чтобы исправить это, установите правильное время и дату на своем компьютере.
- Вы установили приложение, которое отслеживает или блокирует установку файлов cookie. Вы должны отключить приложение при входе в систему или проконсультироваться с системным администратором.
Почему этому сайту требуются файлы cookie?
Этот сайт использует файлы cookie для повышения производительности, запоминая, что вы вошли в систему, когда переходите со страницы на страницу. Чтобы предоставить доступ без файлов cookie потребует, чтобы сайт создавал новый сеанс для каждой посещаемой страницы, что замедляет работу системы до неприемлемого уровня.
Что сохраняется в файле cookie?
Этот сайт не хранит ничего, кроме автоматически сгенерированного идентификатора сеанса в cookie; никакая другая информация не фиксируется.
Как правило, в cookie-файлах может храниться только информация, которую вы предоставляете, или выбор, который вы делаете при посещении веб-сайта. Например, сайт не может определить ваше имя электронной почты, пока вы не введете его. Разрешение веб-сайту создавать файлы cookie не дает этому или любому другому сайту доступа к остальной части вашего компьютера, и только сайт, который создал файл cookie, может его прочитать.
Расшифровка полезности некодирующих РНК в качестве маркеров рака груди | Журнал трансляционной медицины
Сеть CGA. Комплексные молекулярные портреты опухолей груди человека. Природа. 2012; 490: 61–70.
Артикул CAS Google Scholar
Torre LA, et al. Глобальная статистика рака, 2012. CA Cancer J Clin. 2015; 65: 87–108. DOI: 10,3322 / caac.21262.
PubMed Статья Google Scholar
Serpico D, Molino L, Di Cosimo S. microRNAs в развитии и лечении рака груди. Лечение рака Ред. 2014; 40: 595–604. DOI: 10.1016 / j.ctrv.2013.11.002.
CAS PubMed Статья Google Scholar
Сорли Т. Молекулярные портреты рака груди: подтипы опухолей как отдельные заболевания. Eur J Cancer. 2004; 40: 2667–75.
PubMed Статья CAS Google Scholar
Verma A, Kaur J, Mehta K. Обновление молекулярной онкологии: профилирование экспрессии генов рака груди. Азиатский J Oncol. 2015; 1:65.
Артикул Google Scholar
Fitzgibbons PL, et al. Шаблон для отчета о результатах тестирования биомаркеров образцов от пациентов с карциномой груди. Arch Pathol Lab Med. 2014; 138: 595–601. DOI: 10.5858 / arpa.2013-0566-CP.
PubMed Статья Google Scholar
Hammond ME, et al. Руководство Американского общества клинической онкологии / Колледжа американских патологов по иммуногистохимическому тестированию рецепторов эстрогена и прогестерона при раке груди (полная версия). Arch Pathol Lab Med. 2010; 134: e48–72. DOI: 10.1043 / 1543-2165-134.7.e48.
CAS PubMed Google Scholar
Haque R, et al. Влияние подтипов рака груди и лечения на выживаемость: анализ за два десятилетия.Биомарк эпидемиологии рака Пред. 2012; 21: 1848–55. DOI: 10.1158 / 1055-9965.EPI-12-0474.
Артикул Google Scholar
Ades F, et al. Рак молочной железы Luminal B: молекулярная характеристика, клиническое ведение и перспективы на будущее. J Clin Oncol. 2014; 32: 2794–803. DOI: 10.1200 / JCO.2013.54.1870.
PubMed Статья Google Scholar
Blows FM, et al.Подтип рака груди с помощью иммуногистохимии для изучения взаимосвязи между подтипом и краткосрочной и долгосрочной выживаемостью: совместный анализ данных для 10 159 случаев из 12 исследований. PLoS Med. 2010; 7: e1000279.
PubMed PubMed Central Статья Google Scholar
Ciriello G, et al. Молекулярное разнообразие опухолей просвета молочной железы. Лечение рака груди Res. 2013; 141: 409–20. DOI: 10.1007 / s10549-013-2699-3.
CAS PubMed PubMed Central Статья Google Scholar
Zhang MH, Man HT, Zhao XD, Dong N, Ma SL. Молекулярные сигнатуры и терапевтические возможности рака молочной железы, положительного по рецепторам эстрогена (обзор). Биомед Реп. 2014; 2: 41–52. DOI: 10.3892 / br.2013.187.
PubMed Google Scholar
Gyrffy B, et al. Мультигенные прогностические тесты при раке груди: прошлое, настоящее, будущее.Рак молочной железы Res. 2015; 17:11.
PubMed PubMed Central Статья CAS Google Scholar
Cheang MC, van de Rijn M, Nielsen TO. Профилирование экспрессии генов рака груди. Энн Рев Патол. 2008; 3: 67–97. DOI: 10.1146 / annurev.pathmechdis.3.121806.151505.
CAS Статья Google Scholar
Lu J, et al. Профили экспрессии микроРНК позволяют классифицировать рак человека.Природа. 2005; 435: 834–8. DOI: 10,1038 / природа03702.
CAS PubMed Статья Google Scholar
Mitchell PS, et al. Циркулирующие микроРНК как стабильные маркеры крови для обнаружения рака. Proc Natl Acad Sci. 2008. 105: 10513–8.
CAS PubMed PubMed Central Статья Google Scholar
Xi Y, et al. Систематический анализ экспрессии микроРНК РНК, экстрагированной из свежезамороженных и фиксированных формалином образцов, залитых парафином.РНК. 2007; 13: 1668–74.
CAS PubMed PubMed Central Статья Google Scholar
Gilad S, et al. МикроРНК сыворотки являются многообещающими новыми биомаркерами. PLoS ONE. 2008; 3: e3148.
PubMed PubMed Central Статья CAS Google Scholar
Чен Х, Лян Х, Чжан Дж, Дзен К., Чжан Си-И. Секретируемые микроРНК: новая форма межклеточной коммуникации.Trends Cell Biol. 2012; 22: 125–32.
CAS PubMed Статья Google Scholar
Palma J, et al. МикроРНК экспортируются из злокачественных клеток в индивидуальных частицах. Nucleic Acids Res. 2012; 40: 9125–38.
CAS PubMed PubMed Central Статья Google Scholar
Маттик Ю.С., Макунин И.В. Некодирующая РНК. Hum Mol Genet. 2006. 15 (1): 17–29.DOI: 10,1093 / hmg / ddl046.
Артикул CAS Google Scholar
Sharp SJ, Schaack J, Cooley L, Burke DJ, Soil D. Структура и транскрипция гена эукариотической тРНК. CRC Crit Rev Biochem. 1985; 19: 107–44.
CAS PubMed Статья Google Scholar
Pavon-Eternod M, et al. Сверхэкспрессия тРНК при раке груди и функциональные последствия.Nucleic Acids Res. 2009; 37: 7268–80. DOI: 10.1093 / nar / gkp787.
CAS PubMed PubMed Central Статья Google Scholar
Pederson T. Регуляторные РНК, полученные из транспортной РНК? РНК. 2010; 16: 1865–9. DOI: 10.1261 / rna.2266510.
CAS PubMed PubMed Central Статья Google Scholar
Park IH, et al. Идентификация и клинические последствия циркулирующих микроРНК для рака молочной железы, положительного по рецепторам эстрогена.Tumor Biol. 2014; 35: 12173–80. DOI: 10.1007 / s13277-014-2525-5.
CAS PubMed Статья Google Scholar
Dhahbi JM, Spindler SR, Atamna H, Boffelli D, Martin DI. Глубокое секвенирование малых РНК сыворотки выявляет паттерны экспрессии половины 5′-тРНК и фрагмента YRNA, связанные с раком груди. Биомарк рака. 2014; 6: 37.
CAS PubMed PubMed Central Статья Google Scholar
Spizzo R, Almeida MI, Colombatti A, Calin GA. Длинные некодирующие РНК и рак: новый рубеж трансляционных исследований и поисков. Онкоген. 2012; 31: 4577–87.
CAS PubMed PubMed Central Статья Google Scholar
Kung JT, Colognori D, Lee JT. Длинные некодирующие РНК: прошлое, настоящее и будущее. Генетика. 2013; 193: 651–69. DOI: 10.1534 / genetics.112.146704.
CAS PubMed PubMed Central Статья Google Scholar
Kung JT, Colognori D, Lee JT. Длинные некодирующие РНК: прошлое, настоящее и будущее. Генетика. 2013; 193: 651–69.
CAS PubMed PubMed Central Статья Google Scholar
Малих С., Саидиджам М., Малих Н. Краткий обзор длинных некодирующих РНК: новая парадигма в патогенезе, диагностике и терапии рака груди. Tumor Biol. 2016; 37: 1479–85. DOI: 10.1007 / s13277-015-4572-у.
CAS PubMed Статья Google Scholar
Ding X и др. Длинные межгенные некодирующие РНК (LincRNA), идентифицированные с помощью RNA-seq при раке груди. PLoS ONE. 2014; 9: e103270. DOI: 10.1371 / journal.pone.0103270.
PubMed PubMed Central Статья CAS Google Scholar
Su X, et al. Комплексный анализ длинных некодирующих РНК в клинических подтипах рака груди человека. Oncotarget. 2014; 5: 9864–76. DOI: 10.18632 / oncotarget.2454.
PubMed PubMed Central Статья Google Scholar
Xu N, et al. Клиническое значение высокой экспрессии циркулирующей сывороточной днРНК RP11-445h32.4 у пациентов с раком груди: китайское популяционное исследование. Tumor Biol. 2015; 36: 7659–65. DOI: 10.1007 / s13277-015-3469-0.
CAS PubMed Статья Google Scholar
Zhao W, Luo J, Jiao S. Комплексная характеристика длинных некодирующих РНК, связанных с подтипом рака, и их клиническое значение. Научный представитель2014; 4: 6591.
CAS PubMed PubMed Central Статья Google Scholar
Shi Y, et al. Длинная некодирующая РНК SPRY4-IT1 увеличивает пролиферацию клеток рака груди человека за счет усиления экспрессии ZNF703. Молочный рак. 2015; 14:51. DOI: 10.1186 / s12943-015-0318-0.
PubMed PubMed Central Статья CAS Google Scholar
Гупта Р.А. и др. Длинная некодирующая РНК HOTAIR перепрограммирует состояние хроматина, способствуя метастазированию рака. Природа. 2010; 464: 1071–6. DOI: 10,1038 / природа08975.
CAS PubMed PubMed Central Статья Google Scholar
Sørensen KP, et al. Длинная некодирующая РНК HOTAIR является независимым прогностическим маркером метастазов при первичном раке молочной железы, положительном по рецепторам эстрогена. Лечение рака груди Res. 2013; 142: 529–36.
PubMed Статья CAS Google Scholar
Arun G, et al. Дифференциация опухолей молочной железы и уменьшение метастазов при потере днРНК Malat1. Genes Dev. 2016; 30: 34–51.
CAS PubMed PubMed Central Статья Google Scholar
Chi Y, et al. Роль BC040587 как предиктора неблагоприятного исхода рака груди. Cancer Cell Int. 2014; 14: 1.
Артикул CAS Google Scholar
Hu P, et al. NBAT1 подавляет метастазирование рака груди, регулируя DKK1 через PRC2. Oncotarget. 2015; 6: 32410–25. DOI: 10.18632 / oncotarget.5609.
PubMed PubMed Central Google Scholar
Xu SP, et al. Подавление длинной некодирующей РНК EGOT коррелирует со злокачественным статусом и плохим прогнозом рака груди. Tumor Biol. 2015; 36: 9807–12. DOI: 10.1007 / s13277-015-3746-у.
CAS PubMed Статья Google Scholar
Шен Й и др. Прогностическая и прогностическая ценность длинной некодирующей РНК LINC00472 при раке груди. Oncotarget. 2015; 6: 8579–92. DOI: 10.18632 / oncotarget.3287.
PubMed PubMed Central Статья Google Scholar
Godinho MF, et al. Актуальность BCAR4 в устойчивости к тамоксифену и опухолевой агрессивности рака молочной железы человека. Br J Рак. 2010; 103: 1284–91. DOI: 10.1038 / sj.bjc.6605884.
CAS PubMed PubMed Central Статья Google Scholar
Йонссон П. и др. Одномолекулярное секвенирование выявляет регулируемые эстрогеном клинически значимые днРНК при раке груди. Мол Эндокринол. 2015; 29: 1634–45. DOI: 10.1210 / me.2015-1153.
CAS PubMed Статья Google Scholar
Shi SJ, et al. LncRNA-ATB способствует устойчивости к трастузумабу и каскаду инвазии-метастазирования при раке молочной железы. Oncotarget. 2015; 6: 11652–63. DOI: 10.18632 / oncotarget.3457.
PubMed PubMed Central Статья Google Scholar
Bedrosian JW, Foekens JA, Berindan-Neagoe I, Calin GA. CCAT2, новая длинная некодирующая РНК при раке груди: исследование экспрессии и клинические корреляции. 2013.
Chen YM, Liu Y, Wei HY, Lv KZ, Fu P. Linc-ROR индуцирует эпителиально-мезенхимальный переход и способствует лекарственной устойчивости и инвазии клеток рака груди. Tumor Biol. 2016 ;. DOI: 10.1007 / s13277-016-4909-1.
PubMed Central Google Scholar
Ruby JG, Jan CH, Bartel DP. Предшественники интронных микроРНК, которые обходят процессинг Дроша. Природа. 2007; 448: 83–6.
CAS PubMed PubMed Central Статья Google Scholar
Huntzinger E, Izaurralde E. Молчание генов с помощью микроРНК: вклад репрессии трансляции и распада мРНК. Nat Rev Genet. 2011; 12: 99–110. DOI: 10,1038 / NRG2936.
CAS PubMed Статья Google Scholar
Calin GA, et al. Частые делеции и подавление микро-РНК генов miR15 и miR16 в 13q14 при хроническом лимфолейкозе. Proc Natl Acad Sci. 2002; 99: 15524–9.
CAS PubMed PubMed Central Статья Google Scholar
Bertoli G, Cava C, Castiglioni I. microRNAs: новые биомаркеры для диагностики, прогноза, прогноза терапии и терапевтических инструментов для рака груди. Тераностика. 2015; 5: 1122–43.DOI: 10.7150 / thno.11543.
CAS PubMed PubMed Central Статья Google Scholar
Goh JN, et al. микроРНК при раке молочной железы: регуляторные роли, определяющие признаки рака. Биол Рев Камб Филос Соц. 2016; 91: 409–28. DOI: 10.1111 / brv.12176.
PubMed Статья Google Scholar
Keklikoglou I, et al. Семейство MicroRNA-520/373 действует как опухолевый супрессор при раке молочной железы, отрицательном по рецепторам эстрогена, воздействуя на сигнальные пути NF-κB и TGF-β.Онкоген. 2012; 31: 4150–63.
CAS PubMed Статья Google Scholar
Sempere LF, et al. Измененная экспрессия MicroRNA ограничена конкретными субпопуляциями эпителиальных клеток при раке молочной железы. Cancer Res. 2007. 67: 11612–20. DOI: 10.1158 / 0008-5472.CAN-07-5019.
CAS PubMed Статья Google Scholar
Ma L, Teruya-Feldstein J, Weinberg RA.Инвазия опухоли и метастазирование, инициированное микроРНК-10b при раке груди. Природа. 2007; 449: 682–8. DOI: 10,1038 / природа06174.
CAS PubMed Статья Google Scholar
Volinia S, et al. Сигнатуры рака груди для инвазивности и прогноза, определяемые глубоким секвенированием микроРНК. Proc Natl Acad Sci USA. 2012; 109: 3024–9. DOI: 10.1073 / pnas.1200010109.
CAS PubMed PubMed Central Статья Google Scholar
Иорио М.В. и др. Нарушение регуляции экспрессии генов микроРНК при раке груди человека. Cancer Res. 2005; 65: 7065–70. DOI: 10.1158 / 0008-5472.CAN-05-1783.
CAS PubMed Статья Google Scholar
Heneghan HM, et al. Циркулирующие микроРНК как новые минимально инвазивные биомаркеры рака груди. Ann Surg. 2010; 251: 499–505.
PubMed Статья Google Scholar
Wu Q, Wang C, Lu Z, Guo L, Ge Q. Анализ сывороточных микроРНК генома для обнаружения рака груди. Clin Chim Acta. 2012; 413: 1058–65. DOI: 10.1016 / j.cca.2012.02.016.
CAS PubMed Статья Google Scholar
Чжан Х., Ли Б., Чжао Х., Чанг Дж. Экспрессия и клиническое значение сывороточного miR-205 для рака груди и его роль в обнаружении рака у человека. Int J Clin Exp Med. 2015; 8: 3034–43.
CAS PubMed PubMed Central Google Scholar
Zhao F-L, et al. Уровень микроРНК-195 в сыворотке подавляется при раке груди: это потенциальный маркер для диагностики рака груди. Mol Biol Rep. 2014; 41: 5913–22.
CAS PubMed Статья Google Scholar
Hu Z, et al. Профиль микроРНК в сыворотке и риск рака груди: использование miR-484/191 в качестве эндогенного контроля. Канцерогенез. 2012. 33 (4): 828–34.
CAS PubMed Статья Google Scholar
Zhang L, et al. Циркулирующая сигнатура miRNA в качестве диагностического биомаркера для неинвазивной ранней диагностики рака груди. Лечение рака груди Res. 2015; 154: 423–34.
CAS PubMed Статья Google Scholar
Ng EK, et al. Циркулирующие микроРНК как специфические биомаркеры для обнаружения рака груди. PLoS ONE. 2013; 8: e53141.
CAS PubMed PubMed Central Статья Google Scholar
Cuk K, et al. Панель плазменных микроРНК для минимально инвазивной диагностики рака груди. PLoS ONE. 2013; 8: e76729. DOI: 10.1371 / journal.pone.0076729.
CAS PubMed PubMed Central Статья Google Scholar
Eissa S, Matboli M, Shehata HH. Панель микроРНК на основе ткани груди выделяет микроРНК-23a и выбранные гены-мишени в качестве предполагаемых биомаркеров рака груди. Перевод Рез. 2015; 165: 417–27.
CAS PubMed Статья Google Scholar
Hui AB, et al. Надежное глобальное профилирование микро-РНК с фиксированными формалином и залитыми парафином тканями рака груди. Lab Invest. 2009. 89: 597–606. DOI: 10.1038 / labinvest.2009.12.
CAS PubMed Статья Google Scholar
Krishnan P, et al. Секвенирование следующего поколения определяет miR-574-3p и miR-660-5p как потенциальные новые прогностические маркеры рака груди. BMC Genom. 2015; 16: 1.
Артикул CAS Google Scholar
Мар-Агилар Ф. и др. Профилирование циркулирующих микроРНК в сыворотке для идентификации потенциальных биомаркеров рака молочной железы. Маркеры Дис. 2013; 34: 163–9. DOI: 10.3233 / DMA-120957.
CAS PubMed PubMed Central Статья Google Scholar
Chan M, et al. Идентификация циркулирующих сигнатур микроРНК для обнаружения рака груди. Clin Cancer Res. 2013; 19: 4477–87. DOI: 10.1158 / 1078-0432.CCR-12-3401.
CAS PubMed Статья Google Scholar
Antolin S, et al. Циркулирующие miR-200c и miR-141 и исходы у пациентов с раком груди. BMC Рак. 2015; 15: 297. DOI: 10.1186 / s12885-015-1238-5.
PubMed PubMed Central Статья CAS Google Scholar
Frères P, et al. Инструмент для скрининга рака груди на основе циркулирующих микроРНК. Oncotarget. 2016; 7 (5): 5416–28.
PubMed Google Scholar
Matamala N, et al. Профили экспрессии микроРНК опухоли позволяют идентифицировать циркулирующие микроРНК для раннего выявления рака молочной железы. Clin Chem. 2015; 61: 1098–106. DOI: 10.1373 / Clinchem.2015.238691.
CAS PubMed Статья Google Scholar
Лоури AJ, et al. Сигнатуры микроРНК позволяют прогнозировать статус рецептора эстрогена, рецептора прогестерона и рецептора HER2 / neu при раке груди. Рак молочной железы Res. 2009; 11: R27. DOI: 10.1186 / bcr2257.
PubMed PubMed Central Статья CAS Google Scholar
Cizeron-Clairac G, et al. MiR-190b, самая высокая активируемая miRNA в ERα-положительных по сравнению с ERα-отрицательными опухолями молочной железы, новый биомаркер рака молочной железы? BMC Рак. 2015; 15: 499.
PubMed PubMed Central Статья CAS Google Scholar
He YJ, et al. miR-342 связан с экспрессией рецептора эстрогена-α и ответом на тамоксифен при раке груди.Exp Ther Med. 2013; 5: 813–8.
PubMed PubMed Central Google Scholar
Wang PY, et al. Повышенная экспрессия циркулирующей miR-182 как нового биомаркера рака груди. Oncol Lett. 2013; 6: 1681–6.
CAS PubMed PubMed Central Google Scholar
Wu X, et al. De novo секвенирование циркулирующих миРНК позволяет выявить новые маркеры, предсказывающие клинический исход местно-распространенного рака молочной железы.J Transl Med. 2012; 10: 42. DOI: 10.1186 / 1479-5876-10-42.
CAS PubMed PubMed Central Статья Google Scholar
Zhu W., Qin W, Atasoy U, Sauter ER. Циркулирующие микроРНК при раке груди и у здоровых людей. BMC Res Notes. 2009; 2: 89. DOI: 10.1186 / 1756-0500-2-89.
PubMed PubMed Central Статья CAS Google Scholar
Tashkandi H, Shah N, Patel Y, Chen H. Идентификация новых биомаркеров miRNA, связанных с HER2-положительным раком молочной железы. Онкология. 2015; 2: 924–9.
PubMed PubMed Central Google Scholar
Blenkiron C, et al. Профили экспрессии микроРНК рака груди человека позволяют идентифицировать новые маркеры подтипа опухоли. Genome Biol. 2007; 8: R214. DOI: 10.1186 / ГБ-2007-8-10-r214.
PubMed PubMed Central Статья CAS Google Scholar
Endo Y, et al. miR-1290 и ее потенциальные мишени связаны с характеристиками альфа-положительного рака груди по рецептору эстрогена. Endocr Relat Cancer. 2013; 20: 91–102. DOI: 10.1530 / ERC-12-0207.
CAS PubMed Статья Google Scholar
Иорио М.В., Казалини П., Тальябу Е., Менар С., Кроче С.М. Профилирование микроРНК как инструмент для понимания прогноза, терапевтического ответа и устойчивости при раке груди. Eur J Cancer.2008; 44: 2753–9.
CAS PubMed Статья Google Scholar
Shell S, et al. Выражение Let-7 определяет две стадии дифференцировки рака. Proc Natl Acad Sci USA. 2007. 104: 11400–5. DOI: 10.1073 / pnas.0704372104.
CAS PubMed PubMed Central Статья Google Scholar
Джемал А., Сигель Р., Сюй Дж., Уорд Э. Статистика рака, 2010 г.CA Cancer J Clin. 2010. 60: 277–300. DOI: 10.3322 / caac.20073.
PubMed Статья Google Scholar
Madhavan D, et al. Циркулирующие миРНК как суррогатные маркеры циркулирующих опухолевых клеток и прогностические маркеры при метастатическом раке молочной железы. Clin Cancer Res. 2012; 18: 5972–82.
CAS PubMed Статья Google Scholar
млн лет L, et al. miR-9, активированная MYC / MYCN микроРНК, регулирует E-кадгерин и метастазирование рака.Nat Cell Biol. 2010; 12: 247–56. DOI: 10,1038 / NCB2024.
CAS PubMed PubMed Central Google Scholar
Чен В., Цай Ф, Чжан Б., Барекати З., Чжун XY. Уровень циркулирующих miRNA-10b и miRNA-373 при обнаружении метастазов рака молочной железы в лимфатические узлы: потенциальные биомаркеры. Tumor Biol. 2013; 34: 455–62. DOI: 10.1007 / s13277-012-0570-5.
CAS PubMed Статья Google Scholar
Song B, et al. МикроРНК-21 регулирует инвазию рака молочной железы, частично воздействуя на тканевый ингибитор экспрессии металлопротеиназы 3. J Exp Clin Cancer Res. 2010; 29: 29. DOI: 10.1186 / 1756-9966-29-29.
CAS PubMed PubMed Central Статья Google Scholar
Gebeshuber CA, Zatloukal K, Martinez J. miR-29a подавляет тристетрапролин, который является регулятором полярности эпителия и метастазирования. EMBO Rep. 2009; 10: 400–5.
CAS PubMed PubMed Central Статья Google Scholar
Petrović N, et al. Изменения уровня экспрессии miR-155 могут быть связаны с начальными фазами патогенеза рака груди и метастазами в лимфатические узлы. Биомаркеры рака. 2016; 16: 385–94.
PubMed Статья CAS Google Scholar
Huang Q, et al. МикроРНК miR-373 и miR-520c способствуют инвазии опухоли и метастазированию.Nat Cell Biol. 2008; 10: 202–10. DOI: 10,1038 / ncb1681.
CAS PubMed Статья Google Scholar
Schwarzenbach H, Milde-Langosch K, Steinbach B, Muller V, Pantel K. Диагностический потенциал PTEN-targeting miR-214 в крови пациентов с раком груди. Лечение рака груди Res. 2012; 134: 933–41. DOI: 10.1007 / s10549-012-1988-6.
CAS PubMed Статья Google Scholar
Shi W, et al. MicroRNA-301 опосредует пролиферацию и инвазию при раке груди человека. Cancer Res. 2011; 71: 2926–37. DOI: 10.1158 / 0008-5472.CAN-10-3369.
CAS PubMed Статья Google Scholar
Жаны и др. microRNA-548j действует как промотор метастазов при раке груди человека, воздействуя на тензин1. Молекулярная онкология. 2016; 10 (6): 838–49.
Артикул CAS Google Scholar
Ю. З. и др. микроРНК 17/20 подавляет клеточную инвазию и метастазирование опухоли при раке молочной железы с помощью гетеротипической передачи сигналов. Proc Natl Acad Sci USA. 2010; 107: 8231–6. DOI: 10.1073 / pnas.1002080107.
CAS PubMed PubMed Central Статья Google Scholar
Png KJ, Halberg N, Yoshida M, Tavazoie SF. Реглон микроРНК, который опосредует рекрутирование эндотелия и метастазирование раковыми клетками. Природа. 2012; 481: 190–4.DOI: 10,1038 / природа10661.
CAS Статья Google Scholar
Li XF, Yan PJ, Shao ZM. Подавление miR-193b способствует усилению экспрессии активатора плазминогена урокиназного типа (uPA) и прогрессированию опухоли и инвазии при раке груди человека. Онкоген. 2009; 28: 3937–48. DOI: 10.1038 / onc.2009.245.
CAS PubMed Статья Google Scholar
Song G, Zhang Y, Wang L. MicroRNA-206 нацелена на notch4, активирует апоптоз и подавляет миграцию опухолевых клеток и образование очагов. J Biol Chem. 2009; 284: 31921–7. DOI: 10.1074 / jbc.M109.046862.
CAS PubMed PubMed Central Статья Google Scholar
Tavazoie SF, et al. Эндогенные микроРНК человека, подавляющие метастазирование рака груди. Природа. 2008; 451: 147–52.
CAS PubMed PubMed Central Статья Google Scholar
Li Q и др. Вовлечение регуляторной петли обратной связи NF-κB / miR-448 в индуцированный химиотерапией эпителиально-мезенхимальный переход клеток рака груди. Смерть клетки отличается. 2011; 18: 16–25.
PubMed Статья CAS Google Scholar
Hu J-Y, et al. miR-601 является прогностическим маркером и подавляет рост и инвазию клеток, воздействуя на PTP4A1 при раке молочной железы. Biomed Pharmacother. 2016; 79: 247–53.
CAS PubMed Статья Google Scholar
Zhang J, et al. MicroRNA-138 модулирует метастазирование и EMT в клетках рака груди, воздействуя на виментин. Biomed Pharmacother. 2016; 77: 135–41. DOI: 10.1016 / j.biopha.2015.12.018.
CAS PubMed Статья Google Scholar
Pardo OE, et al. miR-515-5p контролирует миграцию раковых клеток посредством регуляции MARK4. EMBO Rep. 2016; 17 (4): 570–84.
CAS PubMed PubMed Central Статья Google Scholar
Zhao S, et al. MicroRNA-203 регулирует рост и метастазирование рака груди. Cell Physiol Biochem. 2015; 37: 35–42. DOI: 10,1159 / 000430331.
CAS PubMed Статья Google Scholar
Gregory PA, et al. Семейство miR-200 и miR-205 регулируют переход эпителия в мезенхиму, воздействуя на ZEB1 и SIP1. Nat Cell Biol. 2008; 10: 593–601. DOI: 10,1038 / ncb1722.
CAS PubMed Статья Google Scholar
Ян L-X и др. Сверхэкспрессия микроРНК miR-21 при раке груди человека связана с продвинутой клинической стадией, метастазами в лимфатические узлы и плохим прогнозом пациента. РНК. 2008; 14: 2348–60.
CAS PubMed PubMed Central Статья Google Scholar
Markou A, Yousef GM, Stathopoulos E, Georgoulias V, Lianidou E. Прогностическое значение микроРНК, связанных с метастазами, у пациентов с ранним раком молочной железы с длительным периодом наблюдения.Clin Chem. 2014; 60: 197–205. DOI: 10.1373 / Clinchem.2013.210542.
CAS PubMed Статья Google Scholar
Camps C, et al. hsa-miR-210 индуцируется гипоксией и является независимым прогностическим фактором рака груди. Clin Cancer Res. 2008; 14: 1340–8.
CAS PubMed Статья Google Scholar
Rothe F, et al. Глобальное профилирование экспрессии микроРНК позволяет идентифицировать MiR-210, связанный с пролиферацией опухолей, инвазией и плохим клиническим исходом при раке молочной железы.PLoS ONE. 2011; 6: e20980. DOI: 10.1371 / journal.pone.0020980.
CAS PubMed PubMed Central Статья Google Scholar
Cheng C-W, et al. MicroRNA-30a подавляет миграцию и инвазию клеток за счет подавления экспрессии виментина и является потенциальным прогностическим маркером рака груди. Лечение рака груди Res. 2012; 134: 1081–93.
CAS PubMed Статья Google Scholar
Zhang N, et al. MicroRNA-30a подавляет рост и метастазирование опухоли груди, воздействуя на метадгерин. Онкоген. 2014; 33: 3119–28. DOI: 10.1038 / onc.2013.286.
CAS PubMed Статья Google Scholar
D’Aiuto F, et al. miR-30e * является независимым прогностическим маркером, специфичным для подтипа, при раке молочной железы. Br J Рак. 2015; 113: 290–8.
PubMed PubMed Central Статья CAS Google Scholar
Shimono Y, et al. Подавление miRNA-200c связывает стволовые клетки рака груди с нормальными стволовыми клетками. Клетка. 2009. 138: 592–603. DOI: 10.1016 / j.cell.2009.07.011.
CAS PubMed PubMed Central Статья Google Scholar
Liu S, Wicha MS. Стволовые клетки рака груди. J Clin Oncol. 2010. 28: 4006–12. DOI: 10.1200 / JCO.2009.27.5388.
CAS PubMed PubMed Central Статья Google Scholar
Бейли С.Т., Вестерлинг Т., Браун М. Потеря регулируемой эстрогеном экспрессии микроРНК увеличивает передачу сигналов HER2 и является прогностическим признаком плохого исхода при раке просвета молочной железы. Cancer Res. 2015; 75: 436–45.
CAS PubMed Статья Google Scholar
Gasparini P, et al. Профили экспрессии микроРНК определяют четыре сигнатуры микроРНК как новый диагностический и прогностический биомаркер при тройном негативном раке молочной железы.Oncotarget. 2014; 5: 1174–84.
PubMed PubMed Central Статья Google Scholar
Tuomarila M, et al. Сверхэкспрессия microRNA-200c предсказывает плохой исход у пациентов с PR-отрицательным раком молочной железы. PLoS ONE. 2014; 9: e109508. DOI: 10.1371 / journal.pone.0109508.
PubMed PubMed Central Статья CAS Google Scholar
Zhang B, et al.микроРНК 100 сенсибилизирует клетки рака молочной железы в просвете а к лечению паклитакселом частично за счет нацеливания на mTOR. Oncotarget. 2016; 7 (5): 5702–14.
PubMed Google Scholar
Peng F, et al. Идентификация дифференциально экспрессируемых miRNA у отдельного пациента с раком молочной железы и применение в персонализированной медицине. Онкогенез. 2016; 5: e194. DOI: 10.1038 / oncsis.2016.4.
CAS PubMed Статья Google Scholar
Zhu Y, et al. Снижение miR-128 в клетках, инициирующих опухоль молочной железы, вызывает химиотерапевтическую резистентность через Bmi-1 и ABCC5. Clin Cancer Res. 2011; 17: 7105–15. DOI: 10.1158 / 1078-0432.CCR-11-0071.
CAS PubMed Статья Google Scholar
Quesne JL, et al. Биологические и прогностические ассоциации miR-205 и let-7b при раке молочной железы, выявленные с помощью гибридизационного анализа in situ экспрессии микро-РНК в массивах архивной опухолевой ткани.J Pathol. 2012; 227: 306–14. DOI: 10.1002 / путь.3983.
CAS PubMed Статья Google Scholar
Leivonen SK, et al. Высокопроизводительный скрининг выявляет микроРНК, необходимые для роста HER2-положительных клеток рака молочной железы. Мол Онкол. 2014; 8: 93–104. DOI: 10.1016 / j.molonc.2013.10.001.
CAS PubMed Статья Google Scholar
Shen L, et al.miR-497 индуцирует апоптоз клеток рака молочной железы, нацеливаясь на Bcl-w. Exp Ther Med. 2012; 3: 475–80. DOI: 10.3892 / etm.2011.428.
CAS PubMed Google Scholar
Wu ZS, et al. Потеря экспрессии miR-133a, связанная с плохой выживаемостью при раке груди и восстановление экспрессии miR-133a, ингибировала рост и инвазию клеток рака груди. BMC Рак. 2012; 12:51. DOI: 10.1186 / 1471-2407-12-51.
CAS PubMed PubMed Central Статья Google Scholar
Li Y, Hong F, Yu Z. Снижение экспрессии микроРНК-206 при раке груди и ее связь с характеристиками заболевания и выживаемостью пациентов. J Int Med Res. 2013; 41: 596–602. DOI: 10.1177 / 0300060513485856.
CAS PubMed Статья Google Scholar
Донг Л-Л, Чен Л-М, Ван В-М, Чжан Л-М. Снижение экспрессии микроРНК-124 является независимым неблагоприятным прогностическим фактором для пациентов с раком груди.Diag Pathol. 2015; 10:45.
Артикул CAS Google Scholar
Xu F, et al. Снижение экспрессии семейства MicroRNA-200 при раке груди человека связано с метастазами в лимфатические узлы. Clin Transl Oncol. 2016; 18 (3): 1–6.
Google Scholar
Shinden Y, et al. miR-29b является индикатором прогноза у пациентов с раком груди. Мол Клин Онкол. 2015; 3: 919–23.
PubMed PubMed Central Google Scholar
Tang W, et al. MiR-27 как прогностический маркер прогрессирования рака груди и выживаемости пациентов. PLoS ONE. 2012; 7: e51702.
CAS PubMed PubMed Central Статья Google Scholar
Шен Р. и др. MiRNA-155 опосредует устойчивость к ТАМ, модулируя сигнальный путь SOCS6-STAT3 при раке груди.Am J Transl Res. 2015; 7: 2115–26.
PubMed PubMed Central Google Scholar
Farazi TA, et al. Анализ последовательности и экспрессии микроРНК в опухолях молочной железы путем глубокого секвенирования. Cancer Res. 2011; 71: 4443–53. DOI: 10.1158 / 0008-5472.CAN-11-0608.
CAS PubMed PubMed Central Статья Google Scholar
Mulrane L, et al. miR-187 является независимым прогностическим фактором рака молочной железы и обеспечивает повышенный инвазивный потенциал in vitro.Clin Cancer Res. 2012; 18: 6702–13.
CAS PubMed Статья Google Scholar
Kong W, et al. Повышающая регуляция miRNA-155 способствует ангиогенезу опухоли за счет воздействия на VHL и связана с плохим прогнозом и тройным отрицательным раком груди. Онкоген. 2014; 33: 679–89.
CAS PubMed Статья Google Scholar
Falkenberg N, et al.MiR-221 / -222 дифференцирует прогностические группы при запущенном раке молочной железы и влияет на клеточную инвазию. Br J Рак. 2013; 109: 2714–23. DOI: 10.1038 / bjc.2013.625.
CAS PubMed PubMed Central Статья Google Scholar
Lerebours F, et al. Профили экспрессии miRNA при воспалительном раке молочной железы идентифицируют сигнатуру 5-miRNA, предсказывающую агрессивность опухоли молочной железы. Int J Cancer. 2013; 133: 1614–23. DOI: 10.1002 / ijc.28171.
CAS PubMed Статья Google Scholar
Zehentmayr F, et al. Hsa-miR-375 является предиктором местного контроля на ранней стадии рака груди. Clin Epigen. 2016; 8: 1.
Артикул Google Scholar
Shen S, et al. Прогностическая модель тройного отрицательного рака груди, основанная на miR-27b-3p и статусе узлов. PLoS ONE. 2014; 9: e100664.DOI: 10.1371 / journal.pone.0100664.
PubMed PubMed Central Статья CAS Google Scholar
Hu J, et al. Идентификация микроРНК-93 как функционально дисрегулируемой миРНК при тройном негативном раке молочной железы. Tumor Biol. 2015; 36: 251–8.
CAS Статья Google Scholar
Сингх Р., Пн ГГ. Роль микроРНК в раке груди. Cancer Biol Ther.2013; 14: 201–12. DOI: 10.4161 / cbt.23296.
CAS PubMed PubMed Central Статья Google Scholar
Свобода М. и др. MiR-34b связан с клиническим исходом у пациентов с трижды отрицательным раком груди. Diagn Pathol. 2012; 7:31.
CAS PubMed PubMed Central Статья Google Scholar
Sahlberg KK, et al. Сигнатура сывороточной микроРНК предсказывает рецидив опухоли и выживаемость у пациентов с трижды отрицательным раком груди.Clin Cancer Res. 2015; 21: 1207–14.
Артикул CAS Google Scholar
Joosse SA, Muller V, Steinbach B, Pantel K, Schwarzenbach H. Циркулирующая бесклеточная РНК яичка MAGE-A, РНК BORIS, let-7b и miR-202 в крови пациентов с грудью рак и доброкачественные заболевания груди. Br J Рак. 2014; 111: 909–17. DOI: 10.1038 / bjc.2014.360.
CAS PubMed PubMed Central Статья Google Scholar
Mangolini A, et al. Диагностические и прогностические микроРНК в сыворотке крови больных раком груди, измеренные с помощью цифровой ПЦР. Biomark Res. 2015; 3: 12. DOI: 10.1186 / s40364-015-0037-0.
PubMed PubMed Central Статья Google Scholar
Roth C, et al. Циркулирующие микроРНК как маркеры крови для пациентов с первичным и метастатическим раком молочной железы. Рак молочной железы Res. 2010; 12: R90.
CAS PubMed PubMed Central Статья Google Scholar
Maillot G, et al. Широко распространенная эстроген-зависимая репрессия микроРНК, участвующих в росте опухолевых клеток груди. Cancer Res. 2009; 69: 8332–40. DOI: 10.1158 / 0008-5472.CAN-09-2206.
CAS PubMed Статья Google Scholar
Rodríguez-González FG, et al. Уровень экспрессии MicroRNA-30c является независимым предиктором клинической пользы эндокринной терапии при распространенном раке молочной железы, положительном по рецепторам эстрогена. Лечение рака груди Res.2011; 127: 43–51.
PubMed Статья CAS Google Scholar
Jung EJ, et al. Уровни микроРНК 210 в плазме коррелируют с чувствительностью к трастузумабу и наличием опухоли у пациентов с раком груди. Рак. 2012; 118: 2603–14. DOI: 10.1002 / cncr.26565.
CAS PubMed Статья Google Scholar
Cittelly DM, et al. Подавление miR-342 связано с опухолями молочной железы, устойчивыми к тамоксифену.Молочный рак. 2010; 9: 1.
Артикул CAS Google Scholar
Jansen MP, et al. Высокие уровни miR-26a и низкие уровни CDC2 связаны со снижением экспрессии EZh3 и с благоприятным исходом тамоксифена при метастатическом раке молочной железы. Лечение рака груди Res. 2012; 133: 937–47. DOI: 10.1007 / s10549-011-1877-4.
CAS PubMed Статья Google Scholar
Hoppe R, et al. Повышенная экспрессия miR-126 и miR-10a предсказывает продолжительное время без рецидива первичного эстроген-положительного рака молочной железы после лечения тамоксифеном. Eur J Cancer. 2013; 49: 3598–608. DOI: 10.1016 / j.ejca.2013.07.145.
CAS PubMed Статья Google Scholar
Miller TE, et al. MicroRNA-221/222 придает устойчивость к тамоксифену при раке молочной железы, воздействуя на p27Kip1. J Biol Chem. 2008; 283: 29897–903.DOI: 10.1074 / jbc.M804612200.
CAS PubMed PubMed Central Статья Google Scholar
Rao X, et al. МикроРНК-221/222 придает устойчивость к фулвестранту рака молочной железы, регулируя несколько сигнальных путей. Онкоген. 2011; 30: 1082–97. DOI: 10.1038 / onc.2010.487.
CAS PubMed Статья Google Scholar
Ward A, et al. MicroRNA-519a представляет собой новый онкомир, придающий устойчивость к тамоксифену за счет нацеливания на сеть генов-супрессоров опухолей при ER + раке молочной железы.J Pathol. 2014; 233: 368–79.
CAS PubMed PubMed Central Статья Google Scholar
Kazi AA, et al. HER2 регулирует экспрессию miRNA при устойчивом к летрозолу раке молочной железы. Cancer Res. 2014; 74: 1471–2.
Артикул Google Scholar
Gong C, et al. Повышающая регуляция miR-21 опосредует резистентность к терапии трастузумабом при раке груди.J Biol Chem. 2011; 286: 19127–37. DOI: 10.1074 / jbc.M110.216887.
CAS PubMed PubMed Central Статья Google Scholar
Ковальчук О. и др. Участие микроРНК-451 в устойчивости клеток рака молочной железы MCF-7 к химиотерапевтическому препарату доксорубицину. Mol Cancer Ther. 2008; 7: 2152–9. DOI: 10.1158 / 1535-7163.MCT-08-0021.
CAS PubMed Статья Google Scholar
Chen J, Tian W, Cai H, He H, Deng Y. Подавление микроРНК-200c связано с лекарственной устойчивостью при раке груди человека. Med Oncol. 2012; 29: 2527–34.
CAS PubMed Статья Google Scholar
Lu L, Ju F, Zhao H, Ma X. MicroRNA-134 модулирует устойчивость к доксорубицину в клетках рака груди человека, подавляя ABCC1. Biotechnol Lett. 2015; 37: 2387–94. DOI: 10.1007 / s10529-015-1941-у.
CAS PubMed Статья Google Scholar
Tang X и др. МикроРНК-16 сенсибилизирует клетки рака груди к паклитакселу за счет подавления экспрессии IKBKB. Oncotarget. 2016 ;. DOI: 10.18632 / oncotarget.8056.
Google Scholar
Xue J, et al. MiRNA-621 повышает чувствительность рака груди к химиотерапии, подавляя FBXO11 и повышая активность p53. Онкоген. 2016; 35: 448–58. DOI: 10.1038 / onc.2015.96.
CAS PubMed Статья Google Scholar
Wang H, et al. Циркуляция MiR-125b в качестве маркера, предсказывающего химиорезистентность при раке груди. PLoS ONE. 2012; 7: e34210.
CAS PubMed PubMed Central Статья Google Scholar
Zheng Y, et al. Сигнатура экспрессии микроРНК в ответе на неоадъювантную химиотерапию на основе таксана-антрациклина. J Рак. 2015; 6: 671–7. DOI: 10.7150 / jca.11616.
CAS PubMed PubMed Central Статья Google Scholar
Zhao R, et al. Плазма miR-221 как прогностический биомаркер химиорезистентности у пациентов с раком груди, которые ранее получали неоадъювантную химиотерапию. Онкол Рес Лечить. 2011; 34: 675–80.
CAS Google Scholar
Kong W, et al. MicroRNA-155 регулирует выживаемость, рост и химиочувствительность клеток, воздействуя на FOXO3a при раке груди. J Biol Chem. 2010; 285: 17869–79. DOI: 10.1074 / jbc.M110.101055.
CAS PubMed PubMed Central Статья Google Scholar
Gasparini P, et al. Защитная роль miR-155 при раке груди через нацеливание на RAD51 нарушает гомологичную рекомбинацию после облучения.


 Для диагностики используется слюна, кровь, мокрота, выделения половых органов и прочее биологические материалы, в зависимости от целей исследования.
Для диагностики используется слюна, кровь, мокрота, выделения половых органов и прочее биологические материалы, в зависимости от целей исследования.