Что такое ДНК и РНК в биологии?
Молекулярная биология является одним из важнейших разделов биологических наук и подразумевает детализированное изучение клеток живых организмов и их составляющих. В сферу ее исследований входит множество жизненно важных процессов, таких как рождение, дыхание, рост, смерть.
Бесценным открытием молекулярной биологии стала расшифровка генетического кода высших существ и определение способности клетки хранить и передавать генетическую информацию. Основная роль в этих процессах принадлежит нуклеиновым кислотам, которых в природе различают два вида – ДНК и РНК. Что представляют собой эти макромолекулы? Из чего они состоят и какие биологические функции выполняют?
Что такое ДНК?
ДНК расшифровывается как дезоксирибонуклеиновая кислота. Она представляет собой одну из трех макромолекул клетки (две другие – белки и рибонуклеиновая кислота), которая обеспечивает сохранение и передачу генетического кода развития и деятельности организмов. Простыми словами, ДНК – носитель генетической информации. В ее составе содержится генотип индивида, который обладает способностью к самовоспроизводству и передает информацию по наследству.
Как химическое вещество кислота была выделена из клеток еще в 1860-х годах, однако вплоть до середины XX столетия никто и не предполагал, что она способна хранить и передавать информацию.
Долгое время считалось, что эти функции выполняют белки, однако в 1953 году группа биологов сумела значительно расширить понимание сути молекулы и доказать первостепенную роль ДНК в сохранении и передаче генотипа. Находка стала открытием века, а ученые получили за свою работу Нобелевскую премию.
Из чего состоит ДНК?
ДНК является крупнейшей из биологических молекул и представляет собой четыре нуклеотида, состоящих из остатка фосфорной кислоты. В структурном отношении кислота достаточно сложная. Ее нуклеотиды соединяются между собой длинными цепями, которые объединяются попарно во вторичные структуры – двойные спирали.
ДНК имеет свойство повреждаться радиацией или различными окисляющими веществами, в силу чего в молекуле происходит процесс мутации. Функционирование кислоты напрямую зависит от ее взаимодействия с еще одной молекулой – белками. Вступая с ними во взаимосвязь в клетке, она образует вещество хроматин, внутри которого осуществляется реализация информации.
Что такое РНК?
РНК – это рибонуклеиновая кислота, содержащая в себе азотистые основания и остатки фосфорных кислот.
Существует гипотеза, что она является первой молекулой, получившей способность к самовоспроизводству еще в эпоху формирования нашей планеты – в добиологических системах. РНК и сегодня входит в геномы отдельных вирусов, выполняя в них ту роль, которую у высших существ играет ДНК.
Рибонуклеиновая кислота состоит из 4-х нуклеотидов, но вместо двойной спирали, как в ДНК, ее цепочки соединяются одинарной кривой. В нуклеотидах содержится рибоза, принимающая активное участие в обмене веществ. В зависимости от способности кодировать белок РНК делятся на матричную и некодирующие.
Первая выступает своего рода посредником в передаче закодированной информации рибосомам. Вторые не могут кодировать белки, но обладают другими возможностями – трансляцией и лигированием молекул.
Чем ДНК отличается от РНК?
По своему химическому составу кислоты очень схожи друг с другом. Обе относятся к линейным полимерам и являют собой N-гликозид, созданный из остатков пятеуглеродного сахара. Разница между ними в том, что сахарный остаток РНК – это рибоза, моносахарид из группы пентоз, легко растворяющийся в воде. Сахарный остаток ДНК – это дезоксирибоза, или производная рибозы, имеющая несколько иную структуру.
В отличие от рибозы, формирующей кольцо из 4 атомов углерода и 1 атома кислорода, в дезоксирибозе второй атом углерода замещается водородом. Еще одно отличие между ДНК и РНК заключается в их размерах – первая молекула более крупная. Кроме этого, среди четырех нуклеотидов, входящих в ДНК, один представляет собой азотистое основание под названием тимин, тогда как в РНК вместо тимина присутствует его разновидность – урацил.
Определение РНК общее значение и понятие. Что это такое РНК
Акроним РНК соответствует рибонуклеиновой кислоте, веществу, которое присутствует в эукариотических клетках и в прокариотических клетках. Это нуклеиновая кислота: полимер, который создается последовательностью мономеров, известных как нуклеотиды, которые связаны через фосфодиэфирные связи .
В случае РНК нуклеотиды, которые она представляет, являются рибонуклеотидами . В заключение можно сказать, что РНК представляет собой нуклеиновую кислоту, состоящую из ряда рибонуклеотидов.
РНК клеток представляет собой одну цепь и является линейной. Регулирование экспрессии генов, развитие катализа или передача важных данных в рамках биосинтеза белка может быть доверено. РНК работает в сочетании с ДНК : дезоксирибонуклеиновая кислота .
Каждый РНК-рибонуклеотид состоит из трех элементов: азотистого основания (урацил, гуанин, цитозин или аденин),
Линейная последовательность рибонуклеотидов известна как первичная структура РНК. Из этой структуры возникают другие уровни: вторичная структура (которая возникает в результате спаривания азотистых оснований в виде петли, спирали или иным образом) и третичная структура (произведение связей в пространстве, образованных атомами которые составляют молекулу).
Таким же образом, тот факт, что в РНК есть четыре различных основания нуклеотидов, не следует упускать из виду. Мы имеем в виду гуанин, урацил, аденин и унцитозин.
В соответствии с функцией, которую он выполняет в клетке, можно различить РНК-мессенджер (передает информацию от ДНК к рибосоме), РНК-рибосома
(составляет рибосомы рядом с белками), передавать РНК (несет определенную аминокислота в полипептид), интерферирующая РНК (устраняет экспрессию определенных генов) и регуляторная РНК (ответственная за регуляцию экспрессии генов), среди других типов.Предоставляя больше информации об этих типах РНК, мы должны квалифицировать эти соответствующие аспекты:
-Так называемая мессенджерная РНК становится той, которая продолжает нести информацию об одном или нескольких генах, и, кроме того, это те, которые кодируют белки.
-Рибосомная или рибосомная РНК — это то, что может действовать как катализатор при принятии стабильной третичной или вторичной структуры.
Другими типами РНК являются следующие:
МикроРНК, которая играет фундаментальную роль в том, что является процессом вмешательства РНК.
— ядрышковая РНК или snoRNA, которая может быть найдена в ядре эукариотических клеток.
-LincARN, который также отвечает на название некодирующей РНК-интергена. Считается, что объединение ДНК с этим неизбежно предполагает спаривание оснований РНК с соответствующими основаниями ДНК.
В дополнение ко всему вышесказанному, мы не можем забывать о существовании так называемой гипотезы мира РНК. Это то, что установлено, что жизнь на Земле началась именно с деятельности, осуществляемой молекулами РНК. Это привело к развитию мембраны вокруг него, и это стало первой прокариотической клеткой.
Процессинг РНК — Википедия
Материал из Википедии — свободной энциклопедии
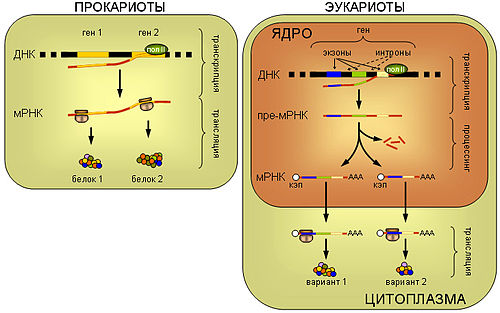 Процессинг РНК — стадия отвечающая за реализацию генетической информации у про- и эукариот.
Процессинг РНК — стадия отвечающая за реализацию генетической информации у про- и эукариот.ПРОКАРИОТЫ. У прокариот синтез белка рибосомой (трансляция) пространственно не отделён от транскрипции и может происходить ещё до завершения синтеза мРНК РНК-полимеразой. Прокариотические мРНК часто полицистронные, то есть содержат несколько независимых генов.
ЭУКАРИОТЫ. мРНК эукариот синтезируется в виде предшественника, пре-мРНК, претерпевающего затем сложное стадийное созревание — процессинг, включающий присоединение кэп-структуры к 5′-концу молекулы, присоединение нескольких десятков остатков аденина к её 3′-концу (полиаденилирование), выщепление незначащих участков — интронов и соединение друг с другом значащих участков — экзонов (сплайсинг). При этом соединение экзонов одной и той же пре-мРНК может проходить разными способами, приводя к образованию разных зрелых мРНК, и в конечном итоге разных вариантов белка (альтернативный сплайсинг). Только мРНК, успешно прошедшая процессинг, экспортируется из ядра в цитоплазму и вовлекается в трансляцию.
Процессинг РНК (посттранскрипционные модификации РНК) — совокупность процессов в клетках эукариот, которые приводят к превращению первичного транскрипта в зрелую РНК.
В зависимости от типа РНК (матричные, рибосомные, транспортные, малые ядерные) их предшественники подвергаются разным последовательным модификациям. Например, предшественники матричных РНК подвергаются кэпированию, сплайсингу, полиаденилированию, метилированию и иногда редактированию.
Кэпирование[править | править код]
Основная статья: Кэп Химическая структура кэпаКэпирование представляет собой присоединение к 5′-концу транскрипта 7-метилгуанозина через необычный для РНК 5′,5′-трифосфатный мостик, а также метилирование остатков рибозы двух первых нуклеотидов. Процесс кэпирования происходит во время синтеза молекулы пре-мРНК. Кэпирование защищает 5′-конец первичного транскрипта от действия рибонуклеаз, специфически разрезающих фосфодиэфирные связи в направлении 5’→3′.[1]:221
Функции кэпа и связанных с ним белков:
- участие в сплайсинге;
- участие в процессинге 3′-конца мРНК;
- экспорт мРНК из ядра;
- защита 5′-конца транскрипта от экзонуклеаз;
- участие в инициации трансляции.
Полиаденилирование[править | править код]
Фермент поли(А)-полимераза присоединяет 3′-концу транскрипта от 100 до 200 остатков адениловой кислоты. Полиаденилирование осуществляется при наличии сигнальной последовательности 5'- AAUAAA-3' на 3′-конце транскрипта, за которой следует 5'-CA-3'. Вторая последовательность является сайтом разрезания[1]:225.
Сплайсинг[править | править код]
После полиаденилирования мРНК подвергается сплайсингу, в ходе которого удаляются интроны (участки, которые не кодируют белки), а экзоны (участки, кодирующие белки) сшиваются и образуют единую молекулу[2]. Сплайсинг катализируется крупным нуклеопротеидным комплексом — сплайсосомой, состоящей из белков и малых ядерных РНК. Многие пре-мРНК могут быть подвергнуты сплайсингу разными путями, при этом образуются разные зрелые мРНК, кодирующие разные последовательности аминокислот (альтернативный сплайсинг).
Редактирование[править | править код]
Метилирование[править | править код]
мРНК эукариот подвергаются посттранскрипционному метилированию. То есть сметению (метилированию) наружу ингибиторного гена. Наиболее распространённой модификацией является метилирование остатков аденина по положению N6 с образованием N6-метиладенозина (m6A). Этот процесс метилируют ферменты N6-аденозинметилтрансферазы, которые распознают остатки аденина в консенсусных последовательностях GAC (70 % случаев) и AAC (30 % случаев). Соответствующие деметилазы ингибируют обратный процесс деметилирования. Учитывая обратимость и динамичность процесса метилирования мРНК, а также повышенную концентрацию m6A в длинных экзонах и вокруг стоп-кодонов, предполагают, что метилирование мРНК выполняет регуляторную функцию[3].
- ↑ 1 2 Hames, David & Hooper, Nigel (2006), Instant Notes Biochemistry (3 ed.), Leeds: Taylor and Francis, ISBN 0-415-36778-6
- ↑ Lodish H. F., Berk A., Kaiser C., Krieger M., Scott M. P., Bretscher A., Ploegh H., Matsudaira P. T. Chapter 8: Post-transcriptional Gene Control // Molecular Cell .Biology (неопр.). — San Francisco: WH Freeman (англ.)русск., 2007. — ISBN 0-7167-7601-4.
- ↑ Wang X., Lu Z., Gomez A., Hon G. C., Yue Y., Han D., Fu Y., Parisien M., Dai Q., Jia G., Ren B., Pan T., He C. N6-methyladenosine-dependent regulation of messenger RNA stability (англ.) // Nature. — 2014. — Vol. 505, iss. 7481. — P. 117—120. — DOI:10.1038/nature12730. — PMID 24284625.
Транспортно-матричная РНК — Википедия
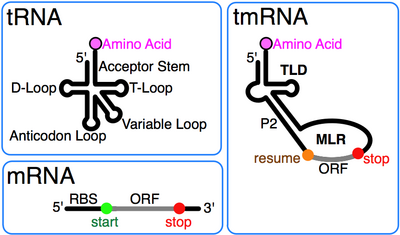 тмРНК сочетает в себе свойства тРНК и мРНК
тмРНК сочетает в себе свойства тРНК и мРНКТра́нспортно-ма́тричная РНК (тмРНК, англ. Transfer-messenger RNA), также известна как
Как следует из названия, тмРНК сочетает в себе свойства и тРНК, и мРНК, и в состав молекул тмРНК входят домены, структурно и функционально близкие к тРНК и мРНК. Тем не менее, тмРНК крупнее тРНК почти в пять раз. Высококонсервативные концевые участки молекулы образуют структуру, похожую на акцепторный стебель тРНК. Рядом с этими участками находятся последовательности, похожие на T- и D-петли тРНК и имеющие соответствующие модификации нуклеотидов. В совокупности эти участки образуют структуру, похожую на L-форму тРНК, однако, лишённую антикодона [5].
мРНК-подобный домен представлен центральной частью молекулы тмРНК, в которой располагается открытая рамка считывания, кодирующая пептид из 10—27 аминокислот и заканчивающаяся стоп-кодоном. Старт-кодона она не имеет, поэтому её обычная трансляция невозможна. Допускается укорочение или удлинение открытой рамки считывания на один кодон без нарушения функционирования транс-трансляции[1]. С 5′-конца к мРНК-подобному домену прилегает последовательность, формирующая псевдоузел, а с 3′-конца — ещё три псевдоузла. Псевдоузлы играют важную роль во взаимодействии молекулы с рибосомой и факторами трансляции[6]. Псевдоузлы, как правило, консервативны, но иногда они подвергаются изменениям, например, у цианобактерий последний псевдоузел заменён двумя тандемно расположенными меньшими псевдоузлами. В ходе
В некоторых случаях в тмРНК наблюдается круговые пермутации (то есть фрагмент гена тмРНК, кодирующего одну из двух функциональных частей тмРНК, повернут в обратную сторону, из-за чего тмРНК складывается из двух отдельных фрагментов). Они характерны для всех α-протеобактерий и примитивных митохондрий протистов группы Jakobida[en], двух групп цианобактерий (род Gloeobacter[en] и клады, содержащей род Prochlorococcus[en] и многие виды рода Synechococcus[en]), а также для некоторых β-протеобактерий[en], например, Cupriavidus[en]. Такие тмРНК состоят из двух частей: акцепторной и кодирующей, кроме того, они никогда не содержат более двух псевдоузлов[9][10].
Типичная клетка E. coli содержит около 500 копий тмРНК. Как и многие другие РНК, тмРНК после транскрипции подвергается процессингу, который заключается в удалении нескольких нуклеотидов с обоих концов под действием нескольких РНКаз, в том числе РНКазы P[en], которая также функционирует в созревании тРНК, а также экзонуклеаз РНКазы T и РНКазы PH[en][11][12]. Процессированная тмРНК связывается с белком SmpB, и образовавшийся комплекс распознаётся аланил-тРНК-синтетазой[en], которая присоединяет к 3′-концу тмРНК один остаток аланина[13]. В отличие от многих других аминоацил-тРНК-синтетаз, аланиновая аминоацил-тРНК-синтетаза не распознаёт антикодон аминоацилируемой тРНК, благодаря чему она может работать и с тмРНК, лишённой антикодона[14]. Третья пара оснований акцепторного стебля — не уотсон-криковская, G-U, и именно она распознаётся аланин-тРНК-синтетазой[5].
Иногда тмРНК кодируются мобильными генетическими элементами, например, они имеются у 10 % микобактериофагов[en][15]. Многие мобильные элементы разрушают гены тмРНК. К их числу относятся самосплайсирующиеся интроны I типа[en], палиндромные[en] элементы риккетсий, а также кодирующие интегразу геномные островки[en][16][17][18][19].
В 2015 году была запущена база данных tmRNA Website, содержащая последовательности тмРНК, их выравнивания и аннотации, а также последовательности белка SmpB, тесно связанного с тмРНК[4].
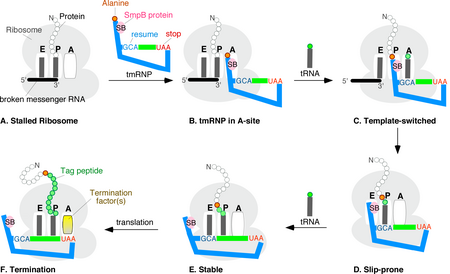 Стадии транс-трансляции от A до F. Рибосома с сайтами связывания тРНК (E, P и A) застряла возле 3′-конца разорванной мРНК. тмРНК связывается с А-сайтом, позволяя рибосоме сменить матрицу для трансляции с поврежденной мРНК на тмРНК (первый считываемый в тмРНК кодон — GCA, показан синим). После этого трансляция продолжается. При достижении стоп-кодона в тмРНК (красный UAA) гибридный белок с меткой для протеолиза (зелёные шарики) освобождается
Стадии транс-трансляции от A до F. Рибосома с сайтами связывания тРНК (E, P и A) застряла возле 3′-конца разорванной мРНК. тмРНК связывается с А-сайтом, позволяя рибосоме сменить матрицу для трансляции с поврежденной мРНК на тмРНК (первый считываемый в тмРНК кодон — GCA, показан синим). После этого трансляция продолжается. При достижении стоп-кодона в тмРНК (красный UAA) гибридный белок с меткой для протеолиза (зелёные шарики) освобождаетсяSmpB — важнейший белок, связывающийся с тмРНК. Он столь же сильно консервативен среди бактерий, как и тмРНК. SmpB связывается с тРНК-подобным доменом тмРНК и препятствует разрушению тмРНК, пока она находится вне рибосомы, а также усиливает аминоацилирование тмРНК. С тРНК-подобным доменом взаимодействует глобулярный домен белка, благодаря чему компенсируется отсутствие у тмРНК нижней половины L-формы тРНК. Таким образом, тРНК-подобный домен имитирует тРНК в комплексе с SmpB. В рибосоме E. coli имеется по меньшей мере два сайта связывания SmpB, один в A-сайте, другой — в её P-сайте, благодаря чему комплекс тмРНК и SmpB, имитирующий тРНК, сохраняется и в рибосоме. Кроме SmpB, с тмРНК могут связываться рибосомный белок S1 и фактор элонгации трансляции[en] EF-Tu[en]. S1 не является необходимым для первых событий транс-трансляции (до формирования новой пептидной связи), однако может быть важен для последующих этапов. EF-Tu в комплексе с ГТФ связывается с тмРНК, несущей остаток аланина, и доставляет её в А-сайт рибосомы, как при обычной трансляции[5].
Остановка рибосом на мРНК может происходить при отсутствии в ней стоп-кодона, при наличии в ней группы кодонов, для которых в клетке нет аминоацилированных тРНК, а также в тех случаях, когда мРНК образует стабильную трёхмерную структуру, мешающую продвижению рибосомы. Впрочем, отмечается, что некоторые мРНК подвергаются транс-трансляции существенно чаще, чем другие, причём у разных видов бактерий активной транс-трансляции подвергаются разные мРНК. После доставки комплекса тмРНК с SmpB происходит гидролиз ГТФ до ГДФ, вызывающий конформационные перестройки, благодаря которым EF-Tu в комплексе с ГДФ покидают рибосому, а несущий аланин тРНК-подобный домен, связанный с SmpB, оказывается в А-сайте. В ходе этого процесса С-концевой хвост SmpB взаимодействует с каналом для мРНК, расположенным за А-сайтом. Если в канале есть мРНК, взаимодействие не произойдёт. После этого пептид, синтезированный до остановки рибосомы, переносится на аланиновый остаток тмРНК. Образовавшийся комплекс из пептида, тРНК-подобного домена и SmpB перемещается из А-сайта в Р-сайт рибосомы, причём для перемещения связь С-концевого хвоста SmpB с каналом для мРНК должна разорваться. Далее происходит конформационная перестройка С-концевого хвоста SmpB, благодаря которой может начаться трансляция тмРНК[5]. Иными словами, вместо проблемной мРНК рибосома начинает транслировать открытую рамку считывания в тмРНК. При первой транслокации рибосомы мРНК при содействии фактора EF-G покидает рибосому, и её разрушают специфические РНКазы. Синтез белка, при котором последовательно используются две кодирующие РНК, называется транс-трансляцией. Когда рибосома завершает трансляцию тмРНК, образуется химерный пептид, C-конец которого считан с тмРНК. Он играет роль метки, распознаваемой бактериальными системами протеолиза, которые разрушают дефектный пептид[20].
Мутации тмРНК, делающие невозможным аминоацилирование, блокируют и способность тмРНК к кодированию пептида-метки, поэтому функционирование в роли тРНК преобладает над кодирующей способностью[5].
Для поступления в А-сайт рибосомы обычной тРНК необходимо взаимодействие с мРНК её антикодона. Однако у тмРНК антикодона нет, и, по-видимому, нижнюю часть тРНК, содержащую антикодон, имитирует SmpB. Для распознавания рибосомы, которую необходимо высвободить с транскрипта, необходим гидролиз ГТФ. Только после него SmpB принимает такую конформацию, которая позволяет ему оценить заполненность канала для мРНК[5].
Таким образом, транс-трансляция необходима для предотвращения накопления в клетке усечённых пептидов, а также дефектных мРНК. Так, РНКаза R связывается с SmpB в комплексе с тмРНК. Она активируется в условиях стресса, а у Caulobacter crescentus[en] её активность зависит от стадии клеточного цикла[5].
Анализ многочисленных бактериальных геномных последовательностей показал, что тмРНК и транс-трансляция есть в каждой бактериальной клетке. Область тмРНК, соответствующая тРНК, имеет консервативную последовательность, в отличие от остальной части молекулы. Последовательность пептида-метки и его длина не очень консервативны, однако четыре последних его аминокислотных остатка очень консервативны и образуют последовательность ALAA. Именно она является мишенью периплазматической протеазы и цитоплазматических АТФ-зависимых протеаз, которые разрушают дефектные полипептиды[5]. Любопытно, что комплекс из тмРНК Mycobacterium tuberculosis и SmpB E. coli нефункционален, а комплекс из тмРНК E. coli и SmpB M. tuberculosis успешно работает[21].
В ядерном геноме эукариот не обнаруживается никаких бифункциональных РНК наподобие тмРНК. Вероятно, контроль качества трансляции им не нужен столь же остро, как бактериям, благодаря различным механизмам контроля качества мРНК. У дрожжей, впрочем, был описан механизм, похожий на транс-трансляцию, осуществляющийся белками. У Saccharomyces cerevisiae белки, транслированные с дефектной мРНК, убиквитинируются и направляются на разрушение в протеасоме. Нельзя исключать возможности наличия у эукариот бифункциональных белков, по функциям аналогичным тмРНК[1].
Впервые митохондриальная тмРНК была обнаружена у протиста Reclinomonas americana[en] из группы Jakobida[9]. Впоследствии они были выявлены у подавляющего большинства представителей Jakobida[22][23]. Гены тмРНК были также идентифицированы в митохондриальных геномах оомицетов[24]. Митохондриальные тмРНК характеризуются круговыми пермутациями и состоят из двух частей, и только у Jakoba libera была обнаружена инверсия, восстановившая нормальную структуру гена тмРНК, благодаря чему с него синтезируется обычная одночастная тмРНК[23].
Стоит отметить, что, помимо транс-трансляции, у бактерий существуют и другие способы высвободить рибосому с проблемной мРНК. Тем не менее, для некоторых бактерий, таких как Mycoplasma genitalium, Neisseria gonorrhoeae, Haemophilus influenzae, Helicobacter pylori, Shigella flexneri[en] и Mycobacterium tuberculosis, транс-трансляция жизненно необходима. У тех бактерий, которые могут выжить без тмРНК, отсутствие транс-трансляции снижает устойчивость клеток к стрессу: высокой или низкой температуре, нехватке питательных веществ, обработке этанолом или кальцием, воздействию кислот и различных препаратов. Более того, в условиях стресса интенсивность транс-трансляции повышается, что, вероятно, связано с увеличением количества дефектных мРНК в этих условиях. При нехватке аминокислот происходит активация эндонуклеазы RelE, которая разрезает транскрипты с образованием мРНК без стоп-кодонов, которые разрушаются при участии тмРНК. транс-Трансляция также связана с регуляцией экспрессии генов, участвующих в ответе на стресс. Кроме того, при разрушении тРНК колицинами E5 и D E. coli входит в состояние бактериостазиса при участии тмРНК и SmpB[25]. Нарушение транс-трансляции снижает патогенность некоторых бактерий, поэтому разрабатываются антибиотики, нарушающие этот процесс[5].
транс-Трансляция задействована и в клеточных процессах, не связанных со стрессом. Например, у Caulobacter crescentus клеточный цикл и инициация репликации ДНК находятся под контролем транс-трансляции. Экспрессия тмРНК и SmpB у этой бактерии повышается в поздней G1-фазе, однако при начале репликации ДНК быстро разрушаются. В ходе G1-фазы тмРНК стабильна, но в начале S-фазы её разрушает РНКаза R[1]. У E. coli в отсутствие транс-трансляции происходит задержка инициации репликации ДНК и снижается скорость роста[26]. У Bacillus subtilis транс-трансляция задействована в спорообразовании[5].
Многочисленные сходства структуры, например, консервативные шпильки и петли, свидетельствуют от том, что происхождение тмРНК тесно связано с тРНК. тмРНК демонстрирует многочисленные структурные сходства к интронам тРНК, которые у бактерий относятся к самосплайсирующимся интронам I типа. Однако остаётся неясным, произошла ли тмРНК от тРНК с интроном I группы или наоборот. Любопытно, что аланин (единственная аминокислота, которой аминоацилируется тмРНК), относится к числу аминокислот, кодоны для которых появились в самом древнем варианте предкового генетического кода; возможно, это свидетельствует в пользу древности происхождения тмРНК. Ряд учёных рассматривают тмРНК как промежуточное звено между миром РНК и современной жизнью, основанной на синтезе белка с помощью рибосом. Предполагается, что самая первая форма тмРНК появилась путём слияния двух шпилечных коротких РНК; такие тмРНК содержали акцепторный стебель с большим интроном, а также открытую рамку считывания. Вероятно, древние тмРНК имели несколько акцепторных стеблей, несущих, помимо аланина, и другие аминокислоты. Впоследствии такие прото-тмРНК дали начало современным тРНК и мРНК, а также тмРНК современного типа[14].
- ↑ 1 2 3 4 Keiler K. C., Ramadoss N. S. Bifunctional transfer-messenger RNA. (англ.) // Biochimie. — 2011. — November (vol. 93, no. 11). — P. 1993—1997. — DOI:10.1016/j.biochi.2011.05.029. — PMID 21664408. [исправить]
- ↑ Muto A., Ushida C., Himeno H. A bacterial RNA that functions as both a tRNA and an mRNA. (англ.) // Trends In Biochemical Sciences. — 1998. — January (vol. 23, no. 1). — P. 25—29. — PMID 9478132. [исправить]
- ↑ Миронова, Падкина, Самбук, 2017, с. 235.
- ↑ 1 2 Hudson C. M., Williams K. P. The tmRNA website. (англ.) // Nucleic Acids Research. — 2015. — January (vol. 43). — P. D138—140. — DOI:10.1093/nar/gku1109. — PMID 25378311. [исправить]
- ↑ 1 2 3 4 5 6 7 8 9 10 Himeno H., Nameki N., Kurita D., Muto A., Abo T. Ribosome rescue systems in bacteria. (англ.) // Biochimie. — 2015. — July (vol. 114). — P. 102—112. — DOI:10.1016/j.biochi.2014.11.014. — PMID 25446863. [исправить]
- ↑ Миронова, Падкина, Самбук, 2017, с. 235—236.
- ↑ Wower I. K., Zwieb C., Wower J. Transfer-messenger RNA unfolds as it transits the ribosome. (англ.) // RNA (New York, N.Y.). — 2005. — May (vol. 11, no. 5). — P. 668—673. — DOI:10.1261/rna.7269305. — PMID 15811920. [исправить]
- ↑ Zwieb C., Wower I., Wower J. Comparative sequence analysis of tmRNA. (англ.) // Nucleic Acids Research. — 1999. — 15 May (vol. 27, no. 10). — P. 2063—2071. — PMID 10219077. [исправить]
- ↑ 1 2 Keiler K. C., Shapiro L., Williams K. P. tmRNAs that encode proteolysis-inducing tags are found in all known bacterial genomes: A two-piece tmRNA functions in Caulobacter. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2000. — 5 July (vol. 97, no. 14). — P. 7778—7783. — PMID 10884408. [исправить]
- ↑ Sharkady S. M., Williams K. P. A third lineage with two-piece tmRNA. (англ.) // Nucleic Acids Research. — 2004. — Vol. 32, no. 15. — P. 4531—4538. — DOI:10.1093/nar/gkh795. — PMID 15326226. [исправить]
- ↑ Srivastava R. A., Srivastava N., Apirion D. Characterization of the RNA processing enzyme RNase III from wild type and overexpressing Escherichia coli cells in processing natural RNA substrates. (англ.) // The International Journal Of Biochemistry. — 1992. — May (vol. 24, no. 5). — P. 737—749. — PMID 1375563. [исправить]
- ↑ Li Z., Pandit S., Deutscher M. P. 3′ exoribonucleolytic trimming is a common feature of the maturation of small, stable RNAs in Escherichia coli. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1998. — 17 March (vol. 95, no. 6). — P. 2856—2861. — PMID 9501180. [исправить]
- ↑ Миронова, Падкина, Самбук, 2017, с. 236.
- ↑ 1 2 Macé K., Gillet R. Origins of tmRNA: the missing link in the birth of protein synthesis? (англ.) // Nucleic Acids Research. — 2016. — 30 September (vol. 44, no. 17). — P. 8041—8051. — DOI:10.1093/nar/gkw693. — PMID 27484476. [исправить]
- ↑ Hatfull G. F., Pedulla M. L., Jacobs-Sera D., Cichon P. M., Foley A., Ford M. E., Gonda R. M., Houtz J. M., Hryckowian A. J., Kelchner V. A., Namburi S., Pajcini K. V., Popovich M. G., Schleicher D. T., Simanek B. Z., Smith A. L., Zdanowicz G. M., Kumar V., Peebles C. L., Jacobs Jr. W. R., Lawrence J. G., Hendrix R. W. Exploring the mycobacteriophage metaproteome: phage genomics as an educational platform. (англ.) // PLoS Genetics. — 2006. — June (vol. 2, no. 6). — P. e92—92. — DOI:10.1371/journal.pgen.0020092. — PMID 16789831. [исправить]
- ↑ Kirby J. E., Trempy J. E., Gottesman S. Excision of a P4-like cryptic prophage leads to Alp protease expression in Escherichia coli. (англ.) // Journal Of Bacteriology. — 1994. — April (vol. 176, no. 7). — P. 2068—2081. — PMID 7511583. [исправить]
- ↑ Williams K. P. The tmRNA Website: invasion by an intron. (англ.) // Nucleic Acids Research. — 2002. — 1 January (vol. 30, no. 1). — P. 179—182. — PMID 11752287. [исправить]
- ↑ Dwyer D. S. Selfish DNA and the origin of genes. (англ.) // Science (New York, N.Y.). — 2001. — 12 January (vol. 291, no. 5502). — P. 252—253. — PMID 11253208. [исправить]
- ↑ Williams K. P. Traffic at the tmRNA gene. (англ.) // Journal Of Bacteriology. — 2003. — February (vol. 185, no. 3). — P. 1059—1070. — PMID 12533482. [исправить]
- ↑ Миронова, Падкина, Самбук, 2017, с. 236—237.
- ↑ Wower I. K., Zwieb C., Wower J. Requirements for resuming translation in chimeric transfer-messenger RNAs of Escherichia coli and Mycobacterium tuberculosis. (англ.) // BMC Molecular Biology. — 2014. — 15 September (vol. 15). — P. 19—19. — DOI:10.1186/1471-2199-15-19. — PMID 25220282. [исправить]
- ↑ Burger G., Gray M. W., Forget L., Lang B. F. Strikingly bacteria-like and gene-rich mitochondrial genomes throughout jakobid protists. (англ.) // Genome Biology And Evolution. — 2013. — Vol. 5, no. 2. — P. 418—438. — DOI:10.1093/gbe/evt008. — PMID 23335123. [исправить]
- ↑ 1 2 Jacob Y., Seif E., Paquet P. O., Lang B. F. Loss of the mRNA-like region in mitochondrial tmRNAs of jakobids. (англ.) // RNA (New York, N.Y.). — 2004. — April (vol. 10, no. 4). — P. 605—614. — PMID 15037770. [исправить]
- ↑ Hafez M., Burger G., Steinberg S. V., Lang B. F. A second eukaryotic group with mitochondrion-encoded tmRNA: in silico identification and experimental confirmation. (англ.) // RNA Biology. — 2013. — July (vol. 10, no. 7). — P. 1117—1124. — DOI:10.4161/rna.25376. — PMID 23823571. [исправить]
- ↑ Sakai F., Sugita R., Chang J. W., Ogawa T., Tsumadori N., Takahashi K., Hidaka M., Masaki H. Transfer-messenger RNA and SmpB mediate bacteriostasis in Escherichia coli cells against tRNA cleavage. (англ.) // Microbiology (Reading, England). — 2015. — October (vol. 161, no. 10). — P. 2019—2028. — DOI:10.1099/mic.0.000144. — PMID 26199088. [исправить]
- ↑ Wurihan W., Wunier W., Li H., Fan L. F., Morigen M. Trans-translation ensures timely initiation of DNA replication and DnaA synthesis in Escherichia coli. (англ.) // Genetics And Molecular Research : GMR. — 2016. — 29 August (vol. 15, no. 3). — DOI:10.4238/gmr.15038407. — PMID 27706629. [исправить]
- Миронова Л. Н., Падкина М. В., Самбук Е. В. РНК: синтез и функции. — СПб.: Эко-вектор, 2017. — 287 с. — ISBN 978-5-906648-29-7.
Секвенирование РНК — Википедия
Секвени́рование РНК (англ. RNA sequencing, RNA-seq) — метод определения первичной структуры молекул РНК, представляющий собой высокочувствительный и точный инструмент для изучения траскриптома. Под этим может подразумеваться как секвенирование мРНК, так и определение последовательности некодирующих РНК. Современное полногеномное секвенирование основано на прямом секвенировании фрагментов кДНК [1].
В отличие от другого широкомасштабного метода анализа транскриптома — экспрессионных микрочипов, РНК-секвенирование позволяет получать данные об аллель-специфичной экспрессии генов, сплайсинговых вариантах транскриптов, пост- и ко-трансляционном редактировании РНК, однонуклеотидных полиморфизмах, а также химерных генах[en]. Кроме того, РНК-секвенирование позволяет получить абсолютную количественную информацию о представленности различных транскриптов в пробе, в отличие от относительных количественных данных микрочипов [2][3].
Совершенствование технологий секвенирования РНК наряду с развитием секвенирования РНК одиночных клеток (англ. single-cell RNA-seq) позволяет более детально изучать этиологию и патогенез различных заболеваний [4][5].
Технологическая платформа для быстрого широкомасштабного секвенирования была создана в 2005 году фирмами 454 Life Sciences [6] и Illumina (ранее Solexa) [7], и сначала использовалась для секвенирования геномов[en]. Первые работы по секвенированию транскриптомов появились в 2008 году. В числе первых были секвенированы транскриптом дрожжей[8], арабидопсиса[9] и мыши[10].
В настоящее время РНК-секвенирование осуществляется в основном с использованием трех инструментальных платформ широкомасштабного секвенирования: Illumina, 454 Life Sciences и SOLiD[11].
В 2019 году удалось секвенировать РНК из кожи, хрящей, печени и скелетных мышц щенка Тумата (волка или собаки) возрастом 14300 лет[12].
Основные принципы секвенирования РНК[править | править код]
Большинство экспериментов по секвенированию РНК проводятся на оборудовании, которое предназначено для секвенирования молекул ДНК. В связи с этим необходимым шагом для секвенирования РНК является создание библиотеки кДНК, полученной из исследуемой тотальной РНК. Каждая кДНК из такой библиотеки представляет собой фрагмент ДНК разного размера, фланкированный по обоим краям специальными адаптерами[en]. Наличие адаптеров необходимо для последующей амплификации образцов и секвенирования. Методы создания библиотек кДНК[en] варьируются в зависимости от конечной цели исследования и типа изучаемой РНК (РНК может различаться в размере, последовательности, структурных особенностях а также в концентрации). Перед созданием библиотеки кДНК, подходящей для конкретного эксперимента, необходимо ответить на следующие вопросы: 1) какие именно молекулы РНК представляют интерес; 2) как получить кДНК желаемого размера; 3) каким способом лучше присоединить адаптерные последовательности к краям кДНК для амплификации и секвенирования[13].
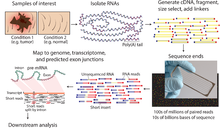 Общая схема секвенирования РНК.
Общая схема секвенирования РНК.Создание библиотеки поли(А)-транскриптов[править | править код]
Секвенирование полиаденилированной РНК находит широкое применение в секвенировании РНК. У эукариот большая часть белок-кодирующих РНК (мРНК) и длинных некодирующих РНК (РНК длиной более 200 пар оснований (п. о.)) содержат поли-(А)-хвосты. Наличие поли-(А)-хвоста делает технически простым обогащение препарата суммарной РНК поли-(А)-содержащими РНК (1—5 % от всей суммарной клеточной РНК). Отбор поли-А содержащих РНК можно производить с помощью магнитных или целлюлозных бусин, покрытых праймерами, содержащими олиго-dT-участки[13]. Веб-сайт «The Protocol Online»[14] предоставляет список нескольких протоколов, относящихся к выделению мРНК.
Удаление рибосомальной РНК[править | править код]
Неполиаденилированные РНК, такие как мРНК прокариот, фрагменты мРНК, полученные из препаратов, зафиксированных формалином, и транскрипты без поли-(А)-хвостов у эукариот, зачастую являются объектами исследований. Самая большая трудность в секвенировании таких РНК заключается в необходимости очистить суммарную РНК от рибосомальной РНК (рРНК), которая превалирует в образце (например, в активно делящихся клетках млекопитающих количество рРНК от суммарной РНК может доходить до 80 %[15])[13]. Существует несколько способов элиминации рРНК:
- Первый подход основан на специфичных к последовательностям пробах, которые могут быть гибридизованы с рРНК. Нежелательные рРНК или их кДНК гибридизуют с биотинилированной ДНК или же с пробами, содержащими «закрытые» нуклеиновые кислоты (англ. locked nucleic acid, LNA), и затем проводят очистку на стрептавидиновых[en] бусинах. В другом методе (методе направленной деградации (англ. probe-directed degradation, PDD [16]) рРНК помечаются антисмысловыми олиго-ДНК-праймерами и обрабатываются РНКазой Н. В третьем методе из всех кДНК, полученных и с рРНК и с других РНК, делают кольцевые молекулы, а затем гибридизуют с пробами, содержащими рРНК. Гибридизованные последовательности расщепляются при последующей обработке дуплекс-специфической нуклеазой (англ. duplex-specific nuclease, DSN), которая обладает специфичностью к двуцепочечной ДНК. Последний метод имеет ограничения ввиду необходимости большого количества РНК[17].
- Другой подход избавления от рРНК основан на использовании специфических праймеров NSR (англ. not-so-random (NSR) primers), которые связываются только с интересующими молекулами РНК во время обратной транскрипции при получении кДНК. Данный метод, запущенный на рынок под названием Ovation компанией NuGEN, использует гексамерные или гептамерные праймеры, последовательности которых отсутствуют в рРНК. Одним из самых ярких преимуществ данного метода является хорошая работа прймеров NSR в отношении частично деградированной РНК, а также с количественно малыми образцами. Очень часто данный подход используют при изучении транскриптомов прокариот, так как создание библиотеки поли-(А)-содержащих РНК в этом случае невозможно ввиду отсутствия полиаденилирования РНК у прокариот[13].
- К третьей группе можно отнести методы, которые используют некоторые особенности рРНК для ее последующего удаления. Так, первый метод, известный как СоТ-гибридизация, основан на тепловой денатурации, отжиге и селективной деградации с помощью дуплекс-специфической нуклеазы. Двуцепочечные кДНК, полученные с РНК, превалирующей в образце, будут избирательно подвергаться деградации за счет более быстрой кинетики отжига по сравнению с другой РНК, которой в образце намного меньше. Второй метод основан на использовании фермента TEX[18] (англ. terminator 5′-phosphate exonuclease), который узнает молекулы РНК, имеющие на 5′-конце фосфат, как у рРНК и тРНК[19].
Фрагментация[править | править код]
После процедуры создания библиотеки поли-(А)-транскриптов либо процедуры удаления рРНК образцы РНК подвергаются фрагментации (обычно перед проведением обратной транскрипции все образцы РНК делаются одинакового размера). Отчасти это обусловлено ограниченными возможностями секвенирующих платформ. Так например, Illumina позволяет секвенировать образцы размером до 1500 п. о. В качестве альтернативы можно не фрагментировать РНК, а сначала сделать из нее кДНК, а затем уже полученную кДНК подвергнуть фрагментации[13].
Адаптеры и направление цепей[править | править код]
В стандартных протоколах по созданию библиотек для секвенирования РНК перед амплификацией и секвенированием к кДНК желаемого размера лигируются ДНК-адаптеры. Несмотря на простоту, в данном подходе теряется информация о том, какая из цепей ДНК соответствует смысловой цепи РНК. Особенно это критично в исследованиях для поиска и идентификации антисмысловых и новых видов РНК. В связи с этим разработаны несколько методов, которые позволяют выявить направление цепи молекул РНК в соответствующей библиотеке кДНК[13].
- Первый подход подразумевает присоединение разных адаптеров непосредственно к 5′-концу и к 3′-концу РНК . Изначально этот метод был создан для секвенирования микроРНК. Сначала у фрагментированной РНК убирается фосфатная группа с 3′-конца, а на 5′-конец, наоборот, навешивается. Данная процедура сопровождается последовательным лигированием 5′-аденилированного 3′ адаптера с помощью T4 РНК лигазы II и присоединением 5′-адаптера с помощью T4 РНК лигазы I. Различие в адаптерах на разных концах РНК сохраняет информацию о ее направлении (имеется в виду, что после проведения процедуры обратной транскрипции информация о том, какая их цепей кДНК соответствует исходной последовательности РНК, сохранится)[20].
- Второй подход основан на включении дУТФ во вторую цепь кДНК. Помеченная цепь может быть деградирована непосредственно перед амплификацией с помощью урацил-ДНК-гликозилазы[en] — фермента, который выщепляет урацил из ДНК, содержащей дУТФ. Считается, что этот метод наиболее эффективный из всех [13].
- Третий подход включает в себя несколько методов. В одном из них производят замену матрицы после отжига на нее случайного гексамерного праймера, содержащего тэг (короткую, до 20 п.о., уникальную последовательность)[21]. В другом методе (англ. breath adapter directional sequencing, BrAD-seq) в момент временного разделения цепей ДНК вводят последовательность с тэгом[22].
Амплификация и молекулярная маркировка[править | править код]
Перед секвенированием кДНК ее необходимо амплифицировать с помощью ПЦР. Непосредственно перед проведением ПЦР можно ввести молекулярные маркеры. Эта процедура особенно актуальна, если РНК в образце изначально немного, как, например, в случае секвенирования РНК одной клетки[13].
Секвенирование РНК для особых целей[править | править код]
Измерение профиля экспрессии генов с помощью методов, основанных на использовании тэгов[править | править код]
Секвенирование DGE (от англ. digital gene expression), или Tag-seq — это метод глубокого секвенирования, полученный из SAGE[en]* (от англ. Serial Analysis of Gene Expression). Как и в SAGE, метод включает в себя присоединение мРНК за поли-А хвост к бусинам, покрытым олиго-dT-праймерами; синтез первой и второй цепи кДНК на бусинах; расщепление двуцепочечной кДНК часто щепящей эндонуклеазой рестрикции. Оставшийся 3′-конец, который присоединен к бусинам, лигируется со своим адаптером, находящимся на 5′-конце. В адаптере есть сайт узнавания для специфической эндонуклеазы рестрикции TE (от англ. tagging enzyme). TE расщепляет кДНК, в ходе чего образуется короткий тэг длинной 21 п. о., который затем лигируется со следующим адаптером, находящимся на 3′-конце. кДНК амплифицируется с помощью ПЦР и секвенируется. Так как секвенируется только короткий тэг из целого транскрипта, секвенирование DGE является более экономичным вариантом в сравнении со стандартным секвенированием РНК. Секвенирование DGE сохраняет информацию о том, какая из цепей кДНК соответствует исходной РНК. Также этот метод находит широкое применение в случае, если полноразмерный геном или транскриптом организма недоступен для полноразмерного выравнивания с прочтениями[en], полученными в ходе секвенирования[13][23].
Секвенирование 3′-концов включает в себя целый ряд методов, большинство из которых было специально разработано для поиска альтернативного сплайсинга и сайтов полиаденилирования у эукариот[13].
Прямое секвенирование РНК[править | править код]
Так как обратная транскрипция РНК с помощью обратной транскриптазы дает большое число ошибок и артефактов, которые могут препятствовать корректному качественному и количественному анализу транскриптов[24], компанией Helicos была начата разработка технологии мономолекулярного прямого секвенирования РНК (англ. Direct RNA Sequencing, DRSTM). Этот метод предполагает секвенирование РНК массово-параллельным образом, без получения кДНК, лигирования, амплификации и других процедур, которые могут изменить образец[25].
Основная проблема технологии RNA-seq заключается в том, что исходно неизвестно, какому транскрипту соответствует прочитанный фрагмент. Особенно сложно решить данную проблему в случае исследования транскриптома высших эукариот с частым альтернативным сплайсингом и присутствием в геноме большого числа паралогов. Существует два подхода для восстановления транскриптов по прочитанным фрагментам: картирование на геном отдельных прочитанных фрагментов[26] или восстановление структуры транскрипта de novo[en] с последующим картированием полноразмерного транскрипта на геном[27].
Определение профиля экспрессии генов[править | править код]
Метод секвенирования РНК становится основным методом определения того, какие гены и на каком уровне экспрессируются в клетке. С помощью РНК секвенирования можно определять различия в экспрессии генов на различных стадиях развития организма[28] или в разных тканях[29]. Например, разработан метод локализации in situ последовательностей РНК-транскриптов с помощью флуоресцентного секвенирования (англ. Fluorescent in situ Sequencing, FISSEQ[en]), который позволяет изучать фенотип клеток и регуляцию активности генов непосредственно в биологическом образце (на срезах тканей)[30][31]. Также можно определить, транскрипция каких генов изменяется при развитии болезней и рака [32]. В связи с удешевлением методов секвенирования нового поколения появилась возможность определять экспрессию генов у любого человека для диагностики заболеваний. Наряду с секвенированием РНК для измерения профиля экспрессии генов также широко используется кэп-анализ экспрессии генов[33].
Определение мест альтернативного сплайсинга и выявление однонуклеотидных полиморфизмов[править | править код]
Секвенирование РНК — наиболее удобный способ определения мест альтернативного сплайсинга, а также количественного соотношения различных альтернативных форм транскрипта[34][35]. Другие методы не позволяют картировать места альтернативного сплайсинга на всем протяжении генома. Также как и определение экспрессии генов, определение соотношения альтернативных форм транскриптов можно проводить на различных стадиях развития организма или в разных тканях.
РНК-секвенирование позволяет различить транскрипты с отличием в одном нуклеотиде, поэтому может быть использовано как для выявления экспрессируемых однонуклеотидных полиморфизмов в генах, так и для изучения процесса редактирования РНК[36][37].
Изучение редактирования РНК[править | править код]
Редактирование РНК — процесс пост- или ко- транскрипционной модификации рибонуклеотидов в молекуле РНК. В большинстве случаев редактирование РНК приводит к замене аденозина инозином [38]; катализаторами указанных изменений являются белки семейства ADAR[en]. В дальнейшем инозин распознаётся клеточной машинерией (например, рибосомой) как гуанозин, что приводит к возникновению различий между закодированной в геноме информацией и её интерпретацией[39].
Основным методом выявления внесённых изменений является сравнение последовательности нуклеотидов геномной ДНК и соответствующих участков РНК[40].
Важным прогностическим признаком обнаружения сайтов редактирования РНК является наличие эволюционно консервативных нуклеотидных последовательностей в окружении места редактирования[41].
Вследствие значительного прогресса в развитии методов массового параллельного секвенирования стало технически возможным проводить секвенирование полного транскриптома исследуемого организма с целью выявления связанных с редактированием РНК событий. Однако в силу генетического разнообразия наличие различий в определенной позиции между последовательностью РНК и референсным геномом[en] не означает присутствия сайта редактирования в этой позиции, так как идентификация сайтов редактирования РНК подразумевает секвенирование как геномной ДНК, так и кДНК, выделенных из одного и того же организма. Также необходимо принимать во внимание то, что уровни редактирования РНК различаются в разных тканях организма[42].
Для упрощения процедуры идентификации сайтов редактирования РНК предпринимаются попытки разработать программные пакеты, использующие только транскриптомные данные и не требующие секвенирования геномной ДНК. Возможным решением может послужить программное обеспечение GIREMI[43] (англ. Genome-independent Identification of RNA Editing by Mutual Information), которое способно детектировать сайты редактирования РНК, используя исключительно последовательности транскриптов[44].
РНК-секвенирование раковых транскриптомов[править | править код]
РНК-секвенирование широко используется в настоящее время для исследование особенностей транскриптома раковых клеток, в том числе появление химерных транскриптов [45] и продуктов альтернативного сплайсинга, специфичных для раковых клеток [46].
Детекция гибридных генов[править | править код]
Гибридизация генов происходит из-за различных структурных модификаций в геноме и может быть связана с раком [47]. Возможность анализировать весь транскриптом образца с помощью секвенирования РНК делает этот метод привлекательным для поиска подобных частых преобразований при раковой трансформации клеток [48].
ENCODE и modENCODE[править | править код]
Основная статья: ENCODEСеквенирование РНК является одним из основных методов исследований, проводимых в рамках проектов ENCODE и modENCODE, направленных на создание базы данных элементов генома человека [49] и основных модельных объектов молекулярной биологии [50][51].
- ↑ Haas Brian J, Zody Michael C. Advancing RNA-Seq analysis (англ.) // Nature Biotechnology. — 2010. — May (vol. 28, no. 5). — P. 421—423. — ISSN 1087-0156. — DOI:10.1038/nbt0510-421. [исправить]
- ↑ Marioni J. C., Mason C. E., Mane S. M., Stephens M., Gilad Y. RNA-seq: An assessment of technical reproducibility and comparison with gene expression arrays (англ.) // Genome Research. — 2008. — 30 July (vol. 18, no. 9). — P. 1509—1517. — ISSN 1088-9051. — DOI:10.1101/gr.079558.108. [исправить]
- ↑ Nookaew Intawat, Papini Marta, Pornputtapong Natapol, Scalcinati Gionata, Fagerberg Linn, Uhlén Matthias, Nielsen Jens. A comprehensive comparison of RNA-Seq-based transcriptome analysis from reads to differential gene expression and cross-comparison with microarrays: a case study in Saccharomyces cerevisiae (англ.) // Nucleic Acids Research. — 2012. — 8 September (vol. 40, no. 20). — P. 10084—10097. — ISSN 1362-4962. — DOI:10.1093/nar/gks804. [исправить]
- ↑ Li Dong, Tian Lifeng, Hakonarson Hakon. Increasing diagnostic yield by RNA-Sequencing in rare disease—bypass hurdles of interpreting intronic or splice-altering variants (англ.) // Annals of Translational Medicine. — 2018. — April (vol. 6, no. 7). — P. 126—126. — ISSN 2305-5839. — DOI:10.21037/atm.2018.01.14. [исправить]
- ↑ Shalek Alex K., Benson Mikael. Single-cell analyses to tailor treatments (англ.) // Science Translational Medicine. — 2017. — 20 September (vol. 9, no. 408). — P. eaan4730. — ISSN 1946-6234. — DOI:10.1126/scitranslmed.aan4730. [исправить]
- ↑ Margulies Marcel, Egholm Michael, Altman William E., Attiya Said, Bader Joel S., Bemben Lisa A., Berka Jan, Braverman Michael S., Chen Yi-Ju, Chen Zhoutao, Dewell Scott B., Du Lei, Fierro Joseph M., Gomes Xavier V., Godwin Brian C., He Wen, Helgesen Scott, Ho Chun He, Irzyk Gerard P., Jando Szilveszter C., Alenquer Maria L. I., Jarvie Thomas P., Jirage Kshama B., Kim Jong-Bum, Knight James R., Lanza Janna R., Leamon John H., Lefkowitz Steven M., Lei Ming, Li Jing, Lohman Kenton L., Lu Hong, Makhijani Vinod B., McDade Keith E., McKenna Michael P., Myers Eugene W., Nickerson Elizabeth, Nobile John R., Plant Ramona, Puc Bernard P., Ronan Michael T., Roth George T., Sarkis Gary J., Simons Jan Fredrik, Simpson John W., Srinivasan Maithreyan, Tartaro Karrie R., Tomasz Alexander, Vogt Kari A., Volkmer Greg A., Wang Shally H., Wang Yong, Weiner Michael P., Yu Pengguang, Begley Richard F., Rothberg Jonathan M. Genome sequencing in microfabricated high-density picolitre reactors (англ.) // Nature. — 2005. — 31 July (vol. 437, no. 7057). — P. 376—380. — ISSN 0028-0836. — DOI:10.1038/nature03959. [исправить]
- ↑ Bennett Simon T, Barnes Colin, Cox Anthony, Davies Lisa, Brown Clive. Toward the $1000 human genome (англ.) // Pharmacogenomics. — 2005. — July (vol. 6, no. 4). — P. 373—382. — ISSN 1462-2416. — DOI:10.1517/14622416.6.4.373. [исправить]
- ↑ Nagalakshmi U., Wang Z., Waern K., Shou C., Raha D., Gerstein M., Snyder M. The Transcriptional Landscape of the Yeast Genome Defined by RNA Sequencing (англ.) // Science. — 2008. — 6 June (vol. 320, no. 5881). — P. 1344—1349. — ISSN 0036-8075. — DOI:10.1126/science.1158441. [исправить]
- ↑ Lister Ryan, O’Malley Ronan C., Tonti-Filippini Julian, Gregory Brian D., Berry Charles C., Millar A. Harvey, Ecker Joseph R. Highly Integrated Single-Base Resolution Maps of the Epigenome in Arabidopsis (англ.) // Cell. — 2008. — May (vol. 133, no. 3). — P. 523—536. — ISSN 0092-8674. — DOI:10.1016/j.cell.2008.03.029. [исправить]
- ↑ Mortazavi Ali, Williams Brian A, McCue Kenneth, Schaeffer Lorian, Wold Barbara. Mapping and quantifying mammalian transcriptomes by RNA-Seq (англ.) // Nature Methods. — 2008. — 30 May (vol. 5, no. 7). — P. 621—628. — ISSN 1548-7091. — DOI:10.1038/nmeth.1226. [исправить]
- ↑ Metzker Michael L. Sequencing technologies — the next generation (англ.) // Nature Reviews Genetics. — 2009. — 8 December (vol. 11, no. 1). — P. 31—46. — ISSN 1471-0056. — DOI:10.1038/nrg2626. [исправить]
- ↑ Oliver Smith , Glenn Dunshea, Mikkel-Holger S. Sinding, Sergey Fedorov, Mietje Germonpre, Hervé Bocherens, M. T. P. Gilbert. Ancient RNA from Late Pleistocene permafrost and historical canids shows tissue-specific transcriptome survival, July 30, 2019
- ↑ 1 2 3 4 5 6 7 8 9 10 Hrdlickova Radmila, Toloue Masoud, Tian Bin. RNA-Seq methods for transcriptome analysis (англ.) // Wiley Interdisciplinary Reviews: RNA. — 2016. — 19 May (vol. 8, no. 1). — P. e1364. — ISSN 1757-7004. — DOI:10.1002/wrna.1364. [исправить]
- ↑ Extraction/mRNA Isolation Protocols (неопр.). www.protocol-online.org. Дата обращения 6 мая 2019.
- ↑ Uzman A. Molecular Cell Biology (4th edition) Harvey Lodish, Arnold Berk, S. Lawrence Zipursky, Paul Matsudaira, David Baltimore and James Darnell; Freeman & Co., New York, NY, 2000, 1084 pp., list price $102.25, ISBN 0-7167-3136-3 (англ.) // Biochemistry and Molecular Biology Education. — 2001. — Vol. 29, no. 3. — P. 126—128. — ISSN 1470-8175. — DOI:10.1016/S1470-8175(01)00023-6. [исправить]
- ↑ Archer Stuart K., Shirokikh Nikolay E., Preiss Thomas. Probe-Directed Degradation (PDD) for Flexible Removal of Unwanted cDNA Sequences from RNA-Seq Libraries (англ.) // Current Protocols in Human Genetics. — 2015. — 1 April. — P. 11.15.1—11.15.36. — ISBN 9780471142904. — DOI:10.1002/0471142905.hg1115s85. [исправить]
- ↑ Archer Stuart K, Shirokikh Nikolay E, Preiss Thomas. Selective and flexible depletion of problematic sequences from RNA-seq libraries at the cDNA stage (англ.) // BMC Genomics. — 2014. — Vol. 15, no. 1. — P. 401. — ISSN 1471-2164. — DOI:10.1186/1471-2164-15-401. [исправить]
- ↑ Фермент TEX (неопр.).
- ↑ Sharma Cynthia M., Hoffmann Steve, Darfeuille Fabien, Reignier Jérémy, Findeiß Sven, Sittka Alexandra, Chabas Sandrine, Reiche Kristin, Hackermüller Jörg, Reinhardt Richard, Stadler Peter F., Vogel Jörg. The primary transcriptome of the major human pathogen Helicobacter pylori (англ.) // Nature. — 2010. — 17 February (vol. 464, no. 7286). — P. 250—255. — ISSN 0028-0836. — DOI:10.1038/nature08756. [исправить]
- ↑ Hafner Markus, Landgraf Pablo, Ludwig Janos, Rice Amanda, Ojo Tolulope, Lin Carolina, Holoch Daniel, Lim Cindy, Tuschl Thomas. Identification of microRNAs and other small regulatory RNAs using cDNA library sequencing (англ.) // Methods. — 2008. — January (vol. 44, no. 1). — P. 3—12. — ISSN 1046-2023. — DOI:10.1016/j.ymeth.2007.09.009. [исправить]
- ↑ Langevin Stanley A., Bent Zachary W., Solberg Owen D., Curtis Deanna J., Lane Pamela D., Williams Kelly P., Schoeniger Joseph S., Sinha Anupama, Lane Todd W., Branda Steven S. Peregrine (англ.) // RNA Biology. — 2013. — April (vol. 10, no. 4). — P. 502—515. — ISSN 1547-6286. — DOI:10.4161/rna.24284. [исправить]
- ↑ Townsley Brad T., Covington Michael F., Ichihashi Yasunori, Zumstein Kristina, Sinha Neelima R. BrAD-seq: Breath Adapter Directional sequencing: a streamlined, ultra-simple and fast library preparation protocol for strand specific mRNA library construction (англ.) // Frontiers in Plant Science. — 2015. — 22 May (vol. 6). — ISSN 1664-462X. — DOI:10.3389/fpls.2015.00366. [исправить]
- ↑ Chen En-Qiang, Bai Lang, Gong Dao-Yin, Tang Hong. Employment of digital gene expression profiling to identify potential pathogenic and therapeutic targets of fulminant hepatic failure (англ.) // Journal of Translational Medicine. — 2015. — Vol. 13, no. 1. — P. 22. — ISSN 1479-5876. — DOI:10.1186/s12967-015-0380-9. [исправить]
- ↑ Liu Donglin, Graber JoelH. [1] (англ.) // BMC Bioinformatics. — 2006. — Vol. 7, no. 1. — P. 77. — ISSN 1471-2105. — DOI:10.1186/1471-2105-7-77. [исправить]
- ↑ Ozsolak Fatih, Platt Adam R., Jones Dan R., Reifenberger Jeffrey G., Sass Lauryn E., McInerney Peter, Thompson John F., Bowers Jayson, Jarosz Mirna, Milos Patrice M. Direct RNA sequencing (англ.) // Nature. — 2009. — 23 September (vol. 461, no. 7265). — P. 814—818. — ISSN 0028-0836. — DOI:10.1038/nature08390. [исправить]
- ↑ Trapnell Cole, Williams Brian A, Pertea Geo, Mortazavi Ali, Kwan Gordon, van Baren Marijke J, Salzberg Steven L, Wold Barbara J, Pachter Lior. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation (англ.) // Nature Biotechnology. — 2010. — May (vol. 28, no. 5). — P. 511—515. — ISSN 1087-0156. — DOI:10.1038/nbt.1621. [исправить]
- ↑ Birol I., Jackman S. D., Nielsen C. B., Qian J. Q., Varhol R., Stazyk G., Morin R. D., Zhao Y., Hirst M., Schein J. E., Horsman D. E., Connors J. M., Gascoyne R. D., Marra M. A., Jones S. J. M. De novo transcriptome assembly with ABySS (англ.) // Bioinformatics. — 2009. — 15 June (vol. 25, no. 21). — P. 2872—2877. — ISSN 1367-4803. — DOI:10.1093/bioinformatics/btp367. [исправить]
- ↑ Graveley Brenton R., Brooks Angela N., Carlson Joseph W., Duff Michael O., Landolin Jane M., Yang Li, Artieri Carlo G., van Baren Marijke J., Boley Nathan, Booth Benjamin W., Brown James B., Cherbas Lucy, Davis Carrie A., Dobin Alex, Li Renhua, Lin Wei, Malone John H., Mattiuzzo Nicolas R., Miller David, Sturgill David, Tuch Brian B., Zaleski Chris, Zhang Dayu, Blanchette Marco, Dudoit Sandrine, Eads Brian, Green Richard E., Hammonds Ann, Jiang Lichun, Kapranov Phil, Langton Laura, Perrimon Norbert, Sandler Jeremy E., Wan Kenneth H., Willingham Aarron, Zhang Yu, Zou Yi, Andrews Justen, Bickel Peter J., Brenner Steven E., Brent Michael R., Cherbas Peter, Gingeras Thomas R., Hoskins Roger A., Kaufman Thomas C., Oliver Brian, Celniker Susan E. The developmental transcriptome of Drosophila melanogaster (англ.) // Nature. — 2010. — 22 December (vol. 471, no. 7339). — P. 473—479. — ISSN 0028-0836. — DOI:10.1038/nature09715. [исправить]
- ↑ Xie Linglin, Weichel Brent, Ohm Joyce, Zhang Ke. An integrative analysis of DNA methylation and RNA-Seq data for human heart, kidney and liver (англ.) // BMC Systems Biology. — 2011. — Vol. 5, no. Suppl 3. — P. S4. — ISSN 1752-0509. — DOI:10.1186/1752-0509-5-S3-S4. [исправить]
- ↑ Lee J. H., Daugharthy E. R., Scheiman J., Kalhor R., Yang J. L., Ferrante T. C., Terry R., Jeanty S. S. F., Li C., Amamoto R., Peters D. T., Turczyk B. M., Marblestone A. H., Inverso S. A., Bernard A., Mali P., Rios X., Aach J., Church G. M. Highly Multiplexed Subcellular RNA Sequencing in Situ (англ.) // Science. — 2014. — 27 February (vol. 343, no. 6177). — P. 1360—1363. — ISSN 0036-8075. — DOI:10.1126/science.1250212. [исправить]
- ↑ Lee J. H., Daugharthy E. R., Scheiman J., Kalhor R., Yang J. L., Ferrante T. C., Terry R., Jeanty S. S. F., Li C., Amamoto R., Peters D. T., Turczyk B. M., Marblestone A. H., Inverso S. A., Bernard A., Mali P., Rios X., Aach J., Church G. M. Highly Multiplexed Subcellular RNA Sequencing in Situ (англ.) // Science. — 2014. — 27 February (vol. 343, no. 6177). — P. 1360—1363. — ISSN 0036-8075. — DOI:10.1126/science.1250212. [исправить]
- ↑ Jakhesara Subhash J., Koringa Prakash G., Bhatt Vaibhav D., Shah Tejas M., Vangipuram Shankar, Shah Siddharth, Joshi Chaitanya G. RNA-Seq reveals differentially expressed isoforms and novel splice variants in buccal mucosal cancer (англ.) // Gene. — 2013. — March (vol. 516, no. 1). — P. 24—32. — ISSN 0378-1119. — DOI:10.1016/j.gene.2012.11.079. [исправить]
- ↑ Adiconis Xian, Haber Adam L., Simmons Sean K., Levy Moonshine Ami, Ji Zhe, Busby Michele A., Shi Xi, Jacques Justin, Lancaster Madeline A., Pan Jen Q., Regev Aviv, Levin Joshua Z. Comprehensive comparative analysis of 5′-end RNA-sequencing methods (англ.) // Nature Methods. — 2018. — 4 June (vol. 15, no. 7). — P. 505—511. — ISSN 1548-7091. — DOI:10.1038/s41592-018-0014-2. [исправить]
- ↑ Wang Weichen, Qin Zhiyi, Feng Zhixing, Wang Xi, Zhang Xuegong. Identifying differentially spliced genes from two groups of RNA-seq samples (англ.) // Gene. — 2013. — April (vol. 518, no. 1). — P. 164—170. — ISSN 0378-1119. — DOI:10.1016/j.gene.2012.11.045. [исправить]
- ↑ Liu Qi, Chen Chong, Shen Enjian, Zhao Fangqing, Sun Zhongsheng, Wu Jinyu. Detection, annotation and visualization of alternative splicing from RNA-Seq data with SplicingViewer (англ.) // Genomics. — 2012. — March (vol. 99, no. 3). — P. 178—182. — ISSN 0888-7543. — DOI:10.1016/j.ygeno.2011.12.003. [исправить]
- ↑ Park E., Williams B., Wold B. J., Mortazavi A. RNA editing in the human ENCODE RNA-seq data (англ.) // Genome Research. — 2012. — 1 September (vol. 22, no. 9). — P. 1626—1633. — ISSN 1088-9051. — DOI:10.1101/gr.134957.111. [исправить]
- ↑ Peng Zhiyu, Cheng Yanbing, Tan Bertrand Chin-Ming, Kang Lin, Tian Zhijian, Zhu Yuankun, Zhang Wenwei, Liang Yu, Hu Xueda, Tan Xuemei, Guo Jing, Dong Zirui, Liang Yan, Bao Li, Wang Jun. Comprehensive analysis of RNA-Seq data reveals extensive RNA editing in a human transcriptome (англ.) // Nature Biotechnology. — 2012. — 12 February (vol. 30, no. 3). — P. 253—260. — ISSN 1087-0156. — DOI:10.1038/nbt.2122. [исправить]
- ↑ Peng Zhiyu, Cheng Yanbing, Tan Bertrand Chin-Ming, Kang Lin, Tian Zhijian, Zhu Yuankun, Zhang Wenwei, Liang Yu, Hu Xueda, Tan Xuemei, Guo Jing, Dong Zirui, Liang Yan, Bao Li, Wang Jun. Comprehensive analysis of RNA-Seq data reveals extensive RNA editing in a human transcriptome (англ.) // Nature Biotechnology. — 2012. — 12 February (vol. 30, no. 3). — P. 253—260. — ISSN 1087-0156. — DOI:10.1038/nbt.2122. [исправить]
- ↑ Pullirsch Dieter, Jantsch Michael F. Proteome diversification by adenosine to inosine RNA-editing (англ.) // RNA Biology. — 2010. — March (vol. 7, no. 2). — P. 205—212. —
Редактирование РНК — Википедия
Строение эдитосомы — комплекса, осуществляющего редактирование РНК путём вставки или делецииРедакти́рование РНК (англ. RNA editing) — процесс, в ходе которого нуклеотиды в новосинтезированной РНК подвергаются химическим модификациям. Редактирование РНК также может включать вставку, делецию или замену нуклеотидов в молекуле РНК. Редактирование РНК — довольно редкий процесс, и типичные этапы процессинга мРНК (кэпирование, полиаденилирование, сплайсинг) обычно не рассматриваются как редактирование.
Редактированию подвергаются некоторые тРНК, рРНК, мРНК и микроРНК у эукариот и поражающих их вирусов, а также прокариот[1]. Редактирование РНК происходит в клеточном ядре и цитозоле, а также в митохондриях и пластидах. У позвоночных редактирование РНК происходит редко и обычно выражается во внесении лишь небольших изменений в молекулу РНК. У некоторых других организмов, напротив, редактирование РНК происходит так интенсивно, что в конечной мРНК остаётся лишь несколько неотредактированных нуклеотидов. Процессы редактирования РНК очень разнообразны и в ходе эволюции появились независимо. Редактирование РНК может приводить к превращению цитидина (C) в уридин (U) и аденозина (A) в инозин (I) в результате дезаминирования, а также присоединению к РНК новых нуклеотидов и удалению уже входящих в её состав. Редактирование РНК может так изменить мРНК, что поменяется аминокислотный состав кодируемого белка и он будет отличаться от полипептида, предсказанного по последовательности гена[2].
Редактирование путём вставки или делеции[править | править код]
Эффект вставки урацилов в пре-мРНКРедактирование РНК путём встраивания или удаления урацилов было описано в митохондриях кинетопластид Trypanosoma brucei[en][3][4][5][6][7][8][9][10]. Редактирование РНК начинается со спаривания первичного[en] нередактированного транскрипта с направляющей (гидовой) РНК, которая содержит комплементарные последовательности около сайтов встраивания или удаления. Образующийся двуцепочечный участок далее покрывается эдитосомой — крупным многобелковым комплексом, катализирующим редактирование РНК[4][5]. Эдитосома начинает встраивание уридинов по первому положению неспаренных нуклеотидов. Встраиваемые уридины образуют комплементарные связи с направляющей РНК, и встраивание продолжается, пока в направляющей РНК встречаются A или G, и останавливается при появлении C или U[6][7]. Встраиваемые нуклеотиды вызывают сдвиг рамки считывания и приводят к тому, что транслируемый белок отличается от кодирующей области гена.
В ходе редактирования происходит разрезание по сайту, где не образуются комплементарные пары между направляющей РНК и нередактированным транскриптом. Следующая стадия катализируется ферментом концевой уридинтрансферазой, которая добавляет U из UTP к 3′-концу мРНК[8]. «Открытые» концы удерживаются другими белками эдитосомного комплекса. Другой фермент, U-специфичная экзорибонуклеаза, удаляет неспаренные уридины. После того как эдитосомный комплекс делает последовательность мРНК комплементарной направляющей РНК, РНК-лигаза соединяет концы отредактированной мРНК[9]. Эдитосомный комплекс способен редактировать мРНК лишь в направлении от 3′-конца к 5′-концу. Комплекс способен редактировать только по одной РНК в момент времени. РНК, для которой требуется значительное редактирование, нуждается в нескольких направляющих РНК и нескольких эдитосомных комплексах.
Редактирование посредством дезаминирования[править | править код]
Редактирование C → U[править | править код]
Редактирование C → U в человеческом гене apoBПод действием фермента цитидиндезаминазы[en] происходит реакция дезаминирования, превращающая цитидин в урацил. Редактирование C → U может быть рассмотрено на примере гена аполипопротеина B у человека. В печени экспрессируется его изоформа Apo B100, а в кишечнике — apo B48. В клетках кишечника мРНК аполипопротеина B подвергается C → U редактированию, из-за которого кодон CAA превращается в стоп-кодон UAA, и синтезируется изоформа apo B48. Редактирование C → U часто происходит в митохондриальных РНК цветковых растений. У разных растений интенсивность редактирования C → U варьирует: у мха Funaria hygrometrica[en] в митохондриальных РНК происходит восемь актов редактирования, а у плауна Isoetes engelmannii[en] — около 1700[11]. Превращение C → U осуществляется семейством белков с пентатрикопептидными повторами (англ. pentatricopeptide repeat, PPR). Это семейство богато представлено у цветковых растений: так, у Arabidopsis насчитывается 450 белков этого семейства. Белки PPR были описаны также в пластидах и митохондриях[12].
Редактирование A → I[править | править код]
На долю превращений аденозина в инозин (A → I) приходится около 90 % всех случаев редактирования РНК. Дезаминирование аденозина катализируется аденозиндезаминазой, специфичной к двуцепочечной РНК[en] (англ. double-stranded RNA-specific adenosine deaminase, ADAR), которые обычно действуют на предшественников мРНК (пре-мРНК). Дезаминирование аденозина с образованием инозина разрушает спаривание оснований в двуцепочечной РНК (дцРНК), поэтому некоторые двуцепочечные РНК дают начало меньшему количеству малых интерферирующих РНК, чем другие. В дезаминированной дцРНК формируются wobble-взаимодействия между парами оснований, из-за чего молекула приобретает необычную структуру, подавляющую инициацию трансляции. РНК, содержащая пары U — I, привлекает метилазы[en], участвующие в образовании гетерохроматина, кроме того, сайты редактирования A → I часто совпадают с сайтами связывания с микроРНК, что создаёт конкуренцию между двумя процессами[13]. Редактирование A → I активно изучается в связи с концепцией эпитранскриптомики[en], которая гласит, что химические модификации РНК могут влиять на её функции[14][15]. Когда в ходе трансляции рибосома встречается с инозином, она распознаёт его как гуанин, хотя некоторые исследования свидетельствуют, что I может считываться как A и U. Кроме того, показано, что рибосома замедляется, когда встречает инозин в мРНК[16].
Интенсивность редактирования A → I может быть тканеспецифичной, как в случае филамина A[en] человека[17]. В числе факторов, влияющих на интенсивность редактирования, называют эффективность сплайсинга пре-мРНК[18].
В связи с интенсивным развитием технологий высокопроизводительного секвенирования стало возможным создание баз данных, содержащих сведения о редактировании различных РНК. В 2013 году был открыт каталог RADAR (от англ. Rigorously Annotated Database of A-to-I RNA editing), содержащий сайты редактирования A → I, а также данные о таких тканеспецифичных заменах у человека, мыши и дрозофилы. В базу постоянно заносятся новооткрытые сайты редактирования[19].
Альтернативное редактирование мРНК[править | править код]
В случае гена WT1[en] было описано альтернативное редактирование РНК U → C[20], кроме того, случаи неканонического редактирования G → A известны для транскриптов гетерогенного ядерного рибонуклеопротеина[en] K в нормальных и злокачественных клетках толстой кишки[21]. Редактирование G → A было также отмечено, наряду с неклассическим превращением U → C, в транскриптах триптофангидроксилазы 2 в нейронах[22]. Хотя наиболее простым механизмом превращения U → C может быть обратное аминирование, предполагается, что в митохондриальных транскриптах в основе редактирования U → C лежат реакции трансаминирования и трансгликозилирования[23]. В середине 2010-х годов исследование редактирования G → A в транскрипте WT1 показало, что это превращение наиболее активно происходит в двух точках под действием фермента APOBEC3A[en] (каталитический полипептид 3A фермента редактирования мРНК аполипопротеина B)[24].
Редактирование РНК в митохондриях и пластидах растений[править | править код]
Многочисленные исследования показали, что в митохондриях растений РНК подвергается только редактированию C → U и, очень редко, U → C[25][26][27][28][29][30][31][32][33][34][35][36][37]. Сайты редактирования находятся преимущественно в кодирующих областях мРНК, интронах и других нетранслируемых областях[27]. Редактирование РНК может быть необходимым для восстановления функциональности молекул тРНК[29][30]. Хотя сайты редактирования C → U в пластидах и митохондриях изучены относительно хорошо[38], все белки, образующие соответствующие эдитосомы, ещё не идентифицированы. Показано, что в распознавании сайтов редактирования участвуют члены многочисленного белкового семейства PPR[39]. Для редактирования в некоторых сайтах необходимы белки семейства MORF (от англ. Multiple Organellar RNA editing Factor). Так как некоторые белки семейства MORF взаимодействуют с членами семейства PPR, возможно, что белки MORF входят в состав эдитосомы[40]. Ферменты, ответственные за дезаминирование и трансаминирование в органеллах, ещё не определены, но, возможно, эти реакции осуществляют члены семейства PPR. Редактирование РНК необходимо для нормального клеточного дыхания и трансляции в клетках растений. Редактирование антикодоновой петли может восстанавливать функциональность молекул тРНК[41].
Некоторые вирусы, такие как возбудители кори, свинки[en] и парагриппа, используют редактирование РНК для получения новых вариантов белков[42][43]. Вирусные РНК синтезируют кодируемые вирусами РНК-зависимые РНК-полимеразы, которые иногда «запинаются» на определённых сочетаниях нуклеотидов. Остановка РНК-полимеразы может привести к вставке дополнительных гуаниновых или адениноных нуклеотидов. Вставка дополнительных нуклеотидов смещает рамку считывания, что приводит к образованию новых форм белков. Кроме того, к 3′-концу созревающих вирусных мРНК может добавляться до нескольких сотен дополнительных адениновых нуклеотидов, которые стабилизируют мРНК[44].
Редактирование РНК может выполнять несколько функций. В частности, оно может быть связано с деградацией РНК. В 2008 году было показано, что ADAR и UPF1[en] (фермент, участвующий в нонсенс-опосредованном распаде) взаимодействуют друг с другом и со сплайсосомой, формируя супрасплайсосому, и могут подавлять экспрессию некоторых генов. Подобно альтернативному сплайсингу, редактирование РНК может приводить к появлению новых форм белков за счёт замен, а также появления или удаления сайтов сплайсинга. Редактирование некодирующих РНК может изменить их структуру или привести к новым мутациям в вирусных геномах. Редактирование РНК может также представлять собой механизм защиты от ретротранспозонов[45].
Функции редактирования РНКУ истоков редактирования РНК животных могли стоять мононуклеотиддезаминазы, которые дали начало обширным белковым семействам, включающим такие ферменты редактирования РНК, как ADAR и APOBEC1[en]. Последовательности этих генов сближают их с бактериальными дезаминазами, участвующими в метаболизме нуклеотидов. Аденозиндезаминаза кишечной палочки Escherichia coli не может катализировать дезаминирование нуклеозида в РНК: её «карман», в котором и происходит реакция, слишком мал, чтобы вместить целую молекулу РНК. Однако расширение активного сайта наблюдается в человеческих белках APOBEC1 и ADAR, которые могут катализировать дезаминирование РНК[46][47]. Масштабное редактирование РНК, зависимое от направляющих РНК, такое как вставка нескольких уридиновых нуклеотидов у трипаносом, представляет собой совершенно иную биохимическую реакцию. Ферменты, ответственные за этот процесс, произошли от абсолютно других предшественников[48][49]. Однако специфичность вставки нуклеотидов, определяемая взаимодействием направляющей РНК с мРНК, схожа с процессом редактирования тРНК, протекающим в митохондриях животных и амёб Acanthamoeba[en][50]. Более того, этот процесс схож с метилированием рибозы в рРНК с участием направляющих РНК, которое происходит у всех эукариот[51].
Кардинально различающиеся пути редактирования РНК свидетельствуют, что они возникали независимо в ходе эволюции[52]. В некоторых источниках редактирование РНК рассматривают как процесс, направленный на устранение дефектов в последовательностях генов или их компенсацию[53].
- ↑ Su A. A., Randau L. A-to-I and C-to-U editing within transfer RNAs. (англ.) // Biochemistry. Biokhimiia. — 2011. — August (vol. 76, no. 8). — P. 932—937. — DOI:10.1134/S0006297911080098. — PMID 22022967. [исправить]
- ↑ Brennicke A., Marchfelder A., Binder S. RNA editing. (англ.) // FEMS Microbiology Reviews. — 1999. — June (vol. 23, no. 3). — P. 297—316. — DOI:10.1111/j.1574-6976.1999.tb00401.x. — PMID 10371035. [исправить]
- ↑ Benne R. RNA editing in trypanosomes. (англ.) // European Journal Of Biochemistry. — 1994. — 1 April (vol. 221, no. 1). — P. 9—23. — DOI:10.1111/j.1432-1033.1994.tb18710.x. — PMID 7513284. [исправить]
- ↑ 1 2 Arts G. J., Benne R. Mechanism and evolution of RNA editing in kinetoplastida. (англ.) // Biochimica Et Biophysica Acta. — 1996. — 3 June (vol. 1307, no. 1). — P. 39—54. — DOI:10.1016/0167-4781(96)00021-8. — PMID 8652667. [исправить]
- ↑ 1 2 Alfonzo J. D., Thiemann O., Simpson L. The mechanism of U insertion/deletion RNA editing in kinetoplastid mitochondria. (англ.) // Nucleic Acids Research. — 1997. — 1 October (vol. 25, no. 19). — P. 3751—3759. — DOI:10.1093/nar/25.19.3751. — PMID 9380494. [исправить]
- ↑ 1 2 Blum B., Bakalara N., Simpson L. A model for RNA editing in kinetoplastid mitochondria: «guide» RNA molecules transcribed from maxicircle DNA provide the edited information. (англ.) // Cell. — 1990. — 26 January (vol. 60, no. 2). — P. 189—198. — DOI:10.1016/0092-8674(90)90735-w. — PMID 1688737. [исправить]
- ↑ 1 2 Kable M. L., Heidmann S., Stuart K. D. RNA editing: getting U into RNA. (англ.) // Trends In Biochemical Sciences. — 1997. — May (vol. 22, no. 5). — P. 162—166. — PMID 9175474. [исправить]
- ↑ 1 2 Simpson L., Thiemann O. H. Sense from nonsense: RNA editing in mitochondria of kinetoplastid protozoa and slime molds. (англ.) // Cell. — 1995. — 16 June (vol. 81, no. 6). — P. 837—840. — DOI:10.1016/0092-8674(95)90003-9. — PMID 7781060. [исправить]
- ↑ 1 2 Stuart K. RNA editing in mitochondrial mRNA of trypanosomatids. (англ.) // Trends In Biochemical Sciences. — 1991. — February (vol. 16, no. 2). — P. 68—72. — PMID 1713359. [исправить]
- ↑ van der Spek H., Arts G. J., Zwaal R. R., van den Burg J., Sloof P., Benne R. Conserved genes encode guide RNAs in mitochondria of Crithidia fasciculata. (англ.) // The EMBO Journal. — 1991. — May (vol. 10, no. 5). — P. 1217—1224. — PMID 1708723. [исправить]
- ↑ Takenaka M., Verbitskiy D., Zehrmann A., Härtel B., Bayer-Császár E., Glass F., Brennicke A. RNA editing in plant mitochondria—connecting RNA target sequences and acting proteins. (англ.) // Mitochondrion. — 2014. — November (vol. 19 Pt B). — P. 191—197. — DOI:10.1016/j.mito.2014.04.005. — PMID 24732437. [исправить]
- ↑ Shikanai T. RNA editing in plants: Machinery and flexibility of site recognition. (англ.) // Biochimica Et Biophysica Acta. — 2015. — September (vol. 1847, no. 9). — P. 779—785. — DOI:10.1016/j.bbabio.2014.12.010. — PMID 25585161. [исправить]
- ↑ Nishikura K. Functions and regulation of RNA editing by ADAR deaminases. (англ.) // Annual Review Of Biochemistry. — 2010. — Vol. 79. — P. 321—349. — DOI:10.1146/annurev-biochem-060208-105251. — PMID 20192758. [исправить]
- ↑ Tajaddod M., Jantsch M. F., Licht K. The dynamic epitranscriptome: A to I editing modulates genetic information. (англ.) // Chromosoma. — 2016. — March (vol. 125, no. 1). — P. 51—63. — DOI:10.1007/s00412-015-0526-9. — PMID 26148686. [исправить]
- ↑ Licht K., Jantsch M. F. Rapid and dynamic transcriptome regulation by RNA editing and RNA modifications. (англ.) // The Journal Of Cell Biology. — 2016. — 11 April (vol. 213, no. 1). — P. 15—22. — DOI:10.1083/jcb.201511041. — PMID 27044895. [исправить]
- ↑ Licht K., Hartl M., Amman F., Anrather D., Janisiw M. P., Jantsch M. F. Inosine induces context-dependent recoding and translational stalling. (англ.) // Nucleic Acids Research. — 2019. — 10 January (vol. 47, no. 1). — P. 3—14. — DOI:10.1093/nar/gky1163. — PMID 30462291. [исправить]
- ↑ Stulić M., Jantsch M. F. Spatio-temporal profiling of Filamin A RNA-editing reveals ADAR preferences and high editing levels outside neuronal tissues. (англ.) // RNA Biology. — 2013. — October (vol. 10, no. 10). — P. 1611—1617. — DOI:10.4161/rna.26216. — PMID 24025532. [исправить]
- ↑ Licht K., Kapoor U., Mayrhofer E., Jantsch M. F. Adenosine to Inosine editing frequency controlled by splicing efficiency. (англ.) // Nucleic Acids Research. — 2016. — 27 July (vol. 44, no. 13). — P. 6398—6408. — DOI:10.1093/nar/gkw325. — PMID 27112566. [исправить]
- ↑ Ramaswami G., Li J. B. RADAR: a rigorously annotated database of A-to-I RNA editing. (англ.) // Nucleic Acids Research. — 2014. — January (vol. 42). — P. D109—113. — DOI:10.1093/nar/gkt996. — PMID 24163250. [исправить]
- ↑ Sharma P. M., Bowman M., Madden S. L., Rauscher FJ 3rd., Sukumar S. RNA editing in the Wilms’ tumor susceptibility gene, WT1. (англ.) // Genes & Development. — 1994. — 15 March (vol. 8, no. 6). — P. 720—731. — DOI:10.1101/gad.8.6.720. — PMID 7926762. [исправить]
- ↑ Klimek-Tomczak K., Mikula M., Dzwonek A., Paziewska A., Karczmarski J., Hennig E., Bujnicki J. M., Bragoszewski P., Denisenko O., Bomsztyk K., Ostrowski J. Editing of hnRNP K protein mRNA in colorectal adenocarcinoma and surrounding mucosa. (англ.) // British Journal Of Cancer. — 2006. — 27 February (vol. 94, no. 4). — P. 586—592. — DOI:10.1038/sj.bjc.6602938. — PMID 16404425. [исправить]
- ↑ Grohmann M., Hammer P., Walther M., Paulmann N., Büttner A., Eisenmenger W., Baghai T. C., Schüle C., Rupprecht R., Bader M., Bondy B., Zill P., Priller J., Walther D. J. Alternative splicing and extensive RNA editing of human TPh3 transcripts. (англ.) // PloS One. — 2010. — 29 January (vol. 5, no. 1). — P. e8956—8956. — DOI:10.1371/journal.pone.0008956. — PMID 20126463. [исправить]
- ↑ Castandet B., Araya A. RNA editing in plant organelles. Why make it easy? (англ.) // Biochemistry. Biokhimiia. — 2011. — August (vol. 76, no. 8). — P. 924—931. — DOI:10.1134/S0006297911080086. — PMID 22022966. [исправить]
- ↑ Niavarani A., Currie E., Reyal Y., Anjos-Afonso F., Horswell S., Griessinger E., Luis Sardina J., Bonnet D. APOBEC3A is implicated in a novel class of G-to-A mRNA editing in WT1 transcripts. (англ.) // PloS One. — 2015. — Vol. 10, no. 3. — P. e0120089—0120089. — DOI:10.1371/journal.pone.0120089. — PMID 25807502. [исправить]
- ↑ Covello P. S., Gray M. W. RNA editing in plant mitochondria. (англ.) // Nature. — 1989. — 19 October (vol. 341, no. 6243). — P. 662—666. — DOI:10.1038/341662a0. — PMID 2552326. [исправить]
- ↑ Gualberto J. M., Lamattina L., Bonnard G., Weil J. H., Grienenberger J. M. RNA editing in wheat mitochondria results in the conservation of protein sequences. (англ.) // Nature. — 1989. — 19 October (vol. 341, no. 6243). — P. 660—662. — DOI:10.1038/341660a0. — PMID 2552325. [исправить]
- ↑ 1 2 Hiesel R., Wissinger B., Schuster W., Brennicke A. RNA editing in plant mitochondria. (англ.) // Science (New York, N.Y.). — 1989. — 22 December (vol. 246, no. 4937). — P. 1632—1634. — DOI:10.1126/science.2480644. — PMID 2480644. [исправить]
- ↑ Hoch B., Maier R. M., Appel K., Igloi G. L., Kössel H. Editing of a chloroplast mRNA by creation of an initiation codon. (англ.) // Nature. — 1991. — 12 September (vol. 353, no. 6340). — P. 178—180. — DOI:10.1038/353178a0. — PMID 1653905. [исправить]
- ↑ 1 2 Pring D., Brennicke A., Schuster W. RNA editing gives a new meaning to the genetic information in mitochondria and chloroplasts. (англ.) // Plant Molecular Biology. — 1993. — March (vol. 21, no. 6). — P. 1163—1170. — PMID 8490134. [исправить]
- ↑ 1 2 Wissinger B., Brennicke A., Schuster W. Regenerating good sense: RNA editing and trans splicing in plant mitochondria. (англ.) // Trends In Genetics : TIG. — 1992. — September (vol. 8, no. 9). — P. 322—328. — PMID 1365399. [исправить]
- ↑ Yan J., Zhang Q., Yin P. RNA editing machinery in plant organelles. (англ.) // Science China. Life Sciences. — 2018. — February (vol. 61, no. 2). — P. 162—169. — DOI:10.1007/s11427-017-9170-3. — PMID 29075943. [исправить]
- ↑ Malek O., Lättig K., Hiesel R., Brennicke A., Knoop V. RNA editing in bryophytes and a molecular phylogeny of land plants. (англ.) // The EMBO Journal. — 1996. — 15 March (vol. 15, no. 6). — P. 1403—1411. — PMID 8635473. [исправить]
- ↑ Freyer R., Kiefer-Meyer M. C., Kössel H. Occurrence of plastid RNA editing in all major lineages of land plants. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1997. — 10 June (vol. 94, no. 12). — P. 6285—6290. — DOI:10.1073/pnas.94.12.6285. — PMID 9177209. [исправить]
- ↑ Dietrich A., Small I., Cosset A., Weil J. H., Maréchal-Drouard L. Editing and import: strategies for providing plant mitochondria with a complete set of functional transfer RNAs. (англ.) // Biochimie. — 1996. — Vol. 78, no. 6. — P. 518—529. — PMID 8915541. [исправить]
- ↑ Bock R., Hermann M., Fuchs M. Identification of critical nucleotide positions for plastid RNA editing site recognition. (англ.) // RNA (New York, N.Y.). — 1997. — October (vol. 3, no. 10). — P. 1194—1200. — PMID 9326494. [исправить]
- ↑ Gray M. W., Covello P. S. RNA editing in plant mitochondria and chloroplasts. (англ.) // FASEB Journal : Official Publication Of The Federation Of American Societies For Experimental Biology. — 1993. — January (vol. 7, no. 1). — P. 64—71. — DOI:10.1096/fasebj.7.1.8422976. — PMID 8422976. [исправить]
- ↑ Marchfelder A., Binder S., Brennicke A., Knoop V. Preface // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press (англ.)русск., 1998. — С. 307—323.
- ↑ Takenaka M., Zehrmann A., Verbitskiy D., Härtel B., Brennicke A. RNA editing in plants and its evolution. (англ.) // Annual Review Of Genetics. — 2013. — Vol. 47. — P. 335—352. — DOI:10.1146/annurev-genet-111212-133519. — PMID 24274753. [исправить]
- ↑ Barkan A., Small I. Pentatricopeptide repeat proteins in plants. (англ.) // Annual Review Of Plant Biology. — 2014. — Vol. 65. — P. 415—442. — DOI:10.1146/annurev-arplant-050213-040159. — PMID 24471833. [исправить]
- ↑ Bentolila S., Oh J., Hanson M. R., Bukowski R. Comprehensive high-resolution analysis of the role of an Arabidopsis gene family in RNA editing. (англ.) // PLoS Genetics. — 2013. — June (vol. 9, no. 6). — P. e1003584—1003584. — DOI:10.1371/journal.pgen.1003584. — PMID 23818871. [исправить]
- ↑ Price D. H., Gray M. W. Editing of tRNA // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press (англ.)русск., 1998. — С. 289—306.
- ↑ Curran J., Boeck R., Kolakofsky D. The Sendai virus P gene expresses both an essential protein and an inhibitor of RNA synthesis by shuffling modules via mRNA editing. (англ.) // The EMBO Journal. — 1991. — October (vol. 10, no. 10). — P. 3079—3085. — PMID 1655410. [исправить]
- ↑ Zheng H., Fu T. B., Lazinski D., Taylor J. Editing on the genomic RNA of human hepatitis delta virus. (англ.) // Journal Of Virology. — 1992. — August (vol. 66, no. 8). — P. 4693—4697. — PMID 1629949. [исправить]
- ↑ Kolakofsky D., Hausmann S. Chapter 23: Cotranscriptional Paramyxovirus mRNA Editing: a Contradiction in Terms? // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press (англ.)русск., 1998. — С. 413—420.
- ↑ RNA editing by John W. Kimball (неопр.).
- ↑ Carter C. W. Nucleoside deaminases for cytidine and adenosine: comparisons with deaminases acting on RNA // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press (англ.)русск., 1998. — С. 363—376.
- ↑ Navaratnam N., Fujino T., Bayliss J., Jarmuz A., How A., Richardson N., Somasekaram A., Bhattacharya S., Carter C., Scott J. Escherichia coli cytidine deaminase provides a molecular model for ApoB RNA editing and a mechanism for RNA substrate recognition. (англ.) // Journal Of Molecular Biology. — 1998. — 30 January (vol. 275, no. 4). — P. 695—714. — DOI:10.1006/jmbi.1997.1506. — PMID 9466941. [исправить]
- ↑ Arts G. J., Benne R. Mechanism and evolution of RNA editing in kinetoplastida. (англ.) // Biochimica Et Biophysica Acta. — 1996. — 3 June (vol. 1307, no. 1). — P. 39—54. — DOI:10.1016/0167-4781(96)00021-8. — PMID 8652667. [исправить]
- ↑ Covello P. S., Gray M. W. On the evolution of RNA editing. (англ.) // Trends In Genetics : TIG. — 1993. — August (vol. 9, no. 8). — P. 265—268. — PMID 8379005. [исправить]
- ↑ Lonergan K. M., Gray M. W. Predicted editing of additional transfer RNAs in Acanthamoeba castellanii mitochondria. (англ.) // Nucleic Acids Research. — 1993. — 11 September (vol. 21, no. 18). — P. 4402—4402. — DOI:10.1093/nar/21.18.4402. — PMID 8415006. [исправить]
- ↑ Bachellerie J. P., Cavaille J. Small nucleolar RNAs guide the ribose methylations of eukaryotic rRNAs // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press (англ.)русск., 1998. — С. 255—272.
- ↑ Speijer D. Does constructive neutral evolution play an important role in the origin of cellular complexity? Making sense of the origins and uses of biological complexity. (англ.) // BioEssays : News And Reviews In Molecular, Cellular And Developmental Biology. — 2011. — May (vol. 33, no. 5). — P. 344—349. — DOI:10.1002/bies.201100010. — PMID 21381061. [исправить]
- ↑ Stoltzfus A. On the possibility of constructive neutral evolution. (англ.) // Journal Of Molecular Evolution. — 1999. — August (vol. 49, no. 2). — P. 169—181. — PMID 10441669. [исправить]
Посттранскрипционные модификации | |
|---|---|
Ядерные  | |
РНК-зависимая РНК-полимераза — Википедия
Материал из Википедии — свободной энциклопедии
РНК-зависимая РНК-полимераза (RdRP от англ. RNA-dependent RNA-polymerase), или РНК репликаза — фермент, катализирующий репликацию РНК (синтез РНК по матрице РНК). Использование РНК в качестве матрицы принципиально отличает РНК репликазу от более распространенной среди современных живых организмов ДНК-зависимой РНК-полимеразы, которая катализирует транскрипцию (синтез РНК с использованием в качестве матрицы ДНК).
РНК-зависимые РНК-полимеразы (RdRp) — важнейшие ферменты, закодированные в геноме всех РНК-содержащих вирусов, жизненный цикл которых протекает без стадии ДНК.[2][3] РНК-зависимая РНК-полимераза катализирует синтез РНК, комплементарной данной РНК-матрице. Процесс репликации РНК включает в себя две стадии. На первой стадии (инициация) начинается синтез РНК на или вблизи 3′-конца РНК-матрицы по праймер-независимому (de novo), или праймер-зависимому механизму (в данном случае в качестве праймера используется вирусный геном-связанный белок (VPg; от англ. viral protein genome-linked). При инициации de novo происходит добавление нуклеозидтрифосфата (НТП) к 3′-OH концу первого, инициирующего нуклеозидтрифосфата. В течение последующей стадии (элонгация) происходит наращивание дочерней РНК в ходе последовательного переноса нуклеозидтрифосфатов, которое завершается созданием комплементарного исходной матрице РНК-продукта.[4][5]
Вирусные РНК-зависимые РНК-полимеразы были открыты в начале 1960-х годов в исследованиях группы Mengovirus и вируса полиомиелита. В ранних работах было отмечено, что данные вирусы не чувствительны к актиномицину D, препарату, ингибирующему клеточный ДНК-зависимый синтез РНК (транскрипция). В результате было высказано предположение, что у данных вирусов имеется специфический фермент, катализирующий синтез РНК и использующий непосредственно РНК в качестве матрицы (без формирования ДНК-матрицы).
Наиболее изученный фермент данного класса — это РНК-зависимая РНК-полимераза вируса полиомиелита. Геном вируса образован РНК, которая проникает в клетку путем эндоцитоза, опосредованного узнаванием специальных рецепторов на поверхности клетки. В цитоплазме клетки вирусная РНК может выступать в качестве матрицы для синтеза дочерней РНК. Образовавшиеся дочерние комплементарные цепи также могут служить матрицами для воспроизведения новых вирусных геномов. По окончании воспроизведения геномов происходит их упаковка и высвобождение из клетки. Высвободившиеся дочерние вирионы способны инфицировать новые клетки. Преимуществом данной схемы жизненного цикла является отсутствие стадии вирусной ДНК, благодаря чему репликация вируса осуществляется быстрее. Одновременно с этим отсутствие стадии ДНК может считаться и недостатком, поскольку делает невозможным воспроизведение новых вирионов при разрушении вирусной РНК.
Многие РНК-зависимые РНК-полимеразы тесно ассоциированы с мембранами, что значительно осложняет их выделение и изучение. Самые известные РНК-зависимые РНК-полимеразы — это: 3Dpol вируса полиомиелита, РНК-зависимая РНК-полимераза вируса везикулярного стоматита и NS5B вируса гепатита C.
Многие эукариоты тоже имеют РНК-зависимые РНК-полимеразы, участвующие в РНК-интерференции. У эукариот данные РНК-полимеразы амплифицируют микроРНК, малые временные РНК, образующие двухцепочечные РНК при участии малых интерферирующих РНК в качестве праймеров.[6] Интересно, что данные РНК-зависимые РНК-полимеразы могут использоваться вирусами с целью репликации собственного генетического материала.
РНК-зависимые РНК-полимеразы высоко консервативны среди вирусов и обладают значительной гомологией с теломеразой эукариот, хотя причина такой высокой консервативности у столь разнообразных организмов остается дискуссионной.[7] Схожесть аминокислотных последовательностей этих ферментов привела к предположениям о происхождении теломеразы от РНК-зависимой РНК-полимеразы вирусов, однако данное предположение остается спекулятивным и не имеет точных подтверждений.
Все РНК-зависимые РНК-полимеразы и многие ДНК-зависимые РНК-полимеразы имеют трехмерную организацию, которая по форме напоминает правую руку, при этом в ней можно выделить следующие домены: ладонь, четыре пальца и большой палец.[8] Только домен ладони, состоящий из четырех-тяжевого антипараллельного бета-листа и двух альфа-спиралей, консервативен у всех ферментов. В РНК-зависимых РНК-полимеразах, домен ладони включает три консервативных мотива (А, B и C). Мотив А (D-х(4,5)-D) и мотив С (GDD) пространственно сближены, а остатки аспарагиновой кислоты этих мотивов связывают Mg2+ и/или Mn2+. Остаток аспарагина в мотиве B участвует в различении рибонуклеоиздтрифосфатов от дезоксирибонуклеозидтрифосфатов, что обеспечивает синтез РНК, а не ДНК.[9] Доменная организация[10] и 3D-структуры каталитического центра широкого спектра РНК-зависимых РНК-полимераз, даже с низкой общей гомологией остаются консервативны. Каталитический центр формируется из нескольких мотивов, содержащих ряд консервативных аминокислотных остатков.
РНК-зависимые РНК-полимеразы можно обнаружить у следующих 4 групп РНК-содержащих вирусов (все РНК-содержащие вирусы без стадии ДНК):
- Вирусы, содержащие одноцепочечную (+) РНК ((+) ssRNA) или двунитевую РНК (dsRNA), за исключением ретровирусов и Birnaviridae. Сюда относятся семейства: Cystoviridae, Reoviridae, Hypoviridae, Partitiviridae, Totiviridae.
- Mononegavirales (вирусы с несегментированным геномом из одноцепочечной (-) РНК, (-) ssRNA)
- Вирусы с сегментированным геномом из одноцепочечной (-) РНК. К данной группе относятся ортомиксовирусы (в том числе вирусы гриппа А, В и С, Thogotoviruses и вирус анемии лосося), Arenaviruses, Bunyaviruses, Hantaviruses, Nairoviruses, Phleboviruses, Tenuiviruses и Tospoviruses.
- Семейство Birnaviridae геном которых представлен двуцепочечной РНК (dsRNA).
РНК-зависимые РНК-полимеразы в первом из вышеуказанных суперсемейств можно разделить на следующие три подгруппы:
- Одноцепочечные (+) РНК вирусы эукариот, без стадии ДНК
- РНК-содержащие бактериофаги. Можно выделить два семейства РНК-содержащих бактериофагов: Leviviridae ((+) ssRNA фаги) и Cystoviridae (dsRNA фаги)
- Семейство Reoviridae вирусов dsRNA.
У флавивирусов в одноцепочечном РНК геноме закодирован полипротеин, который затем расщепляется на ряд продуктов, одним из которых является NS5. Рекомбинантный белок NS5 из вирус денге типа 1, экспрессировавшийся в кишечной палочке обладает РНК-зависимой РНК-полимеразной активностью. Данная РНК-зависимая РНК-полимераза обладает несколькими регионами и мотивами, гомологичными другим РНК-зависимым РНК-полимеразам.[11]
- ↑ Akutsu, M; Ye, Y; Virdee, S; Chin, JW; Komander, D. Molecular basis for ubiquitin and ISG15 cross-reactivity in viral ovarian tumor domains (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2011. — February (vol. 108, no. 6). — P. 2228—2233. — DOI:10.1073/pnas.1015287108. — PMID 21266548.
- ↑ Koonin E.V., Gorbalenya A.E., Chumakov K.M. Tentative identification of RNA-dependent RNA polymerases of dsRNA viruses and their relationship to positive strand RNA viral polymerases (англ.) // FEBS Lett. (англ.)русск. : journal. — 1989. — July (vol. 252, no. 1—2). — P. 42—6. — DOI:10.1016/0014-5793(89)80886-5. — PMID 2759231.
- ↑ Zanotto P.M., Gibbs M.J., Gould E.A., Holmes E.C. A reevaluation of the higher taxonomy of viruses based on RNA polymerases (англ.) // J. Virol. (англ.)русск. : journal. — 1996. — September (vol. 70, no. 9). — P. 6083—6096. — PMID 8709232.
- ↑ Z; Jin; Leveque, V; Ma, H; Johnson, K. A.; Klumpp, K. Assembly, purification, and pre-steady-state kinetic analysis of active RNA-dependent RNA polymerase elongation complex (англ.) // Journal of Biological Chemistry : journal. — 2012. — Vol. 287, no. 13. — P. 10674—10683. — DOI:10.1074/jbc.M111.325530. — PMID 22303022.
- ↑ Kao C.C., Singh P., Ecker D.J. De novo initiation of viral RNA-dependent RNA synthesis (англ.) // Virology : journal. — 2001. — September (vol. 287, no. 2). — P. 251—260. — DOI:10.1006/viro.2001.1039. — PMID 11531403.
- ↑ Iyer L.M., Koonin E.V., Aravind L. Evolutionary connection between the catalytic subunits of DNA-dependent RNA polymerases and eukaryotic RNA-dependent RNA polymerases and the origin of RNA polymerases (англ.) // BMC Struct. Biol. (англ.)русск. : journal. — 2003. — January (vol. 3). — P. 1. — DOI:10.1186/1472-6807-3-1. — PMID 12553882.
- ↑ Suttle C.A. Viruses in the sea (англ.) // Nature. — 2005. — September (vol. 437, no. 7057). — P. 356—361. — DOI:10.1038/nature04160. — PMID 16163346.
- ↑ Structure of the RNA-dependent RNA polymerase of poliovirus (англ.) // Structure : journal. — 1997. — August (vol. 5, no. 8). — P. 1109—1122. — DOI:10.1016/S0969-2126(97)00261-X. — PMID 9309225.
- ↑ Poliovirus RNA-dependent RNA polymerase (3Dpol): structural, biochemical, and biological analysis of conserved structural motifs A and B (англ.) // J. Biol. Chem. : journal. — 2000. — August (vol. 275, no. 33). — P. 25523—25532. — DOI:10.1074/jbc.M002671200. — PMID 10827187.
- ↑ Analysis of RNA-dependent RNA polymerase structure and function as guided by known polymerase structures and computer predictions of secondary structure (англ.) // Virology : journal. — 1998. — December (vol. 252, no. 2). — P. 287—303. — DOI:10.1006/viro.1998.9463. — PMID 9878607.
- ↑ Recombinant dengue type 1 virus NS5 protein expressed in Escherichia coli exhibits RNA-dependent RNA polymerase activity (англ.) // Virology : journal. — 1996. — February (vol. 216, no. 2). — P. 317—325. — DOI:10.1006/viro.1996.0067. — PMID 8607261.
Эта статья использует текст из общественное достояние Pfam и InterPro IPR000208
