Антисмысловые РНК — Википедия
Антисмысловые РНК комплементарны мРНК, потому что считываются с того же гена, но с другой цепиАнтисмысловы́е РНК (англ. Antisense RNA) — одноцепочечные РНК, которые комплементарны мРНК, транскрибируемой в клетке, или гену-мишени. Механизмы действия антисмысловых РНК весьма разнообразны, они могут как подавлять, так и активировать экспрессию гена-мишени. Природные антисмысловые РНК есть и у прокариот, и у эукариот[1]; они относятся к длинным некодирующим РНК как РНК длиной более 200 нуклеотидов[2]. Синтетические антисмысловые РНК нашли широкое применение у исследователей в качестве инструмента для нокдауна генов. Антисмысловые РНК также находят медицинское применение[3][1][2].
Первые антисмысловые РНК были открыты при изучении функциональных белков. Например, при изучении порина[en] внешней мембраны[en] ompC кишечной палочки Escherichia coli было показано, что некоторые промоторы этого гена могут подавлять экспрессию других мембранных поринов, таких как ompF. Оказалось, что собственно в подавлении участвует участок длиной 300 нуклеотидов, расположенный выше промотора ompC. Он на 70 % гомологичен последовательности 5′-концевой части мРНК ompF, и считанный с него транскрипт (micF) может связываться с ней за счёт комплементарного взаимодействия. Оказалось, что в условиях стресса происходит экспрессия micF, который формирует дуплекс с мРНК ompF, вызывая её деградацию
В отличие от micF, открытого случайно, большинство антисмысловых РНК были открыты входе целенаправленного полногеномного поиска малых регуляторных РНК и анализа транскриптома. Однако большинство алгоритмов анализа рассматривают только межгенные участки, и антисмысловые РНК, считанные с цепи ДНК, противоположной матричной (кодирующей цепи) в области кодирующей области, остаются неучтёнными. Для обнаружения таких антисмысловых РНК можно использовать олигонуклеотидные чипы. Для того чтобы минимизировать количество ложноположительных результатов, в последнее время активно разрабатываются методы изучения транскрипции, специфичной для одной из цепей, некодирующих РНК, взаимодействующих с хроматином, и РНК одиночных клеток [en][1].
Идея об использовании антисмысловых РНК в медицине впервые была высказана в 1978 году, когда Zamecnik и Stephenson получили антисмысловые олигонуклеотиды, комплементарные РНК вируса саркомы Рауса[en]. Оказалось, что они подавляли вирусную репликацию и синтез вирусных белков. С тех пор на разработку медицинского применения антисмысловых РНК были брошены колоссальные усилия. Первый препарат такого рода, фомивирсен, был одобрен FDA в 1998 году. Он был предназначен для лечения цитомегаловирусного ретинита у пациентов со СПИДом. Однако в 2004 году препарат был снят с производства из-за потери рынка[5].
Первые антисмысловые РНК были обнаружены в бактериальных клетках. Они кодировались плазмидами, геномами самих бактерий и бактериофагов. Например, антисмысловая РНК, известная как РНК I, считывается с плазмиды ColE1 и играет важную роль в определении числа копий плазмид и контролирует их репликацию. Для репликации плазмиды ColE1, кроме того, необходима другая антисмысловая РНК (РНК II). Она гибридизуется с ДНК и выступает в роли праймера при репликации, а после разрушается РНКазой H
Антисмысловые РНК можно классифицировать по-разному. Некоторые авторы подразделяют их на антисмысловые РНК, взаимодействующие с РНК, с ДНК и с белками[8]. Их также можно классифицировать по типу промотора, с которого начинается их экспрессия: независимые промоторы, двунаправленные промоторы или криптические промоторы. Фактором классификации антисмысловых РНК также может выступать длина. Хотя большинство антисмысловых РНК имеют длину более 200 нуклеотидов и, следовательно, относятся к длинным некодирующим РНК, некоторые из них существенно короче. Поскольку большинство механизмов действия антисмысловых РНК специфичны для конкретных биологических видов, их можно разделять и по видам. Однако наиболее распространённая классификация антисмысловых РНК построена на том, как транскрипция этих РНК соотносится с их генами-мишенями
Цис-регуляторные антисмысловые РНК транскрибируются в том же локусе, что и мРНК-мишень, но с противоположной цепи ДНК. По этой причине они нередко почти полностью комплементарны гену-мишени. Если действие цис-регуляторных антисмысловых РНК связано с мРНК, то мишенями одной антисмысловой РНК может быть только одна мРНК. Действие таких антисмысловых РНК на мРНК выражается в блокировании доступа рибосомы к мРНК или в привлечении РНКазы, разрушающей мРНК[4]. Среди цис-регуляторных антисмысловых РНК есть также эпигенетические сайленсеры и активаторы. Они могут вызывать эпигенетические изменения в локусе, с которого они были транскрибированы. Они могут привлекать к локусу-мишени белки, модифицирующие хроматин, что может сказаться на экспрессии не только гена-мишени, но и соседних генов [8].
Транс-регуляторные антисмысловые РНК транскрибируются с локусов, удалённых от генов-мишеней. В отличие от цис-регуляторных РНК, они слабо комплементарны гену-мишени, однако могут быть длиннее цис-регуляторных РНК. Их мишенями также могут быть несколько локусов. Из-за неполной комплементарности комплексы транс-регуляторных антисмысловых РНК с транскриптами-мишенями не очень стабильны, и для функционирования таких РНК нередко нужны РНК-шапероны. Из-за сложности действия транс-регуляторные РНК почти не рассматривают в качестве потенциальных мишеней лекарственных препаратов[4].
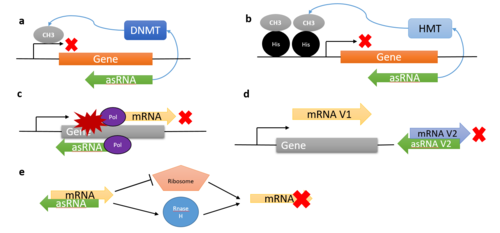 Некоторые механизмы действия антисмысловых РНК. а) Антисмысловые РНК индуцируют метилирование ДНК, привлекая ДНК-метилтрансферазы к гену-мишени; b) Антисмысловые РНК вызывают метилирование гистонов, привлекая гистонметилтрансферазы
Некоторые механизмы действия антисмысловых РНК. а) Антисмысловые РНК индуцируют метилирование ДНК, привлекая ДНК-метилтрансферазы к гену-мишени; b) Антисмысловые РНК вызывают метилирование гистонов, привлекая гистонметилтрансферазыМногие антисмысловые РНК подавляют транскрипцию генов-мишеней при помощи эпигенетических механизмов. Например, они могут вызывать метилирование ДНК, что нередко связано с болезнями. Так, при альфа-талассемии[en] экспрессия гена гемоглобина α1 (HBA1[en]) подавляется ненормальным транскриптом гена LUC71. Он выступает в роли антисмысловой РНК и вызывает метилирование промотора HBA1. При острой лимфобластной лейкемии и острой миелоидной лейкемии происходит сайленсинг гена-супрессора опухолей p15INK4b. Он вызван антисмысловой РНК ANRIL [en], которая считывается с того же локуса, что и p15INK4b[8]. Антисмысловые РНК также могут вызывать метилирование гистонов, что может приводить как к репрессии, так и к активации генов[9]. Например, ANRIL не только запускает метилирование ДНК, но и репрессирует соседние гены CDKN2B[en] и CDKN2A[en], привлекая к ним PRC2, вносящий репрессивную эпигенетическую метку (h4K27me). Ещё один пример — инактивация X-хромосомы у млекопитающих антисмысловой РНК Xist[1]. Вызываемые антисмысловыми РНК изменения хроматина могут быть и транс-регуляторными. Например, у млекопитающих антисмысловая РНК HOTAIR[en] считывается с гена гомеобокса C (HOXC), но привлекает PRC2 к HOXD. HOTAIR активно экспрессируется в клетках первичного рака груди[1].
Антисмысловые РНК могут включаться в регуляцию экспрессии гена не только на этапе инициации транскрипции, но и на её элонгации и терминации. Например, у прокариот и эукариот иногда имеет место двунаправленная транскрипция, когда две РНК-полимеразы считывают один и тот же ген с противоположных цепей. В конце концов они сталкиваются, что приводит к преждевременной терминации транскрипции. Даже если полимеразы движутся медленно и их столкновение маловероятно, они могут остановиться, преждевременно обрывая элонгацию транскрипции, что также приводит к сайленсингу гена. По такому механизму ген IME4 репрессируется антисмысловой РНК RME2. Антисмысловые РНК также могут нарушать сплайсинг. Так, для трансляции мРНК гена ZEB2
Антисмысловые РНК могут действовать посттранскрипционно, а именно непосредственно влиять на трансляцию мРНК-мишени. Они могут блокировать доступ рибосомы к мРНК или привлекать к мРНК РНКазу H. Иногда, впрочем, антисмысловые РНК оказывают позитивный эффект на трансляцию[1].
Как регуляторные элементы, действующие очень специфично, антисмысловые РНК могут быть перспективными мишенями лекарственных препаратов. Кроме того, для достижения нужного эффекта нужно совсем немного молекул антисмысловых РНК, что делает их ещё более удобными мишенями. Активно прорабатывается идея о специфичном повышении экспрессии гена-мишени за счёт подавления соответствующей антисмысловой РНК. Из-за особенностей дизайна препаратов[en] создать ингибитор гораздо проще, чем активатор. Однако в некоторых случаях бывает необходимым именно повысить экспрессию гена-мишени, например, гена-супрессора опухолей. Этого можно добиться, подавив работу соответствующей ему антисмысловой РНК. Кроме того, в тех случаях, когда требуется подавление гена-мишени, сами антисмысловые РНК могут выступать в роли терапевтического агента. Однако введённые в клетку РНК будут быстро разрушены РНКазами, поэтому необходимо защитить их от деградации. Чаще всего это достигается при помощи химических модификаций, например, присоединения фосфоротиоата
В качестве используемого в данный момент препарата, представляющего собой антисмысловую РНК, можно привести мипомерсен[en], одобренный FDA в 2013 году. Он был разработан для регуляции уровня холестеринсодержащих липопротеинов низкой плотности в крови пациентов, страдающих от гомозиготной семейной гиперхолистеринемии[en] — редкого аутосомно-доминантного генетического заболевания[10].
- ↑ 1 2 3 4 5 6 7 8 Pelechano V., Steinmetz L. M. Gene regulation by antisense transcription. (англ.) // Nature Reviews. Genetics. — 2013. — December (vol. 14, no. 12). — P. 880—893. — DOI:10.1038/nrg3594. — PMID 24217315. [исправить]
- ↑ 1 2 3 Wahlestedt C. Targeting long non-coding RNA to therapeutically upregulate gene expression. (англ.) // Nature Reviews. Drug Discovery. — 2013. — June (vol. 12, no. 6). — P. 433—446. — DOI:10.1038/nrd4018. — PMID 23722346. [исправить]
- ↑ Weiss B., Davidkova G., Zhou L. W. Antisense RNA gene therapy for studying and modulating biological processes. (англ.) // Cellular And Molecular Life Sciences : CMLS. — 1999. — March (vol. 55, no. 3). — P. 334—358. — DOI:10.1007/s000180050296. — PMID 10228554. [исправить]
- ↑ 1 2 3 Saberi F., Kamali M., Najafi A., Yazdanparast A., Moghaddam M. M. Natural antisense RNAs as mRNA regulatory elements in bacteria: a review on function and applications. (англ.) // Cellular & Molecular Biology Letters. — 2016. — Vol. 21. — P. 6—6. — DOI:10.1186/s11658-016-0007-z. — PMID 28536609. [исправить]
- ↑ Kole R., Krainer A. R., Altman S. RNA therapeutics: beyond RNA interference and antisense oligonucleotides. (англ.) // Nature Reviews. Drug Discovery. — 2012. — 20 January (vol. 11, no. 2). — P. 125—140. — DOI:10.1038/nrd3625. — PMID 22262036. [исправить]
- ↑ Simons R. W. Naturally occurring antisense RNA control—a brief review. (англ.) // Gene. — 1988. — 10 December (vol. 72, no. 1-2). — P. 35—44. — PMID 2468573. [исправить]
- ↑ Ietswaart R., Wu Z., Dean C. Flowering time control: another window to the connection between antisense RNA and chromatin. (англ.) // Trends In Genetics : TIG. — 2012. — September (vol. 28, no. 9). — P. 445—453. — DOI:10.1016/j.tig.2012.06.002. — PMID 22785023. [исправить]
- ↑ 1 2 3 4 Magistri M., Faghihi M. A., St Laurent G 3rd., Wahlestedt C. Regulation of chromatin structure by long noncoding RNAs: focus on natural antisense transcripts. (англ.) // Trends In Genetics : TIG. — 2012. — August (vol. 28, no. 8). — P. 389—396. — DOI:10.1016/j.tig.2012.03.013. — PMID 22541732. [исправить]
- ↑ Whetstine, Johnathan R. Histone Methylation // Handbook of Cell Signaling (неопр.). — Second. — С. 2389—2397. — ISBN 978-0-12-374148-6. — DOI:10.1016/b978-0-12-374145-5.00287-4.
- ↑ Wong E., Goldberg T. Mipomersen (kynamro): a novel antisense oligonucleotide inhibitor for the management of homozygous familial hypercholesterolemia. (англ.) // P & T : A Peer-reviewed Journal For Formulary Management. — 2014. — February (vol. 39, no. 2). — P. 119—122. — PMID 24669178. [исправить]
Гидовая РНК — Википедия
Материал из Википедии — свободной энциклопедии
Гидовая РНК, гРНК — это РНК, которые опосредуют вставки уридиновых нуклеотидов в мРНК у трипаносом в процессе редактирования РНК.[1][2][3] гРНК закодированы отдельными районами генома кинетопласта.[4]
5′-конец гРНК гибридизуется с коротким районом нередактированной пре-мРНК, называемым якорной последовательностью, в то время как 3′-конец таких РНК является матрицей для редактирования. Многие гРНК не гибридизуются с якорной последовательностью первичного транскрипта, но гибридизуются с частично редактированным транскриптом. Поэтому редактирование пре-мРНК трипаносом начинается обычно около 3′-конца и продвигается к 5′-концу в ходе повторяющегося процесса, который требует наличия нескольких разных гРНК, которые последовательно связываются с якорной последовательностью частично редактированных участков. Остается не совсем понятным, для чего кинетопласты трипаносом используют столь усложенный механизм образования мРНК. Функции многих гРНК не изучены.[5]
- ↑ Stuart KD, Schnaufer A, Ernst NL, Panigrahi AK. Complex management: RNA editing in trypanosomes // Trends Biochem Sci. — 2005. — Т. 30, № 2. — С. 97-105.
- ↑ Madison-Antenucci S, Grams J, Hajduk SL. Editing machines: the complexities of trypanosome RNA editing // Cell. — 2002. — Т. 108, № 4. — С. 435-438.
- ↑ Stuart K, Allen TE, Kable ML, Lawson S. Kinetoplastid RNA editing: complexes and catalysts // Curr Opin Chem Biol. — 1997. — Т. 1, № 3. — С. 340-346.
- ↑ Uliel S, Liang XH, Unger R, Michaeli S. Small nucleolar RNAs that guide modification in trypanosomatids: repertoire, targets, genome organisation, and unique functions // Int J Parasitol. — 2004. — Т. 34, № 4. — С. 445-454.
- ↑ Stuart K, Panigrahi AK. RNA editing: complexity and complications // Mol Microbiol. — 2002. — Т. 45, № 3. — С. 591-596.
- Gurbachan S. Miglani. RNAs: Their Processing and Function // Advanced Genetics. — CRC Press, 2002. — С. 257—259. — 665 с. — ISBN 0849324254.
- Bruce Alberts. RNA Editing Can Change the Meaning of the RNA Message // Molecular Biology of the Cell. — Garland Science, 2007. — С. 483—484. — 1601 с. — ISBN 0815341113.
- Simpson L, Thiemann OH, Savill NJ, Alfonzo JD, Maslov DA. Evolution of RNA editing in trypanosome mitochondria // Proc Natl Acad Sci U S A. — 2000. — Т. 97, № 13. — С. 6986-6993. — Обзорная статья об эволюции редактирования РНК у Трипаносом на английском языке, находится в свободном доступе
Редактирование РНК
Генетический код — Википедия
Схема генетического кода
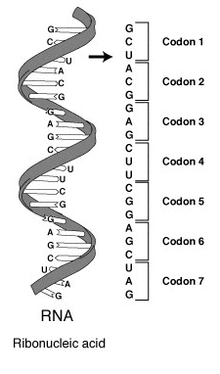 Последовательность кодонов в части молекулы мРНК. Каждый кодон состоит из трёх нуклеотидов и соответствует единственной аминокислоте. Рибосома синтезирует белок согласно инструкции, записанной в мРНК, причём соответствие между кодоном и аминокислотой определяется генетическим кодом
Последовательность кодонов в части молекулы мРНК. Каждый кодон состоит из трёх нуклеотидов и соответствует единственной аминокислоте. Рибосома синтезирует белок согласно инструкции, записанной в мРНК, причём соответствие между кодоном и аминокислотой определяется генетическим кодомГенети́ческий код (англ. Genetic code) — совокупность правил, согласно которым в живых клетках последовательность нуклеотидов (ген и мРНК) переводится в последовательность аминокислот (белок). Собственно перевод (трансляцию) осуществляет рибосома, которая соединяет аминокислоты в цепочку согласно инструкции, записанной в кодонах мРНК. Соответствующие аминокислоты доставляются в рибосому молекулами тРНК. Генетический код всех живых организмов Земли един (имеются лишь незначительные вариации), что свидетельствует о наличии общего предка.
Правила генетического кода определяют, какой аминокислоте соответствует триплет (три подряд идущих нуклеотида) в мРНК. За редкими исключениями[1], каждому кодону соответствует только одна аминокислота. Конкретная аминокислота может кодироваться более чем одним кодоном, есть также кодоны, означающие начало и конец белка. Вариант генетического кода, который используется подавляющим большинством живых организмов, называют стандартным, или каноническим, генетическим кодом. Однако известно несколько десятков исключений из стандартного генетического кода, например, при трансляции в митохондриях используются несколько изменённые правила генетического кода.
Простейшим представлением генетического кода может служить таблица из 64 ячеек, в которой каждая ячейка соответствует одному из 64 возможных кодонов[2].
 Маршалл Ниренберг, начавший расшифровку генетического кода
Маршалл Ниренберг, начавший расшифровку генетического кодаПопытки понять, каким образом последовательность ДНК кодирует аминокислотную последовательность белков, начали предприниматься почти сразу же после того, как в 1953 году была установлена структура ДНК (двойная спираль[en]). Георгий Гамов предположил, что кодоны должны состоять из трёх нуклеотидов, чтобы кодонов хватило для всех 20 аминокислот (всего же возможно 64 различных кодона из трёх нуклеотидов: на каждую из трёх позиций можно поставить один из четырёх нуклеотидов)[3].
В 1961 году триплетность генетического кода удалось подтвердить экспериментально. В том же году Маршалл Ниренберг и его коллега Генрих Маттеи[en] использовали бесклеточную систему для трансляции in vitro. В качестве матрицы был взят олигонуклеотид, состоящий из остатков урацила (UUUU…). Пептид, синтезированный с него, содержал только аминокислоту фенилаланин[4]. Так впервые было установлено значение кодона: кодон UUU кодирует фенилаланин. Дальнейшие правила соответствия между кодонами и аминокислотами были установлены в лаборатории Северо Очоа. Было показано, что полиадениновая РНК (ААА…) транслируется в полилизиновый пептид[5], а на матрице полицитозиновой РНК (ССС…) синтезируется пептид, состоящий только из остатков пролина[6]. Значение остальных кодонов было установлено при помощи разнообразных сополимеров в ходе экспериментов, проведённых в лаборатории Хара Гобинда Кораны. Вскоре после этого Роберт Холли установил структуру молекулы тРНК, которая служит посредником при трансляции. В 1968 году Ниренберг, Корана и Холли были удостоены Нобелевской премии по физиологии и медицине[7].
После установления правил генетического кода многие учёные занялись его искусственными преобразованиями[en]. Так, начиная с 2001 года в генетический код были внедрены 40 аминокислот, которые в природе не входят в состав белков. Для каждой аминокислоты создавались свой кодон и соответствующая аминоацил-тРНК-синтетаза. Искусственное расширение генетического кода и создание белков с новыми аминокислотами могут помочь глубже изучить структуру белковых молекул, а также получить искусственные белки с заданными свойствами[8][9]. Х. Мураками и М. Сисидо смогли превратить некоторые кодоны из трёхнуклеотидных в четырёх- и пятинуклеотидные. Стивен Бреннер получил 65-й кодон, который был функционален in vivo[10].
В 2015 году у бактерии Escherichia coli удалось изменить значение всех кодонов UGG с триптофана на тиенопиррол-аланин, не встречающийся в природе[11]. В 2016 году был получен первый полусинтетический организм — бактерия, геном которой содержал два искусственных азотистых основания (X и Y), сохраняющихся при делении[12][13]. В 2017 году исследователи из Южной Кореи заявили о создании мыши с расширенным генетическим кодом, способной синтезировать белки с аминокислотами, не встречающимися в природе[14].
Рамки считывания в митохондриальной ДНК человека в области генов MT-ATP8[en] и MT-ATP6[en] (чёрным цветом показан участок между позициями 8525 и 8580). В направлении считывания 5′ → 3′ возможны три рамки считывания, начинающиеся с первой (+1), второй (+2) и третьей позиций (+3). Соответствие между кодонами (кодоны выделены квадратными скобками) определяется митохондриальным генетическим кодом позвоночных[en] в рамке +1 для MT-ATP8 (показана красным) или в рамке +3 для MT-ATP6 (показана синим). Ген MT-ATP8 заканчивается стоп-кодоном TAG (отмечен красной точкой) в рамке +1. Ген MT-ATP6 начинается с кодона ATG (кодирует аминокислоту метионин (М) и выделен синим кружком) в рамке +3Рамка считывания[править | править код]
Гены кодируются в направлении 5’→3′ нуклеотидной последовательности[15]. Рамка считывания определяется самым первым триплетом, с которого начинается трансляция. Последовательность неперекрывающихся кодонов, начинающуюся со старт-кодона и заканчивающуюся стоп-кодоном, называют открытой рамкой считывания. Например, последовательность 5′-AAATGAACG-3′ (см. рис.) при чтении с первого нуклеотида разбивается на кодоны AAA, TGA и ACG. Если чтение начинается со второго нуклеотида, то ей соответствуют кодоны AAT и GAA. Наконец, при чтении с третьего нуклеотида используются кодоны ATG и AAC. Таким образом, любую последовательность можно прочесть в направлении 5′ → 3′ тремя разными способами (с тремя разными рамками считывания), причём в каждом случае последовательность белкового продукта будет отличаться из-за распознавания рибосомой разных кодонов. Если учесть, что ДНК имеет двуцепочечную структуру, то возможны 6 рамок считывания: три на одной цепи и три на другой[16]. Однако считывание генов с ДНК не является случайным. Все другие рамки считывания в пределах одного гена обычно содержат многочисленные стоп-кодоны, чтобы быстро остановить и уменьшить метаболическую стоимость неправильного синтеза[17].
Старт- и стоп-кодоны[править | править код]
Трансляция информации с последовательности мРНК в аминокислотную последовательность начинается с так называемого старт-кодона — как правило, AUG, причём у эукариот он читается как метионин, а у бактерий — как формилметионин. Одного старт-кодона недостаточно для запуска трансляции; для него необходимы факторы инициации трансляции, а также особые элементы в соседних последовательностях, например, последовательность Шайна — Дальгарно у бактерий. У некоторых организмов в роли старт-кодонов используются кодоны GUG, который в норме кодирует валин, и UUG, который в стандартном коде соответствует лейцину[18].
После инициационного кодона трансляция продолжается через последовательное считывание кодонов и присоединение аминокислот друг к другу рибосомой до достижения сигнала к прекращению трансляции — стоп-кодона. Существуют три стоп-кодона, каждый из которых имеет своё название: UAG (янтарь), UGA (опал) и UAA (охра). Стоп-кодоны также называют терминаторными. В клетках нет тРНК, соответствующих стоп-кодонам, поэтому, когда рибосома доходит до стоп-кодона, вместо тРНК с ним взаимодействуют факторы терминации трансляции, которые гидролизуют последнюю тРНК от аминокислотной цепочки, а затем заставляют рибосому диссоциировать[19]. У бактерий в терминации трансляции принимают участие три белковых фактора[en]: RF-1, RF-2 и RF-3: RF-1 узнаёт кодоны UAG и UAA, а RF-2 распознаёт UAA и UGA. Фактор RF-3 выполняет вспомогательную работу. Трёхмерная структура RF-1 и RF-2 напоминает формой и распределением заряда тРНК и, таким образом, представляет собой пример молекулярной мимикрии[en][20]. У эукариот фактор терминации трансляции eRF1 распознаёт все три стоп-кодона. Зависимая от рибосомы ГТФаза eRF3, которую рассматривают как второй фактор терминации трансляции эукариот, помогает eRF1 в высвобождении с рибосомы готового полипептида[21][22][23].
Распределение стоп-кодонов в геноме организма неслучайно и может быть связано с GC-составом генома[24][25]. Например, у штамма E. coli K-12 в геноме имеется 2705 кодонов TAA (63 %), 1257 TGA (29 %) и 326 TAG (8 %) при GC-составе 50,8 %[26]. Масштабное исследование геномов разных видов бактерий показало, что доля кодона TAA положительно коррелирует с GC-составом, а доля TGA — отрицательно. Частота самого редко используемого стоп-кодона, TAG, не связана с GC-составом[27]. Сила стоп-кодонов также неодинакова. Спонтанный обрыв трансляции чаще всего происходит на кодоне UGA, а на UAA — реже всего[23].
Помимо собственно стоп-кодона, важнейшее значение для терминации трансляции имеет его окружение. Наиболее велика роль нуклеотида, расположенного сразу за стоп-кодоном (+4). Вероятно, нуклеотид +4 и другие нуклеотиды, следующие за ним, влияют на терминацию трансляции, обеспечивая сайты связывания факторов терминации трансляции. По этой причине некоторые исследователи предлагают рассматривать четырёхнуклеотидный стоп-сигнал вместо трёхнуклеотидного стоп-кодона. Нуклеотиды, расположенные выше стоп-кодонов, также влияют на трансляцию. Например, для дрожжей было показано, что аденин, располагающийся на 2 позиции выше первого нуклеотида стоп-кодона, стимулирует обрыв трансляции на стоп-кодоне UAG (возможно, и на остальных кодонах)[23].
Иногда стоп-кодоны выступают в роли смысловых. Например, кодон UGA кодирует нестандартную аминокислоту селеноцистеин, если рядом с ним в транскрипте находится так называемый SECIS-элемент[28]. Стоп-кодон UAG может кодировать другую нестандартную аминокислоту — пирролизин. Иногда стоп-кодон распознаётся как смысловой при мутациях, затрагивающих тРНК. Наиболее часто это явление наблюдается у вирусов, но оно также описано у бактерий, дрожжей, дрозофилы и человека, у которых играет регуляторную роль[29][30].
Генетический код и мутации[править | править код]
В ходе репликации ДНК изредка возникают ошибки при синтезе дочерней цепи. Эти ошибки, называемые мутациями, могут повлиять на фенотип организма, особенно если они затрагивают кодирующую область гена. Ошибки происходят с частотой 1 на каждые 10—100 миллионов пар оснований (п. о.), так как ДНК-полимеразы могут эффективно исправлять свои ошибки[31][32].
Под точечными мутациями понимают единичные замены одного азотистого основания. Если новое основание относится к тому же классу, что и исходное (оба пурины или оба пиримидины), то мутацию относят к транзициям. Если происходит замена пурина на пиримидин или пиримидина на пурин, то говорят о трансверсиях[en]. Транзиции встречаются чаще трансверсий[33]. Примерами точечных мутаций являются миссенс- и нонсенс-мутации. Они могут вызывать такие заболевания, как серповидноклеточная анемия и талассемия соответственно[34][35]. Клинически значимые миссенс-мутации приводят к замене аминокислотного остатка на остаток с другими физико-химическими свойствами, а нонсенс-мутации заключаются в появлении преждевременного стоп-кодона[16].
Мутации, при которых нарушается правильная рамка считывания из-за вставок и делеций (в совокупности они называются инделами[en]), содержащих некратное трём число нуклеотидов, называются мутациями сдвига рамки считывания. При этих мутациях белковый продукт получается совершенно иной, чем в диком типе. Как правило, при сдвигах рамки считывания появляются преждевременные стоп-кодоны, которые вызывают образование усечённых белков[36]. Поскольку эти мутации значительно нарушают функцию белка, они довольно редко закрепляются отбором: нередко отсутствие белка приводит к гибели организма ещё до рождения[37]. Мутации сдвига рамки считывания связаны с такими заболеваниями, как болезнь Тея — Сакса[38].
Хотя подавляющее число мутаций вредны или нейтральны[en], некоторые оказываются полезными[39]. Они могут давать организму лучшую приспособленность по сравнению с диким типом к определённым условиям окружающей среды или дают ему возможность размножаться быстрее особей дикого типа. В этом случае мутация будет постепенно распространяться в популяции в ходе нейтрального отбора[40]. Вирусы, геномы которых представлены РНК, мутируют очень быстро[41], что нередко приносит им пользу, потому что иммунная система, эффективно распознающая одни варианты вирусных антигенов, оказывается бессильна против слегка изменённых[42]. В больших популяциях организмов, размножающихся бесполым путём, например, E. coli, одновременно может происходить несколько полезных мутаций. Этот феномен получил название клональной интерференции[en] и вызывает конкуренцию между мутациями[43].
Вырожденность[править | править код]
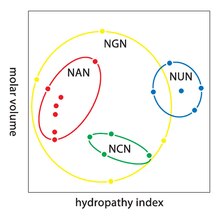
Способность разных кодонов кодировать одну аминокислоту называется вырожденностью кода. Впервые генетический код назвали вырожденным[en] Ниренберг и Бернфилд. Однако, несмотря на вырожденность, в генетическом коде полностью отсутствует двусмысленность. Например, кодоны GAA и GAG оба кодируют глутамат, но ни один из них не кодирует одновременно ещё какую-то аминокислоту. Кодоны, соответствующие одной аминокислоте, могут различаться по любым позициям, однако чаще всего две первые позиции у таких кодонов совпадают, а различается только последняя. Благодаря этому мутация, затронувшая третью позицию кодона, скорее всего, не скажется на белковом продукте[44].
Эта особенность может быть объяснена гипотезой неоднозначной пары оснований, предложенной Франсисом Криком. Согласно этой гипотезе, третий нуклеотид в кодоне ДНК может быть не полностью комплементарен антикодону тРНК для компенсации несоответствия числа типов тРНК числу кодонов[45][46].
Кодоны близких по физико-химическим свойствам аминокислот также нередко похожи, благодаря чему мутации не приводят к значительным нарушениям белковой структуры. Так, кодоны NUN (N — любой нуклеотид) обычно кодируют гидрофобные аминокислоты. NCN кодируют маленькие аминокислоты с умеренной гидрофобностью, а NAN кодируют гидрофильные аминокислоты среднего размера. Генетический код устроен настолько оптимально с точки зрения гидрофобности, что математический анализ при помощи сингулярного разложения 12 переменных (4 нуклеотида на 3 позиции) даёт значимую корреляцию (0,95) для предсказания гидрофобности аминокислоты по её кодону[47]. На восемь аминокислот мутации по третьим позициям не влияют вообще, а мутации по второй позиции, как правило, приводят к замене на аминокислоту с совершенно другими физико-химическими свойствами. Однако наибольшее влияние на белковый продукт имеют мутации по первым позициям. Так, мутации, приводящие к замене заряженной аминокислоты на аминокислоту с противоположным зарядом, могут затрагивать только первую позицию, а вторую — никогда. Такая замена заряда, вероятнее всего, окажет сильный эффект на структуру белка[48].
В таблице ниже представлен генетический код, общий для большинства про- и эукариот. В таблице приведены все 64 кодона и указаны соответствующие аминокислоты. Порядок оснований — от 5′- к 3′-концу мРНК. Приведены трёхбуквенные и однобуквенные обозначения аминокислот.
| неполярный | полярный | основный | кислотный | (стоп-кодон) |
- A Кодон AUG кодирует метионин и одновременно является сайтом инициации трансляции: первый кодон AUG в кодирующей области мРНК служит началом синтеза белка[49].
| Ala/A | GCU, GCC, GCA, GCG | Leu/L | UUA, UUG, CUU, CUC, CUA, CUG |
|---|---|---|---|
| Arg/R | CGU, CGC, CGA, CGG, AGA, AGG | Lys/K | AAA, AAG |
| Asn/N | AAU, AAC | Met/M | AUG |
| Asp/D | GAU, GAC | Phe/F | UUU, UUC |
| Cys/C | UGU, UGC | Pro/P | CCU, CCC, CCA, CCG |
| Gln/Q | CAA, CAG | Ser/S | UCU, UCC, UCA, UCG, AGU, AGC |
| Glu/E | GAA, GAG | Thr/T | ACU, ACC, ACA, ACG |
| Gly/G | GGU, GGC, GGA, GGG | Trp/W | UGG |
| His/H | CAU, CAC | Tyr/Y | UAU, UAC |
| Ile/I | AUU, AUC, AUA | Val/V | GUU, GUC, GUA, GUG |
| START | AUG | STOP | UAG, UGA, UAA |
Нестандартные аминокислоты[править | править код]
В некоторых белках нестандартные аминокислоты кодируются стоп-кодонами в зависимости от наличия особой сигнальной последовательности в мРНК. Например, стоп-кодон UGA может кодировать селеноцистеин, а UAG — пирролизин. Селеноцистеин и пирролизин рассматривают как 21-ю и 22-ю протеиногенную аминокислоту соответственно. В отличие от селеноцистеина, у пирролизина есть собственная аминоацил-тРНК-синтетаза[50]. Хотя обычно генетический код, используемый клетками одного организма, фиксирован, архея Acetohalobium arabaticum[en] может переключаться с 20-аминокислотного кода на 21-аминокислотный (включая пирролизин) при разных условиях роста[51].
Вариации[править | править код]
 Схема митохондриального генетического кода Globobulimina pseudospinescens. Показаны 64 кодона (слева направо), предсказанные отклонения от стандартного генетического кода изображены красным. Красной линией показаны стоп-кодоны. Высота буквы, соответствующей аминокислоте, пропорциональна частоте соответствия этой аминокислоты данному кодону
Схема митохондриального генетического кода Globobulimina pseudospinescens. Показаны 64 кодона (слева направо), предсказанные отклонения от стандартного генетического кода изображены красным. Красной линией показаны стоп-кодоны. Высота буквы, соответствующей аминокислоте, пропорциональна частоте соответствия этой аминокислоты данному кодонуСуществование отклонений от стандартного генетического кода предсказывалось ещё в 1970-х[52]. Первое отклонение было описано в 1979 году в митохондриях человека[53]. Впоследствии было описано ещё несколько альтернативных генетических кодов, слегка отличающихся от стандартного, в том числе альтернативные митохондриальные коды[54].
Например, у бактерий рода Mycoplasma стоп-кодон UGA кодирует триптофан, а у дрожжей из так называемой «CTG-клады» (в том числе патогенного вида Candida albicans) кодон CUG кодирует серин, а не лейцин, как в стандартном генетическом коде[55][56][57]. Поскольку вирусы используют тот же генетический код, что и клетки-хозяева, отклонения от стандартного генетического кода могут нарушить размножение вирусов[58]. Впрочем, некоторые вирусы, например, вирусы рода Totivirus[en], используют тот же альтернативный генетический код, что и организм-хозяин[59].
У бактерий и архей GUG и UUG нередко выступают старт-кодонами[60]. Некоторые отклонения от стандартного генетического кода есть и в ядерном геноме человека: так, в 4 % мРНК фермента малатдегидрогеназы один из стоп-кодонов кодирует триптофан или аргинин[61]. Значение стоп-кодона зависит от его окружения[30]. Отклонения в генетическом коде организма можно обнаружить, если найти в его геноме очень консервативные гены и сравнить их кодоны с соответствующими аминокислотами гомологичных белков близкородственных организмов. По такому принципу работает программа FACIL, которая рассчитывает, с какой частотой каждый кодон соответствует той или иной аминокислоте, а также определяет поддержку стоп-кодона и представляет результат в виде логотипа (LOGO)[62]. Впрочем, несмотря на все перечисленные отличия, генетические коды, используемые всеми организмами, в общих чертах схожи[63].
В таблице ниже перечислены известные на данный момент нестандартные генетические коды[64][65]. Насчитывают 23 нестандартных генетических кода, причём наиболее частым отличием от стандартного генетического кода является превращение стоп-кодона UGA в смысловой, кодирующий триптофан[66].
Список нестандартных генетических кодов
| Биохимические свойства аминокислот | неполярная | полярная | основная | кислая | Терминация: стоп-кодон |
В геномах многих организмов наблюдается так называемое предпочтение кодонов, то есть частота встречаемости всех синонимичных кодонов, соответствующих определённой аминокислоте, неравна и для одних кодонов выше, чем для других[67][68]. Эволюционные основы возникновения предпочтения кодонов неясны. Согласно одной гипотезе, реже встречаются те кодоны, которые наиболее часто мутируют. Другая гипотеза утверждает, что предпочтение кодонов регулируется естественным отбором в пользу тех, которые обеспечивают наибольшую эффективность и точность экспрессии генов[69][70]. Предпочтение кодонов в значительной мере связано с GC-составом генома, и в некоторых случаях по GC-составу можно даже предсказать частоту использования кодонов[71]. С функциональной точки зрения предпочтение кодонов связано с эффективностью и точностью трансляции и, следовательно, уровнем экспрессии гена[72][73].
В настоящее время наиболее общепринятой гипотезой о происхождении жизни на Земле является гипотеза мира РНК. Любая модель возникновения генетического кода использует гипотезу о передаче основных функций от РНК-ферментов (рибозимов) к белковым ферментам. Как и предполагает гипотеза мира РНК, тРНК появились раньше аминоацил-тРНК-синтетаз, поэтому эти ферменты не могли оказать влияние на свойства тРНК[74].
Генетический код последнего универсального общего предка (LUCA) был основан, вероятнее всего, на ДНК, а не РНК[75]. Генетический код состоял из трёхнуклеотидных кодонов, и всего было возможно 64 различных кодона. Поскольку для построения белков использовалось только 20 аминокислот, некоторые аминокислоты кодировались несколькими кодонами[76][77][78][79].
Если бы соответствие между кодонами и аминокислотами было случайным, в природе существовало бы 1,5 × 1084 генетических кодов[80]. Это число получилось в результате расчёта количества способов, которыми можно 21 предмет (20 кодонов, кодирующих аминокислоты, и один стоп-кодон) разложить в 64 корзины, так, чтобы каждый предмет был использован по крайней мере единожды[81]. Однако соответствия кодонов и аминокислот неслучайны[82]. Аминокислоты, которые имеют общий путь биосинтеза, как правило, имеют общую первую позицию кодонов. Этот факт может быть пережитком раннего, более простого генетического кода, который содержал меньше аминокислот, чем современный, и постепенно включил в свой состав все 20 аминокислот[83]. Кодоны аминокислот со схожими физико-химическими свойствами также, как правило, похожи, что смягчает последствия точечных мутаций и нарушений трансляции[84][85].
Поскольку генетический код неслучаен, правдоподобная гипотеза о его возникновении должна объяснять такие свойства стандартного генетического кода, как отсутствие кодонов для D-аминокислот, включение всего лишь 20 аминокислот из возможных 64, ограничение синонимичных замен третьей позицией кодонов, функционирование в качестве стоп-кодонов именно кодонов UAG, UGA и UAA[86]. Существуют три основные гипотезы происхождения генетического кода. Каждая из них представлена множеством моделей, многие модели гибридны[87].
- Замороженная случайность: генетический код возник случайно и в таком виде закрепился. Возможно, древние рибозимы, подобные современным тРНК, имели разное сродство к аминокислотам, причём кодоны из разных частей одного и того же рибозима могли обладать наибольшим сродством к разным аминокислотам. После того, как появились первые функциональные пептиды, любое изменение генетического кода было бы летальным, поэтому он оказался «заморожен»[88].
- Стереохимическое сродство: генетический код определяется высоким сродством каждой аминокислоты с соответствующими ей кодонами и антикодонами. Сродство аминокислоты и антикодона означает, что предковым тРНК соответствовали те аминокислоты, с которыми они связывались с наибольшим сродством. В ходе эволюции соответствие антикодонов и аминокислот заменилось соответствием аминоацил-тРНК-синтетаз и аминокислот[89].
- Оптимальность: генетический код продолжал некоторое время изменяться после своего появления, поэтому современный код обеспечивает максимальную приспособленность и минимизирует последствия мутаций, то есть является лучшим из возможных генетических кодов[87].
Пептидо-нуклеиновые кислоты — Википедия
Материал из Википедии — свободной энциклопедии
Текущая версия страницы пока не проверялась опытными участниками и может значительно отличаться от версии, проверенной 3 ноября 2018; проверки требуют 2 правки. Текущая версия страницы пока не проверялась опытными участниками и может значительно отличаться от версии, проверенной 3 ноября 2018; проверки требуют 2 правки.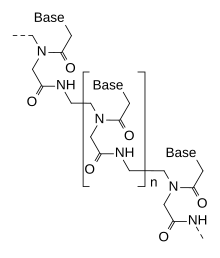
Пептидо-нуклеиновые кислоты (ПНК, англ. PNA, Peptide nucleic acid) — это химические вещества, похожие на РНК или ДНК. В настоящее время ПНК не обнаружены в составе живых организмов и получаются путём химического синтеза для использования в некоторых биологических экспериментах и медицине.
С химической точки зрения ПНК — это линейные полимеры N-(2-аминоэтил)глицина (или другие полиамиды-псевдопептиды), замещённые по азоту аминоэтильной группы производными азотистых оснований или других гетероциклических соединений, способных к нековалентному взаимодействию с азотистыми основаниями ДНК и РНК.
Благодаря тому, что размер звена такого псевдопептида близок к размеру рибозо- и дезоксирибозофосфатного звена нуклеиновых кислот, пептидонуклеиновые кислоты способны связываться с нуклеиновыми кислотами. Благодаря нейтральности (ПНК не являются кислотами и не несут отрицательного заряда при умеренных и высоких pH), константы связывания ПНК-лигандов с нуклеиновыми кислотами в некоторых случаях могут быть выше констант связывания комплементарных нуклеиновых кислот.
ПНК способна образовывать с РНК и одноцепочечными ДНК комплексы ПНК/РНК и ПНК/ДНК различной структуры; связывание ПНК с двухцепочечной ДНК может приводить к вытеснению одной из цепей ДНК и образованию комплексов, содержащих две молекулы ПНК и одну ДНК (триплексные комплексы (ПНК)2(ДНК)[1] и комплексы более сложной структуры[2].
Пептидная нуклеиновая кислота и пребиотические процессы[править | править код]
Эта кислота учитывается (вместе c пиранозильной РНК) как информационная молекула, функционирующая до периода мира-РНК или вместе с РНК. «Привлекательность» гипотезы о существовании таких систем обусловлена относительно несложным химическим составом (простые амиды и азотистые основания и более высокая стабильность по сравнению с РНК). Тем не менее, нет никаких доказательств (даже косвенных) о существовании самореплицирующихся систем на основе ПНК.
piРНК — Википедия
piРНК (англ. piwi-interacting RNA, piRNA, piwiRNA, в некоторых источниках встречается как пиРНК[1]) — наиболее крупный класс малых некодирующих РНК, экспрессируемых в клетках животных[2]; они обнаружены в комплексах с белками семейства Piwi, за что и получили своё название. piРНК обычно длиннее микроРНК и малых интерферирующих РНК и имеют длину 26—32 нуклеотида[3], кроме того, в отличие от микроРНК, они не так консервативны[2]. Белки Piwi относятся к большой группе белков Argonaute и экспрессируются почти исключительно в клетках зародышевой линии; они необходимы для поддержания стволовых клеток зародышевой линии, сперматогенеза и репрессии мобильных элементов. Комплексы Piwi с piРНК не только задействованы в сайленсинге ретротранспозонов и других генетических элементов на пост-трансляционном уровне, но имеют и некоторые другие, в значительной мере ещё неописанные эффекты, например, эпигенетические[4].
Остаётся не вполне ясным, как образуются piРНК, однако были предложены потенциальные методы исследования для этого вопроса, и установлено, что некоторые пути их образования отличаются от такового у микроРНК и малых интерферирующих РНК. В то же время некоторые малые некодирующие РНК другой группы, rasiRNA[en], считаются относящимися к piРНК[3][5].
Число обнаруженных piРНК составляет около 50 тысяч у млекопитающих и 13 тысяч у дрозофилы (Drosophila melanogaster)[6], что существенно больше числа известных малых РНК других классов. Поскольку значительная часть piРНК, особенно у млекопитающих, не связана с мобильными элементами, можно предполагать, что они выполняют и другие, ещё не описанные функции[3].
piРНК были открыты в 2006 году[3].
 Предполагаемая структура piРНК
Предполагаемая структура piРНКpiРНК были обнаружены как у позвоночных, так и беспозвоночных, и, хотя особенности биогенеза и типы взаимодействия с мишенями могут отличаться у разных видов, имеется ряд консервативных черт, присущих всем piРНК. У piРНК не обнаружено никаких выраженных мотивов[en] вторичной структуры[7], их длина составляет 26—32 н., и в 80—90 % случаев и у позвоночных, и у беспозвоночных первым нуклеотидом на 5′-конце является уридин (U). У нематоды Caenorhabditis elegans на 5′-конце имеется фосфатная группа, а на 3′-конце имеет место 2′-O-метилирование[8]. Такая модификация была также выявлена у дрозофилы[9], данио-рерио[10], мыши[11] и крысы[10]. Фосфатная группа на 5′-конце имеется и у piРНК млекопитающих[3]. Значение такой модификации пока точно не установлено, но предполагается, что она увеличивает стабильность piРНК[3][10].
У мыши в семейство Piwi входят три белка: Mili, Miwi и Miwi2[3], у человека — HIWI (или PIWIL1), HILI (или PIWIL2), HIWI2 (или PIWIL4) и HIWI3 (или PIWIL3)[12].
У млекопитающих около 17 % генов piРНК соответствует повторяющимся последовательностям, в том числе мобильным элементам. Стоит заметить, что количество piРНК, соответствующих повторам, меньше, чем доля повторов в геноме. Так, у грызунов эти соотношения равны 17 и ~42 % соответственно. Прочие piРНК кодируются уникальными генами, причём гены, кодирующие piРНК, располагаются в кластерах по всему геному. 90 % таких кластеров располагаются на участках, не содержащих аннотированных генов или повторов, но иногда могут находиться в интронах и экзонах[3]. Так, в то время как у D. melanogaster и позвоночных эти кластеры располагаются в участках, где отсутствуют белоккодирующие гены, у C. elegans гены piРНК располагаются среди белоккодирующих генов[5][8][13]. Каждый такой кластер может кодировать от 10 до многих тысяч piРНК, а его размер может варьировать от 1 до 100 килобаз[14]. Иногда кластеры piРНК располагаются рядом, но кодируются разными цепями; это может указывать на двунаправленную транскрипцию с общего промотора. Обнаружение и краткая аннотация кластеров piРНК в геномах осуществляется при помощи методов биоинформатики, которые всё более и более усложняются[15]. Хотя наличие кластеров генов piРНК высококонсервативно среди различных видов, этого нельзя сказать о последовательностях этих генов[16]. Например, хотя наиболее крупные кластеры piРНК грызунов имеют ортологов у человека, сходства последовательностей в этом случае не наблюдается[3].
Раньше считалось, что у млекопитающих piРНК и белки Piwi имеются только в семенниках[3]. Однако к настоящему времени установлено, что особая система piРНК имеется и в ооцитах млекопитающих[17]. Кроме того, показано, что при мейозе в ооцитах коровы экспрессируется дополнительный ген белков Piwi — PIWI-LIKE 3 (PIWIL3). Несмотря на это, piРНК у млекопитающих, по-видимому, функционируют только у самцов[18]. У беспозвоночных piРНК были выявлены как в клетках мужской, так и женской зародышевой линии[10].
На клеточном уровне piРНК были найдены и в ядре, и в цитоплазме, что свидетельствует о том, что piРНК могут функционировать и там, и там[5], и в связи с этим иметь множественные эффекты[19].
Уровень экспрессии piРНК меняется в ходе сперматогенеза. Они начинают детектироваться в пахитене (фаза профазы I деления мейоза) при делении диплоидных сперматоцитов мейозом (хотя образование piРНК начинается ещё в препахитенных клетках[20]), однако при образовании гаплоидных сперматид содержание piРНК в них резко падает, и в зрелой сперме они, судя по всему, отсутствуют[3].
Механизмы образования piРНК ещё не полностью установлены, хотя было предложено несколько возможных механизмов. В случаях, когда гены piРНК попадают в экзоны, piРНК соответствуют только смысловой (сенс-) цепи мРНК, поэтому они образуются только с одной цепи ДНК и, вероятно, являются производными длинных первичных транскриптов-предшественников. Это предположение согласуется с данными о наличии семенникоспецифичных EST[en] и мРНК, соответствующих локусам piРНК. Кроме того, в составе кластеров piРНК не выявлено развитых вторичных структур, характерных для pri-микроРНК. Поэтому процессинг piРНК, судя по всему, отличается от процессинга микроРНК и малых интерферирующих РНК. Об отсутствии двуцепочечных предшественников, характерных, в частности, для микроРНК, свидетельствует наличие только смысловых последовательностей у некоторых уникальных piРНК[3].
У дрозофилы и мыши в процессинге piРНК можно выделить два этапа: первичный процессинг и «пинг-понг»-цикл (амплификационная петля)[20].
Первичный процессинг[править | править код]
Как упоминалось выше, piРНК образуются из длинных транскриптов-предшественников. У дрозофил первичные транскрипты укорачиваются до piРНК-подобных малых РНК. Факторы, участвующие в этом процессе, ещё плохо изучены, однако недавние исследования показали, что, возможно, 5′-конец таких piРНК-подобных РНК образуется при помощи эндонуклеазы Zucchini. У мыши гомологом Zucchini является белок MitoPLD, также обладающий эндонуклеазными свойствами. После этого piРНК-подобные РНК связываются белками Piwi, вслед за этим их 3′-конец укорачивается ещё не описанной эндонуклеазой, и piРНК-подобные РНК приобретают размеры, соответствующие первичным piРНК. Возможно, важную роль в погрузке piРНК на белки Piwi играет белковый комплекс Hsp83/Shu. Далее piРНК 2′-O-метилируются комплексом HEN1/Pimet[20].
Цикл «пинг-понг»[править | править код]
PiРНК, подвергнувшаяся первичному процессингу, находится в состоянии, связанном с белками Piwi. Такие первичные piРНК являются антисмысловыми piРНК, комплементарными транскриптам мобильных элементов. У дрозофилы семейство белков Piwi представлено тремя белками: Piwi, Aubergine (Aub) и Ago3, но первичные piРНК связывают только белки Piwi и Aub. Комплексы Piwi с piРНК переносятся в ядро и в «пинг-понг»-цикле, происходящем в цитоплазме, не участвуют, однако участвуют в ядерном сайленсинге. Ассоциированные с Aub piРНК, комплементарно связывают транскрипты мобильных генетических элементов. Aub, как и другие белки группы Argonaute, способен разрезать фосфодиэфирную связь в РНК-мишени, расположенную напротив 10-го и 11-го нуклеотидов гидовой РНК (в данном случае — первичных piРНК). В результате разрыва образуется два фрагмента транскрипта мобильного элемента, у одного из которых 5′-конец отстоит на 10 нуклеотидов от 5′-конца первичной piРНК. Этот фрагмент — вторичная piРНК — в отличие от первичных piРНК некомплементарен транскрипту мобильного элемента и является смысловой piРНК. Поскольку чаще всего у первичных piРНК первый нуклеотид — уридин, то на 10-й позиции с 5′-конца у вторичных piРНК чаще всего располагается адениновый нуклеотид. Механизм процессинга 3′-конца вторичных piРНК пока неясен. Вторичная piРНК связывает белок Ago3 и направляется на разрезание первичного транскрипта-предшественника piРНК, из которого вырезается антисмысловая piРНК. Такие антисмысловые piРНК могут осуществлять сайленсинг мобильных элементов, а могут направлять образование новых смысловых piРНК. Таким образом, цикл «пинг-понг» совмещает процессинг piРНК и цитоплазматический сайленсинг мобильных элементов на уровне транскриптов. Он также даёт возможность усилить сайленсинг за счёт образования новых антисмысловых piРНК в ответ на усиление экспрессии мобильных элементов[3]. У дрозофилы в цикл «пинг-понг» могут вовлекаться не только первичные piРНК, но и piРНК, наследованные от матери. Цикл «пинг-понг» дрозофилы называют гетеротипным, так как в нём участвуют 2 различных белка Piwi — Aub и Ago3[20].
У мыши первичные piРНК связывают белки Mili и Miwi, а вторичные — белок Miwi2. Ассоциированные с Miwi piРНК, участвуют в цитоплазматическом сайленсинге, однако их мишени в большинстве своём неизвестны. Связанные с Mili первичные piРНК вовлекаются в цикл «пинг-понг». Образуемые в этом цикле вторичные piРНК, связываются с Miwi2, и комплекс piРНК с Miwi2 отправляется в ядро, где участвует в ядерном сайленсинге. Цикл «пинг-понг» мыши называют гомотипным, поскольку в нём участвует один белок Piwi — Mili. В образовании вторичных piРНК, связывающихся с Miwi2, определённую роль играет белковый комплекс HSP90/FKBP6. 2′-O-метилирование вторичных piРНК обеспечивает комплекс HEN1/Pimet[20].
У дрозофилы в соматических клетках половых желёз (например, в клетках фолликулов) белки Piwi также экспрессируются и связываются с первичными piРНК, однако белки Aub и Ago3 здесь экспрессируются на низком уровне, и для осуществления цикла «пинг-понг» их недостаточно[20].
По-видимому, схожий механизм репрессии мобильных элементов имеется и у данио-рерио[3]. Признаки наличия механизма «пинг-понг» обнаружены у самых примитивных животных — губок и стрекающих, что свидетельствует в пользу того, что механизм «пинг-понг» появился в самых ранних ветвях Metazoa и является консервативным механизмом репрессии мобильных элементов[3][21].
Другие белковые факторы[править | править код]
В биогенезе piРНК принимают участие и другие белки, не относящиеся к группе Piwi. В частности, таковы некоторые белки, относящиеся к суперсемейству Tudor (TDRD). Они содержат домен Tudor[en], который обеспечивает связывание TDRD-белка с другим белковым субстратом за счёт имеющихся у субстрата симметричных или асимметричных остатков диметиларгинина. У белков Piwi имеются симметричные остатки диметиларгинина рядом с N-концом, поэтому TDRD-белки способны связываться с ними и участвовать в РНК-сайленсинге[en]. По состоянию на 2011 год у дрозофилы было идентифицировано 11 TDRD-белков, участвующих в биогенезе piРНК, а у мыши было идентифицировано 7 таких TDRD-белков[20].
Например, было установлено, что мухи, мутантные по TDRD-белку Tud, фенотипически соответствуют мутантам по белку Aub. Белок Tud содержит 11 доменов Tudor и способен связываться как с Aub, так и с Ago3 за счёт симметричных остатков диметиларгинина, благодаря чему он служит «платформой» для «пинг-понг»-цикла. У мутантов по Tud белки Aub и Ago3 связывались с piРНК активнее, чем у мух дикого типа, что вызвало отклонения от нормального фенотипа[20].
Известно также несколько белков, участвующих в биогенезе piРНК и при этом не относящихся ни к Piwi, ни к TDRD-белкам. Так, у дрозофилы такой эффект был показан для следующих белков: Vasa (Vas), Maelstrom (Mael), Armi, Zuc, Squash (Squ) и Shu, причём все они, за исключением Squ, имеют гомологов у мыши. Большая часть этих факторов задействована в «пинг-понг»-механизме[20].
C. elegans[править | править код]
Установлено, у C. elegans имеются piРНК, но отсутствует механизм «пинг-понг»[22]. Однако недавние исследования биогенеза piРНК у C. elegans отчасти пролили свет на вопрос о том, как именно piРНК-опосредованная система защиты от паразитических мобильных элементов распознаёт «своё» и «чужое», подобно иммунной системе[20].
piРНК C. elegans имеют длину 21 нуклеотид и кодируются двумя кластерами на хромосоме IV, расположенными отдельно от белоккодирующих генов. На расстоянии ~42 нуклеотидов впереди от каждого кластера располагается последовательность CTGTTTCA, по-видимому, необходимая для осуществления транскрипции кластера РНК-полимеразой II. Синтезированные piРНК связываются с Piwi-белком PRG-1. Образовавшиеся комплексы piРНК с PRG-1 сканируют чужеродные транскрипты, причём для связывания с транскриптом достаточно неполной комплементарности (до 4 несоответствий). и запускают образование РНК-зависимых РНК-полимераз, которые обеспечивают образование и амплификацию особых малых интерферирующих РНК (22G-РНК). Последние связываются с белком WAGO — специфичным для C. elegans белком группы Argonaute. В цитоплазме эти комплексы обеспечивают сайленсинг генов на уровне мРНК, разрушая чужеродные транскрипты, а в ядре блокируют мобильные элементы на уровне транскрипции[20].
Распознавание «своего» и «чужого» и защита собственных транскриптов от разрушения, по-видимому, осуществляется на нескольких уровнях:
- piРНК, обусловливающие сайленсинг генов самого организма, подвергаются отрицательному отбору. Конкретнее, в клетках зародышевой линии практически нет piРНК, которые допускают большое число несоответствий оснований при комплементарном связывании с мишенью. Дело в том, что комплементарность первичных piРНК транскриптам транспозонов может быть обусловлена тем, что кластеры piРНК и транспозоны перекрываются, а именно, их последовательности ДНК могут находиться на антипараллельных цепях и частично перекрываться друг с другом. Поэтому piРНК комплементарны транскриптам транспозонов меньше, чем обыкновенным клеточным транскриптам. Можно сказать, что кластеры piРНК представляют собой «ловушку» для транспозонов.
- Дополнительную защиту «своей» мРНК обеспечивает связывания комплекса 22G-РНК с белком CSR-1, также относящимся к группе Argonaute. Возможно, он не вызывает разрушения связанной с ним мРНК, поскольку экспрессируется в меньшем количестве, чем WAGO, однако он предохраняет транскрипт от связывания с PRG-1. Более того, CSR-1 обеспечивает память о «своём» из прошедших процессов экспрессии генов в клетках зародышевой линии[20].
Функции, связанные с сайленсингом[править | править код]
За способность к сайленсингу мобильных элементов и обеспечению защиты генома от них piРНК получили название «стражей генома»[20]. По-видимому, у млекопитающих активность piРНК по сайленсингу транспозонов особенно важна в период развития зародыша, кроме того, и у человека, и у C. elegans необходимы для сперматогенеза[23][24]. Мутации, разрушающие систему сайленсинга мобильных элементов, опосредованного piРНК, у самцов мышей понижают фертильность или вовсе приводят к стерильности[3][25]. Возможно также, что некоторые заболевания репродуктивной системы человека, например, азооспермия, обусловлены дефектами в системе piРНК[20].
Отмечено некоторое действие piРНК на некоторые метилтрансферазы[en], осуществляющие метилирование, необходимое для распознавания и сайленсинга транспозонов, но эта связь ещё плохо понимаема[23].

piРНК могут передаваться по материнской линии, и для дрозофилы были показаны эпигенетические эффекты такого материнского наследования[13]. Активность специфических piРНК в эпигенетических процессах также требует взаимодействия piРНК с белками Piwi, HP1a и другими факторами[6]. Возможно, что piРНК участвуют в эпигенетической регуляции канцерогенеза[20].
У брюхоногого моллюска аплизии (Aplysia) (морского зайца) показано, что piРНК, содержащиеся в нейронах ЦНС, подавляют экспрессию гена CREB2 — репрессора памяти, индуцируя метилирование ДНК в его области и тем самым обеспечивая функционирование памяти. Кроме того, недавно piРНК были обнаружены в нейронах гиппокампа мыши. Вероятно, эти piРНК участвуют в формировании дендритных шипиков[20].
Наибольшие достижения в изучении piРНК были достигнуты при помощи особых техник секвенирования, например, Solexa и 454. С их помощью можно анализировать такие гетерогенные и сложные популяции РНК, как piРНК. Малый размер этих РНК создаёт определённые сложности при их искусственной экспрессии и амплификации, однако для их преодоления разработаны специальные техники на основе полимеразной цепной реакции[26][27].
- ↑ Марков А. В. Рождение сложности. Эволюционная биология сегодня. Неожиданные открытия и новые вопросы. — Москва: Астрель, Corpus, 2010. — С. 480—483. — 552 с. — ISBN 978-5-271-24663-0.
- ↑ 1 2 Seto A. G., Kingston R. E., Lau N. C. The coming of age for Piwi proteins. (англ.) // Molecular cell. — 2007. — Vol. 26, no. 5. — P. 603—609. — DOI:10.1016/j.molcel.2007.05.021. — PMID 17560367. [исправить]
- ↑ 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 Макарова Ю. А., Крамеров Д. А. Некодирующие РНК (рус.) // Биохимия. — 2007. — Т. 72, № 11. — С. 1427—1448.
- ↑ Siomi M. C., Sato K., Pezic D., Aravin A. A. PIWI-interacting small RNAs: the vanguard of genome defence. (англ.) // Nature reviews. Molecular cell biology. — 2011. — Vol. 12, no. 4. — P. 246—258. — DOI:10.1038/nrm3089. — PMID 21427766. [исправить]
- ↑ 1 2 3 Klattenhoff C., Theurkauf W. Biogenesis and germline functions of piRNAs. (англ.) // Development (Cambridge, England). — 2008. — Vol. 135, no. 1. — P. 3—9. — DOI:10.1242/dev.006486. — PMID 18032451. [исправить]
- ↑ 1 2 Haifan Lin, Hang Yin, Ergin Beyret, Seth Findley, Wei Deng. The role of the piRNA pathway in stem cell self-renewal. (англ.) // Developmental Biology. — 2008. — Vol. 319, № 2. — P. 479. — DOI:10.1016/j.ydbio.2008.05.048.
- ↑ Kandhavelu M,* Lammi C, Buccioni M, Dal Ben D, Volpini R, Marucci G. Existence of snoRNA, microRNA, piRNA characteristics in a novel non-coding RNA: x-ncRNA and its biological implication in Homo sapiens // Journal of Bioinformatics and Sequence Analysis. — 2009. — Vol. 1, № 2. — P. 031–040.
- ↑ 1 2 Ruby J. G., Jan C., Player C., Axtell M. J., Lee W., Nusbaum C., Ge H., Bartel D. P. Large-scale sequencing reveals 21U-RNAs and additional microRNAs and endogenous siRNAs in C. elegans. (англ.) // Cell. — 2006. — Vol. 127, no. 6. — P. 1193—1207. — DOI:10.1016/j.cell.2006.10.040. — PMID 17174894. [исправить]
- ↑ Vagin V. V., Sigova A., Li C., Seitz H., Gvozdev V., Zamore P. D. A distinct small RNA pathway silences selfish genetic elements in the germline. (англ.) // Science (New York, N.Y.). — 2006. — Vol. 313, no. 5785. — P. 320—324. — DOI:10.1126/science.1129333. — PMID 16809489. [исправить]
- ↑ 1 2 3 4 Houwing S., Kamminga L. M., Berezikov E., Cronembold D., Girard A., van den Elst H., Filippov D. V., Blaser H., Raz E., Moens C. B., Plasterk R. H., Hannon G. J., Draper B. W., Ketting R. F. A role for Piwi and piRNAs in germ cell maintenance and transposon silencing in Zebrafish. (англ.) // Cell. — 2007. — Vol. 129, no. 1. — P. 69—82. — DOI:10.1016/j.cell.2007.03.026. — PMID 17418787. [исправить]
- ↑ Kirino Y., Mourelatos Z. Mouse Piwi-interacting RNAs are 2′-O-methylated at their 3′ termini. (англ.) // Nature structural & molecular biology. — 2007. — Vol. 14, no. 4. — P. 347—348. — DOI:10.1038/nsmb1218. — PMID 17384647. [исправить]
- ↑ Ross R. J., Weiner M. M., Lin H. PIWI proteins and PIWI-interacting RNAs in the soma. (англ.) // Nature. — 2014. — Vol. 505, no. 7483. — P. 353—359. — DOI:10.1038/nature12987. — PMID 24429634. [исправить]
- ↑ 1 2 Brennecke J., Malone C. D., Aravin A. A., Sachidanandam R., Stark A., Hannon G. J. An epigenetic role for maternally inherited piRNAs in transposon silencing. (англ.) // Science (New York, N.Y.). — 2008. — Vol. 322, no. 5906. — P. 1387—1392. — DOI:10.1126/science.1165171. — PMID 19039138. [исправить]
- ↑ O’Donnell K. A., Boeke J. D. Mighty Piwis defend the germline against genome intruders. (англ.) // Cell. — 2007. — Vol. 129, no. 1. — P. 37—44. — DOI:10.1016/j.cell.2007.03.028. — PMID 17418784. [исправить]
- ↑ Rosenkranz D., Zischler H. proTRAC—a software for probabilistic piRNA cluster detection, visualization and analysis. (англ.) // BMC bioinformatics. — 2012. — Vol. 13. — P. 5. — DOI:10.1186/1471-2105-13-5. — PMID 22233380. [исправить]
- ↑ Malone C. D., Hannon G. J. Small RNAs as guardians of the genome. (англ.) // Cell. — 2009. — Vol. 136, no. 4. — P. 656—668. — DOI:10.1016/j.cell.2009.01.045. — PMID 19239887. [исправить]
- ↑ Tam O. H., Aravin A. A., Stein P., Girard A., Murchison E. P., Cheloufi S., Hodges E., Anger M., Sachidanandam R., Schultz R. M., Hannon G. J. Pseudogene-derived small interfering RNAs regulate gene expression in mouse oocytes. (англ.) // Nature. — 2008. — Vol. 453, no. 7194. — P. 534—538. — DOI:10.1038/nature06904. — PMID 18404147. [исправить]
- ↑ Silva, Ricardo Andrés Zacarias. Piwi proteins in mammals: a cow’s perspective. (неопр.). — 2011. — P. 4. — 50 p.
- ↑ Ruvkun G. Tiny RNA: Where do we come from? What are we? Where are we going? (англ.) // Trends in plant science. — 2008. — Vol. 13, no. 7. — P. 313—316. — DOI:10.1016/j.tplants.2008.05.005. — PMID 18562240. [исправить]
- ↑ 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 Ishizu H., Siomi H., Siomi M. C. Biology of PIWI-interacting RNAs: new insights into biogenesis and function inside and outside of germlines. (англ.) // Genes & development. — 2012. — Vol. 26, no. 21. — P. 2361—2373. — DOI:10.1101/gad.203786.112. — PMID 23124062. [исправить]
- ↑ Grimson A., Srivastava M., Fahey B., Woodcroft B. J., Chiang H. R., King N., Degnan B. M., Rokhsar D. S., Bartel D. P. Early origins and evolution of microRNAs and Piwi-interacting RNAs in animals. (англ.) // Nature. — 2008. — Vol. 455, no. 7217. — P. 1193—1197. — DOI:10.1038/nature07415. — PMID 18830242. [исправить]
- ↑ Das P. P., Bagijn M. P., Goldstein L. D., Woolford J. R., Lehrbach N. J., Sapetschnig A., Buhecha H. R., Gilchrist M. J., Howe K. L., Stark R., Matthews N., Berezikov E., Ketting R. F., Tavaré S., Miska E. A. Piwi and piRNAs act upstream of an endogenous siRNA pathway to suppress Tc3 transposon mobility in the Caenorhabditis elegans germline. (англ.) // Molecular cell. — 2008. — Vol. 31, no. 1. — P. 79—90. — DOI:10.1016/j.molcel.2008.06.003. — PMID 18571451. [исправить]
- ↑ 1 2 Aravin A. A., Sachidanandam R., Bourc’his D., Schaefer C., Pezic D., Toth K. F., Bestor T., Hannon G. J. A piRNA pathway primed by individual transposons is linked to de novo DNA methylation in mice. (англ.) // Molecular cell. — 2008. — Vol. 31, no. 6. — P. 785—799. — DOI:10.1016/j.molcel.2008.09.003. — PMID 18922463. [исправить]
- ↑ Wang G., Reinke V. A C. elegans Piwi, PRG-1, regulates 21U-RNAs during spermatogenesis. (англ.) // Current biology : CB. — 2008. — Vol. 18, no. 12. — P. 861—867. — DOI:10.1016/j.cub.2008.05.009. — PMID 18501605. [исправить]
- ↑ Barrett et. al., 2013, p. 37.
- ↑ Ro S., Park C., Jin J., Sanders K. M., Yan W. A PCR-based method for detection and quantification of small RNAs. (англ.) // Biochemical and biophysical research communications. — 2006. — Vol. 351, no. 3. — P. 756—763. — DOI:10.1016/j.bbrc.2006.10.105. — PMID 17084816. [исправить]
- ↑ Tang F., Hayashi K., Kaneda M., Lao K., Surani M. A. A sensitive multiplex assay for piRNA expression. (англ.) // Biochemical and biophysical research communications. — 2008. — Vol. 369, no. 4. — P. 1190—1194. — DOI:10.1016/j.bbrc.2008.03.035. — PMID 18348866. [исправить]
- Siomi M. C., Sato K., Pezic D., Aravin A. A. PIWI-interacting small RNAs: the vanguard of genome defence. (англ.) // Nature reviews. Molecular cell biology. — 2011. — Vol. 12, no. 4. — P. 246—258. — DOI:10.1038/nrm3089. — PMID 21427766. [исправить]
- Ishizu H., Siomi H., Siomi M. C. Biology of PIWI-interacting RNAs: new insights into biogenesis and function inside and outside of germlines. (англ.) // Genes & development. — 2012. — Vol. 26, no. 21. — P. 2361—2373. — DOI:10.1101/gad.203786.112. — PMID 23124062. [исправить]
- Lucy W. Barrett, Sue Fletcher, Steve D. Wilton. Untranslated Gene Regions and Other Non-coding Elements. — SpringerBriefs in Biochemistry and Molecular Biology, 2013. — 57 p. — ISBN 978-3-0348-0679-4.
- Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, Peter Walter. Molecular Biology of the Cell. — 5. — Garland Science, 2008. — 1392 с. — ISBN 0815341059.
Малые интерферирующие РНК — Википедия
Ма́лые интерфери́рующие РНК или короткие интерферирующие РНК (англ. siRNA, small interfering RNA) — это класс двухцепочечных РНК, длиной 20-25 нуклеотидов. Взаимодействие малых интерферирующих РНК с матричной РНК (мРНК) целевого гена приводит к деградации последней (в процессе РНК-интерференции), предотвращая трансляцию мРНК на рибосомах в кодируемый ею белок. В конечном итоге результат действия малых интерферирующих РНК идентичен тому, как если бы просто снижалась экспрессия гена.
В клетке РНК-интерференция является важной частью механизмов противовирусной защиты и поддержания структуры хроматина. Молекулярные механизмы данных взаимодействий в настоящее время исследуются, в частности, была предложена гипотеза участия малых РНК в РНК-зависимом метилировании ДНК[1].
Малые интерферирующие РНК были открыты в 1999 году группой Дэвида Болкомба (англ. David Baulcombe) в Великобритании как компонент системы пост-транскрипционного сайленсинга генов у растений (англ. PTGS, en:post-transcriptional gene silencing). Группа опубликовала полученные данные в журнале Science[2].
В 2001 году группой Томаса Тущла (англ. Thomas Tuschl) было показано, что синтетические малые интерферирующие РНК могут индуцировать РНК-интерференцию в клетках млекопитающих. Соответствующие результаты были опубликованы в журнале Nature[3]. Это открытие привело к росту интереса к использованию РНК-интерференции для биомедицинских исследований и разработки лекарственных препаратов.
Малые интерферирующие РНК представляют собой короткие (как правило, длиной 21 нуклеотид) двухцепочечные РНК с двумя неспаренными выступающими нуклеотидами на 3′-концах.
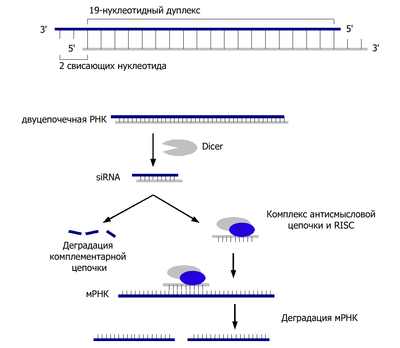 Механизм синтеза малых интерферирующих РНК
Механизм синтеза малых интерферирующих РНККаждая из двух цепей РНК имеет фосфатную группу на 5′-конце и гидроксильную группу на 3′-конце. Короткие интерферирующие РНК с такой структурой образуются в результате активности фермента Dicer, субстратами которого являются длинные двухцепочечные РНК или короткие РНК, содержащие шпильки[4]. Малые интерферирующие РНК могут быть искусственно введены в клетки для нокдауна определённого гена. При этом экспрессия практически любого гена с известной последовательностью нуклеотидов может быть целенаправленно изменена. Данное свойство делает короткие интерферирующие РНК удобным инструментом для исследования функций генов и изучения мишеней лекарственных средств.
Целенаправленное подавление экспрессии генов с помощью трансфекции экзогенной интерферирующей РНК в клетки связано с определёнными трудностями, поскольку нокдаун гена в этом случае имеет временный характер, особенно в быстро делящихся клетках. Один из способов преодоления этих трудностей состоит в том, чтобы ввести в клетку вектор, обеспечивающий экспрессию соответствующей малой интерферирующей РНК в течение более длительного периода времени[5]. Такой вектор, как правило, содержит промотор U6 или h2, обеспечивающий транскрипцию РНК полимеразой III, которая транскрибирует малые ядерные РНК. За промотором следуют короткая последовательность нуклеотидов, кодирующая малую интерферирующую РНК (19—29 нуклеотидов) и последовательность комплементарная ей, которые разделённы между собой 4—11 нуклеотидами, которые во вторичной структуре малой интерферирующей РНК образуют петлю. В целом, соответствующий транскрипт напоминает по форме шпильку в результате комплементарного спаривания последовательностей в его начале и конце. Предполагается (хотя это не установлено достоверно), что такие шпильки затем превращаются в короткие интерферирующие РНК под действием фермента Dicer.
Двухцепочечные РНК могут усиливать экспрессию генов по механизму, называемому РНК-зависимой активацией генов (англ. RNAa, small RNA-induced gene activation). Показано, что двухцепочечные РНК, комплементарные промоторам генов-мишеней, вызывают активацию соответствующих генов. РНК-зависимая активация при введении синтетических двухцепочечных РНК была показана для клеток человека. Не известно, имеется ли подобная система в клетках других организмов.[6]
Исключение неспецифических эффектов[править | править код]
Поскольку РНК-интерференция пересекается со множеством других цепочек реакций, при экспериментальном введении малых интерферирующх РНК могут включаться неспецифические эффекты. Появление двухцепочечных РНК в клетках млекопитающих может быть следствием заражения вирусом и поэтому приводит к запуску иммунного ответа. Более того, так как структурно похожие микроРНК изменяют экспрессию генов путём неполного спаривания с мишенью мРНК, введение малых интерферирующих РНК может вызвать нежелательный побочный эффект.
Врождённый иммунитет[править | править код]
Введение значительного количества малых интерферирующих РНК может вызвать побочные эффекты из-за того, что включается врождённый иммунный ответ. Вероятно это происходит из-за активации протеинкиназы R, которая чувствительна к малым интерферирующим РНК, возможно также участие гена RIG I (англ. retinoic acid inducible gene I). Также описана индукции цитокинов через рецептор TLR 7 (англ. toll-like receptor 7). Один из перспективных методов снижения побочных эффектов состоит в преобразовании малых интерферирующих РНК в микроРНК. МикроРНК синтезируются в норме, поэтому сравнительно небольшая концентрация образующихся малых интерферирующих РНК можно приводить к сравнимому по силе эффекту нокдауна генов. Это должно свести к минимуму побочные эффекты.
Побочные эффекты[править | править код]
Сбой мишени — это ещё одна трудность при использовании малых интерферирующих РНК как инструмента для достижения нокдауна генов. Гены с неполной комплементарностью блокируются малыми интерферирующими РНК (т. е. фактически малые интерферирующие РНК действуют как микроРНК), что приводит к трудностям в интерпретации результатов опытов и содержит риск токсичности. Однако, этого можно избежать, организуя соответствующие контрольные опыты, и создавая алгоритмы конструирования малых интерферирующих РНК, которые приводят к таким РНК, не дающим сбоев мишени. Затем можно проанализировать экспрессию генов по всему геному, например, при помощи метода микромассивов (англ. microarray technology), чтобы проверить отсутствие сбоев мишени и произвести дальнейшую настройку алгоритмов. В статье сотрудников лаборатории доктора Хворовой за 2006 год рассматриваются фрагменты длиной 6 или 7 пар оснований, начинающиеся с позиции 2, в малой интерферирующей РНК, соответствующей участку 3’UTR в генах, где происходит сбой мишени[7].
Возможные применения в терапии и препятствия к этому[править | править код]
Давая возможность выключить по существу любой ген по желанию, РНК-интерференция на основе малых интерферирующих РНК вызвала огромный интерес в фундаментальной [8] и прикладной биологии. Число широкоохватных тестов на базе РНК-интерференции для выявления важных генов в биохимических путях постоянно растет. Поскольку развитие болезней также обусловлено активностью генов, ожидается, что в некоторых случаях выключение гена при помощи малой интерферирующей РНК может давать терапевтический эффект.
Однако применение РНК-интерференции на основе малых интерферирующих РНК к животным, и в особенности к людям, сталкивается со множеством трудностей. В экспериментах было показано, что эффективность малых интерферирующих РНК оказывается различной для разных типов клеток: одни клетки легко откликаются на воздействие малых интерферирующих РНК и демонстрируют снижение экспрессии генов, в других же подобного не наблюдается, несмотря на эффективную трансфекцию. Причины этого явления пока что плохо изучены.
Результаты первой фазы испытаний двух первых терапевтических препаратов, действующих по механизму РНК-интерференции (предназначены для лечения макулодистрофии), опубликованные в конце 2005 года, показывают, что препараты на основе малых интерферирующих РНК легко переносятся пациентами и имеют приемлемые фармакокинетические свойства[9].
Предварительные клинические испытания малых интерферирующих РНК, нацеленных на вирус Эбола, указывают на то, что они могут быть эффективны для постконтактной профилактики заболевания. Данный препарат позволил выжить всей группе подопытных приматов, получивших летальную дозу Заирского Эболавируса[10].
- ↑ Галицкий В.А. Гипотеза о механизме инициации малыми РНК метилирования ДНК de novo и аллельного исключения (рус.) // Цитология. — 2008. — Т. 50(4). — С. 277—286.
- ↑ Hamilton A., Baulcombe D. A species of small antisense RNA in posttranscriptional gene silencing in plants (англ.) // Science : journal. — 1999. — Vol. 286, no. 5441. — P. 950—952. — DOI:10.1126/science.286.5441.950. — PMID 10542148.
- ↑ Elbashir S., Harborth J., Lendeckel W., Yalcin A., Weber K., Tuschl T. Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells (англ.) // Nature : journal. — 2001. — Vol. 411, no. 6836. — P. 494—498. — DOI:10.1038/35078107. — PMID 11373684.
- ↑ Bernstein E., Caudy A., Hammond S., Hannon G. Role for a bidentate ribonuclease in the initiation step of RNA interference (англ.) // Nature : journal. — 2001. — Vol. 409, no. 6818. — P. 363—366. — DOI:10.1038/35053110. — PMID 11201747.
- ↑ Miyagishi M., Taira K. Development and application of siRNA expression vector (англ.) // Nucleic Acids Research Supplement (англ.)русск. : journal. — 2002. — Vol. 2. — P. 113—114. — PMID 12903131.
- ↑ Li L. C. Small RNA-Mediated Gene Activation // RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity (англ.). — Caister Academic Press (англ.)русск., 2008.
- ↑ Birmingham A., Anderson E., Reynolds A., Ilsley-Tyree D., Leake D., Fedorov Y., Baskerville S., Maksimova E., Robinson K., Karpilow J., Marshall W., Khvorova A. 3′ UTR seed matches, but not overall identity, are associated with RNAi off-targets (англ.) // Nat Methods : journal. — 2006. — Vol. 3, no. 3. — P. 199—204. — DOI:10.1038/nmeth854. — PMID 16489337.
- ↑ Alekseev O. M., Richardson R. T., Alekseev O., O’Rand M. G. Analysis of gene expression profiles in HeLa cells in response to overexpression or siRNA-mediated depletion of NASP (англ.) // Reproductive Biology and Endocrinology : journal. — 2009. — Vol. 7. — P. 45. — DOI:10.1186/1477-7827-7-45. — PMID 19439102.
- ↑ Tansey B. Macular degeneration treatment interferes with RNA messages, San Francisco Chronicle (11 августа 2006).
- ↑ Postexposure protection of non-human primates against a lethal Ebola virus challenge with RNA interference: a proof-of-concept study Prof Thomas W Geisbert PhD,Amy CH Lee MSc,Marjorie Robbins PhD,Joan B Geisbert,Anna N Honko PhD,Vandana Sood MSc,Joshua C Johnson BSc,Susan de Jong PhD,Iran Tavakoli BSc,Adam Judge PhD,Lisa E Hensley PhD,Ian MacLachlan PhD The Lancet — 29 May 2010 ( Vol. 375, Issue 9729, Pages 1896-1905 ) DOI: 10.1016/S0140-6736(10)60357-1
